小果手把手教你用miniconda安装EDTA(下)
书接上回,我们已经安装好miniconda,现在该安装EDTA了。 小果安装的时候一开始走了不少弯路,比如说试图通过以下代码来安装EDTA
#创建一个新的conda环境,并指定python版本
conda create -n bioinfo python=3.9
# 激活这个环境
conda activate bioinfo
#在bioconda channel下载edta软件包
conda install -c bioconda edta
结果总是报错!每次安装新软件都觉得自己是呆瓜的小果研究了半天,认为应该是环境依赖出了问题,但不会解决,搜寻网络各种教程后果断删掉了刚刚创建的环境,使用了另一种安装方式((11条消息) EDTA 最简易安装方法_tesorter_Plaichat-123的博客-CSDN博客),很快成功了,感谢来自CSDN的馈赠~
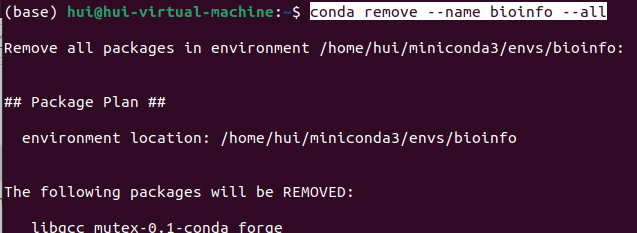
conda remove –name bioinfo –all#删除名为bioinfo的环境
git clone https://github.com/oushujun/EDTA#在github上克隆(下载)EDTA项目
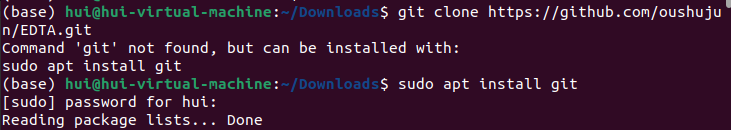
没有git?装!
sudo apt install git
装完git以后再次执行
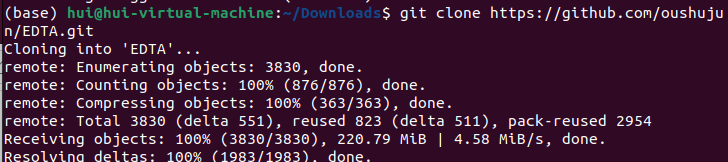
git clone https://github.com/oushujun/EDTA
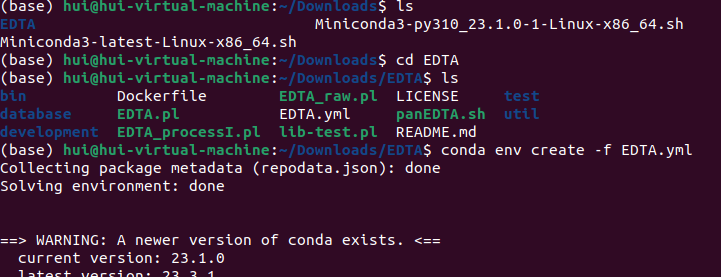
装好了!使用EDTA.yml继续创建命名为EDTA的conda环境。
conda env create -f EDTA.yml
conda info –envs#查看已安装的环境
conda activate EDTA#激活EDTA
conda create –name repeatmasker repeatmasker
#创建新的环境并安装RepeatMasker
conda install -c bioconda samtools#通过bioconda安装samtools
然后我们就可以注释基因组中的重复序列了,大概流程如下所示:
- 下载了小鼠的全基因组序列文件(.fasta)
- 下载小鼠的CDS序列文件(可以在gencode下载)
- 构建小鼠的repeat library
- 对小鼠全基因组序列进行重复序列注释(用到了RepeatMasker)
- 统计注释结果
小果下期分析详细的注释教程哦!不要错过~
欢迎使用:云生信 – 学生物信息学 (biocloudservice.com)
如果想用服务器可以联系微信:18502195490