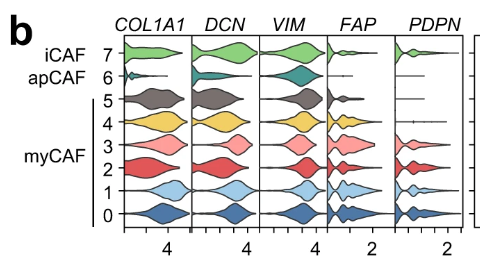
小图最近阅读文献时候发现有关单细胞分析是越来越多,并且许多高分文章中的单细胞图都非常的好看。小图就突发奇想想去复现这些高分文章中的图片,想到他们能发这么高分的文章,这里面图想必加了不少分,毕竟数据可视化才最能体现数据的美观性,才能够吸引读者。所以,我们有时候想不知道如何可视化数据时候,可以借鉴高分文章中的图片,用在自己的文章中。因此呢,小图就找了一篇文章,《Single cell transcriptomic analyses implicate an immunosuppressive tumor microenvironment in pancreatic cancer liver metastasis》IF16.6,这么高分的文章,小图看了看这篇文章的图,发现许多图都是我们见过的,还很好复现,这些图都是拼接而成的,组成一个非常好看的图。就比如下面这个图b
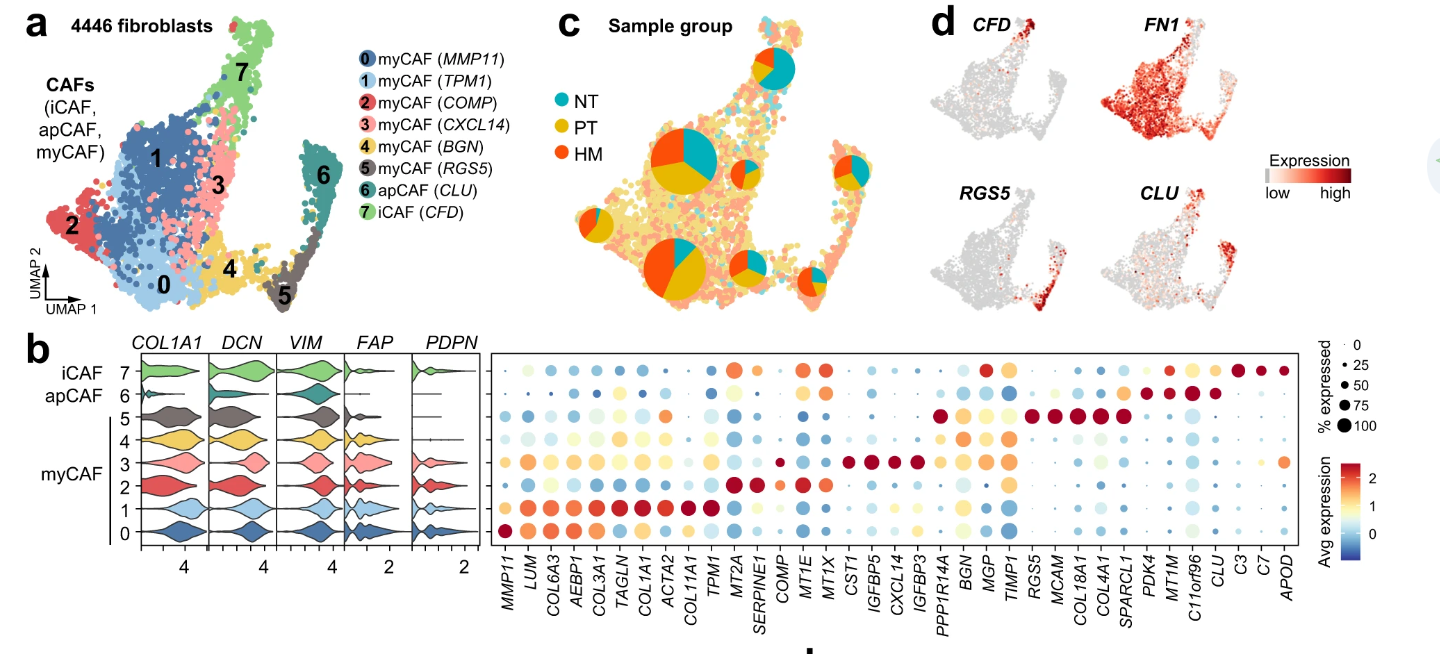
上图中的图b是不是挺好看的,但是小伙伴发现没,这个图b是我们非常熟悉的图,可能我们平时都绘制过,但是又不一样,小图这里就带小伙伴去复现这个图b,
我们想要复现这个图,肯定需要从哪里下手,首先就是了解这个图的组成,仔细观察,小图发现这是由两种图组成,一个是堆叠的小提琴图,右边是气泡热图,好了我们知道这两个组图的名字之后就是去实现他们。
我们从右边的图下手,因为这个文章是单细胞的文章,小图自己也用了自己单细胞数据,小伙伴可以套用数据格式去复现哦,
我们开始载入几个包,
library(scRNAtoolVis) # 小图加载这个包,为了使用这个包的数据,
library(Seurat) # dotplot,单细胞绘图使用
library(cowplot) # plot_grid
httest <- system.file(“extdata”, “htdata.RDS”, package = “scRNAtoolVis”)
pbmc <- readRDS(httest)
然后我们载入数据
# 添加分组
pbmc$groups <- rep(c(‘stim’,’control’),each = 1319)
# 添加细胞类型
pbmc$celltype <- Seurat::Idents(pbmc)
# 载入 markergene
data(“top3pbmc.markers”)
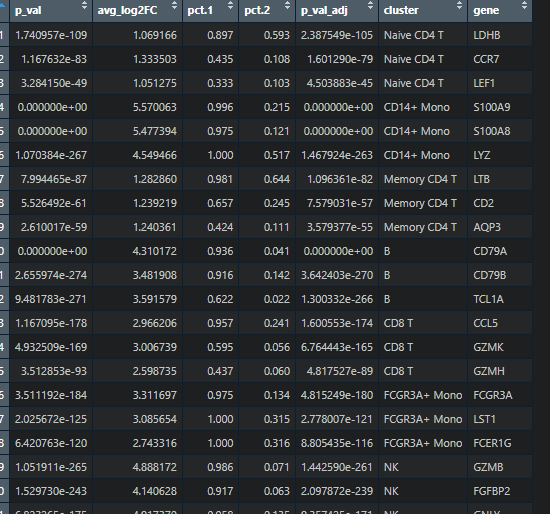
这个其实就是我们一般单细胞操作官方流程后,跑完第一个数据结果,小伙伴可以直接使用
后面我们可以使用这个数据去绘制图片
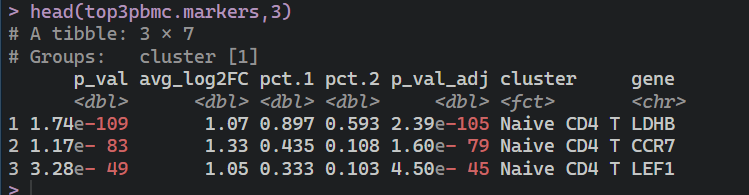
# check
head(top3pbmc.markers,3)
p <- DotPlot(pbmc,
features = split(top3pbmc.markers$gene, top3pbmc.markers$cluster),
cols = c(“#dddddd”, “#448444”)
) +
RotatedAxis() + # 来自Seurat
theme(
panel.border = element_rect(color = “black”),
panel.spacing = unit(1, “mm”),
axis.title = element_blank(),
axis.text.y = element_blank(),
)#设置主题,
p$data$feature.groups2 <- factor(p$data$feature.groups,
levels = c(“Platelet”,”DC”,”NK”,
“FCGR3A+ Mono”,”CD8 T”,”B”,
“Memory CD4 T”,”CD14+ Mono”,”Naive CD4 T”))#注释单细胞数据
P
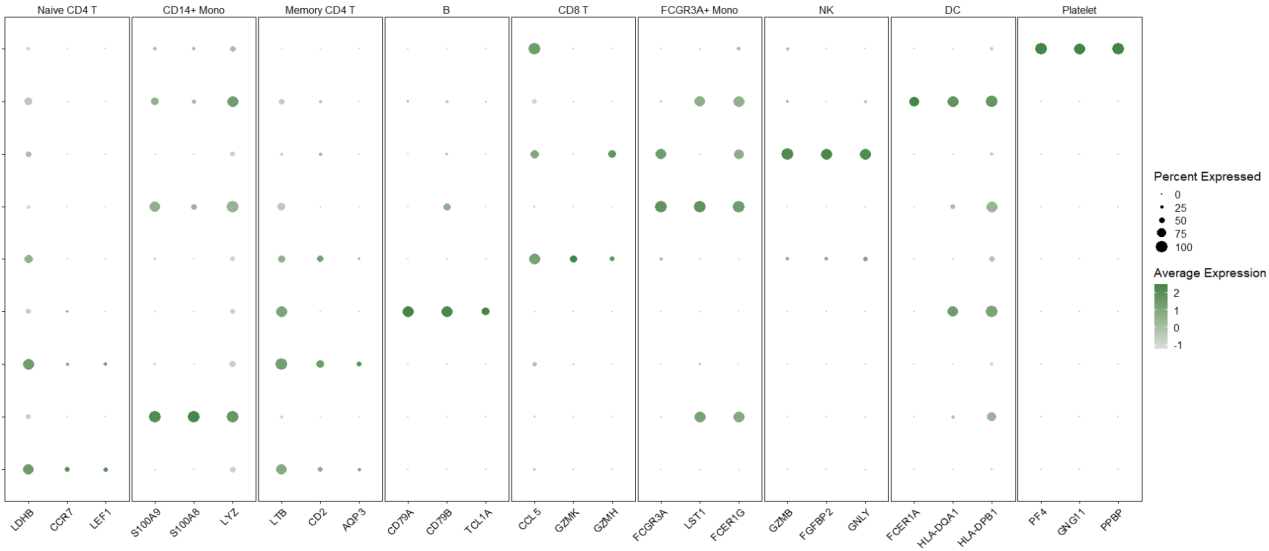
这样一来文章中右边的图就大致的绘制出来了,上面小图还加了单细胞的注释结果,更加丰富,但是发现有一些小细节没有处理到位,中间还有分割的线,下面我们在继续绘制,我们对细节的修改需要用到ggplot2中的参数处理来看看如何处理
我们使用ggplot2从头绘制
library(ggh4x)#需要用到里面的函数处理数据
library(RColorBrewer)#加载这个包只是为了填充颜色
#下面把数据填充一下,
strip <- strip_themed(
background_x = elem_list_rect(fill = brewer.pal(9, “Paired”)))
#下面绘制图片,从主题颜色等,
p$data %>%
ggplot(aes(x = features.plot,
y = id)) +
geom_point(aes(size = pct.exp,
color = avg.exp.scaled)) +
facet_wrap2(~feature.groups2,
scales = “free_x”,
strip = strip,
nrow = 1) +
#主题的修正
theme_classic() +
theme(axis.text.x = element_text(angle = 90,
hjust = 0.5, #气泡间距
vjust = 0.3,
color = “black”),
axis.title = element_blank(),
strip.background = element_rect(color = “white”),
axis.text.y = element_blank()) +
scale_color_gradient(low = “#ffffff”,#底数据颜色设置
high = “#448444”,
name = “avg.exp”) -> p
#上述的绘图,许多都是默认的参数,小伙伴可以方向去使用
P#查看一下图
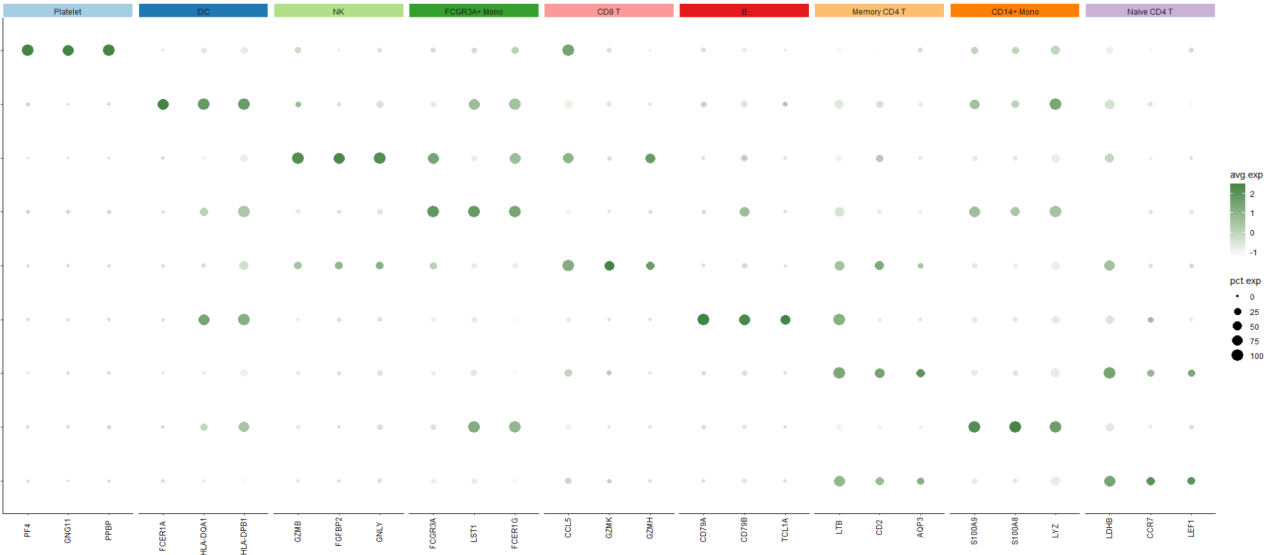
这样一来我们右边的图就复现的差不多,对比一下
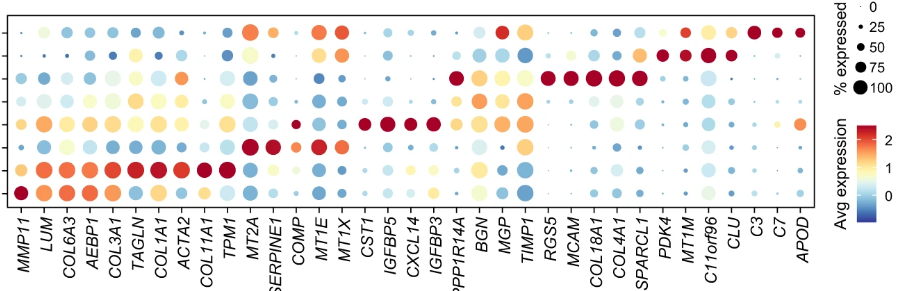
发现都已经差不多了,只是颜色的设置,小伙伴对此可以自行去设置,还有小图自己加的单细胞分组信息在上方,小伙伴不喜欢这个单细胞分组可以删除,只需要把strip这个信息注释掉就可以,
好了我们实现了右边的图片,去复现一下左边的堆叠小提琴图吧
我们先载入使用的包
library(Seurat)
library(ggpubr)
library(ggsignif)
载入我们实例的数据
其实堆叠小提琴图,小伙伴不少见,做单细胞数据都会用得到,来看看如何绘制的
# 加载数据对象
#
pbmc <- readRDS(“pbmc_2k_1.rds”)
identity <- readRDS(“pbmc_2k_2.rds”)
features <- c(“CD79A”, “MS4A1”, “CD8A”, “CD8B”, “LYZ”,
“LGALS3”)
我们提取几个基因绘制
# 提取数据框子集
pbmc <- pbmc[,features]
# 添加细胞name和细胞群身份
pbmc$Cell <- rownames(pbmc)
pbmc$Idents <- identity
对上述数据处理一下
# melt数据变长
pbmc <- reshape2::melt(pbmc,
id.vars = c(“Cell”,”Idents”),
measure.vars = features,
variable.name = “Feat”,
value.name = “Expr”)
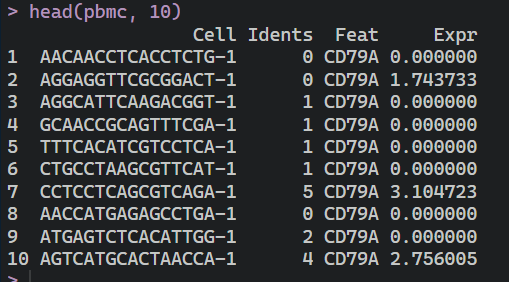
# 查看数据前10行
head(pbmc, 10)
下面我们就去绘制图片
#主题theme(大小,颜色),legend
p2 <- ggplot(pbmc, aes(Feat, Expr, fill = Feat)) +
geom_violin(scale = “width”, adjust = 1, trim = TRUE) +
scale_y_continuous(expand = c(0, 0),
position=”right”,
labels = function(x)#对主题水平的设置
c(rep(x = “”, times = length(x)-2), x[length(x) – 1], “”)) +
facet_grid(rows = vars(Idents), scales = “free”, switch = “y”) +
#下面是对主题的设置
theme_cowplot(font_size = 12) +
theme(legend.position = “none”,
panel.spacing = unit(0, “lines”),#图中线的设置
plot.title = element_text(hjust = 0.5),
panel.background = element_rect(fill = NA, color = “black”),#颜色填充
plot.margin = margin(6, 6, 0, 6, “pt”),#图中小提琴图的间距
strip.background = element_blank(),
strip.text = element_text(face = “bold”),
strip.text.y.left = element_text(angle = 0),
axis.title.x = element_blank(),
axis.ticks.x = element_blank(),
axis.text.x = element_blank()) +
ggtitle(“”) +#添加主题名字,可以不写
ylab(“Expression Level”)#Y轴的名称
#coord_flip()#可以修改横纵坐标
p2
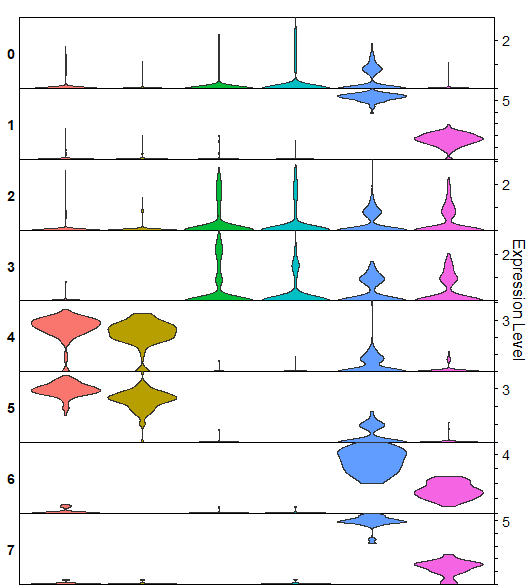
这个图是不是和文章中差不多了
一些细节还没处理到位,小伙伴要是需要将小提琴反转可以使用coord_flip()
我们下面对数据颜色的设置一下
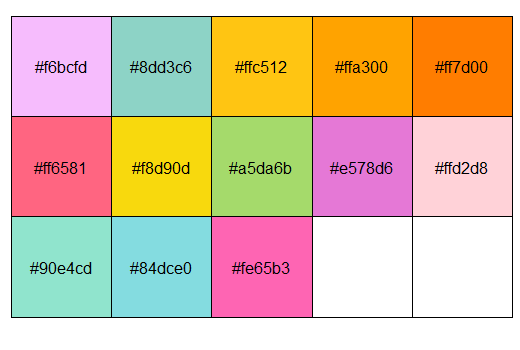
spring <- c(“#f6bcfd”,”#8dd3c6″,”#ffc512″,”#ffa300″,”#ff7d00″,”#ff6581″,”#f8d90d”,”#a5da6b”,”#e578d6″,”#ffd2d8″,”#90e4cd”,”#84dce0″,”#fe65b3″)
对颜色赋值
library(scales)
show_col(spring,ncol=5)#查看一下有那些颜色
p1 <- ggplot(pbmc, aes(Feat, Expr, color = Feat, fill = Feat)) +
geom_violin(scale = “width”, adjust = 1, trim = TRUE) +
scale_y_continuous(expand = c(0, 0),
position=”right”,
labels = function(x)
c(rep(x = “”, times = length(x)-2), x[length(x) – 1], “”)) +
facet_grid(rows = vars(Idents), scales = “free”, switch = “y”) +
theme_cowplot(font_size = 12) +
theme(legend.position = “none”,
panel.spacing = unit(0, “lines”),
plot.title = element_text(hjust = 0.5),
panel.background = element_rect(fill = NA, color = “black”),
plot.margin = margin(6, 6, 0, 6, “pt”),
strip.background = element_blank(),
strip.text = element_text(face = “bold”),
strip.text.y.left = element_text(angle = 0),
axis.title.x = element_blank(),
axis.ticks.x = element_blank(),
axis.text.x = element_blank()) +
ggtitle(“”) +
ylab(“Expression Level”) +
scale_fill_manual(values = spring) +
scale_color_manual(values = spring)
和上述代码一样,小伙伴多多理解一下
p1
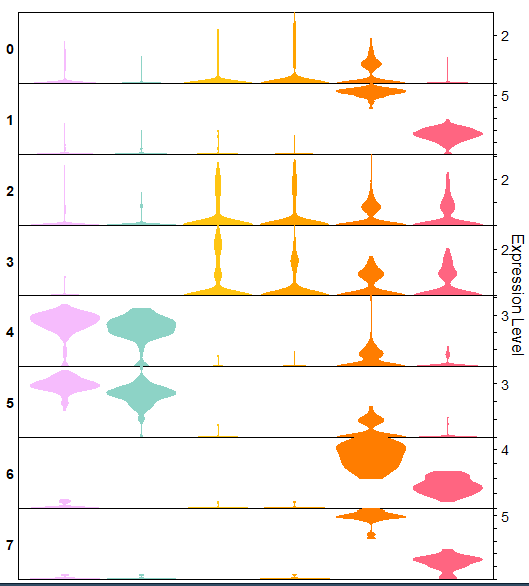
颜色可能没做到一样,但是也挺好看,那么这两个图我们都绘制完成了,我们就去组合一下这两个图,来看看最终的步骤
plot_grid(p1,p, align =”h”, axis=”bt”, rel_widths = c(2,7))
rel_widths这个可以设置两个组组合的比例
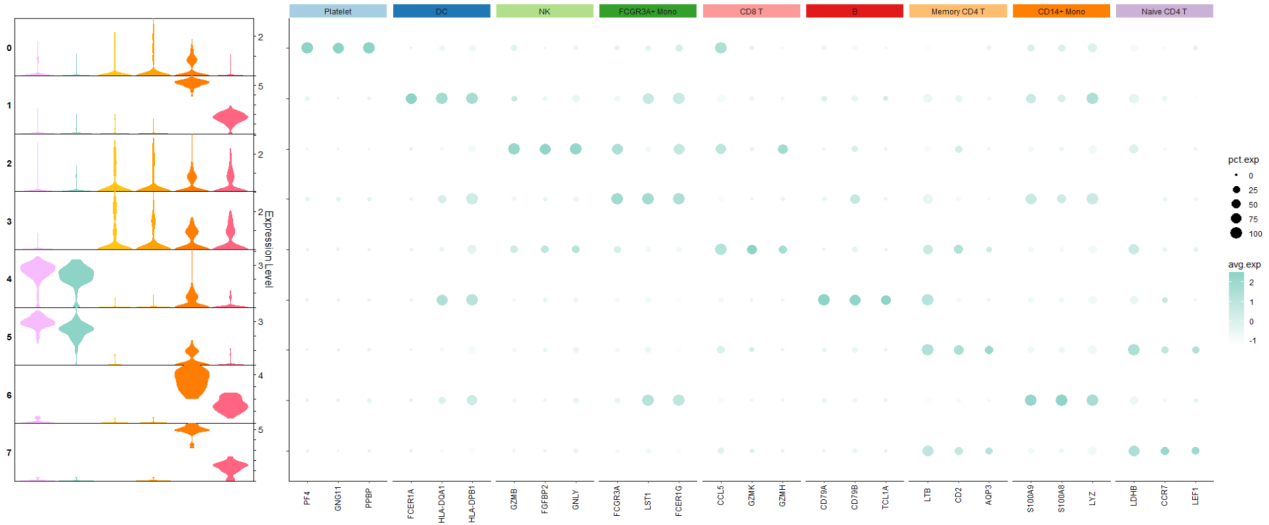
好了,我们的组合图就绘制完成了
对于上述的数据,小伙伴可以使用自己的数据去设置一下,要多多理解代码的意思,其实许多高分文章中的图片都是一步一步组合而来的,理解代码的意思,把自己的图绘制高级起来,小伙伴快去动手试试吧!