
小云最近发现单细胞的热度最近越来越火热了,许多的高分文章都添加了单细胞数据分析的模块。所以,小云便找了几篇高分文献看了看,发现高分文章中,他们对单细胞数据不止分析的很完整,绘制出的图也很好看,并且他们当中的图中的配色也是一大亮点。
小云觉得小伙伴在平时分析单细胞数据的时候,不知道如何绘制的可以学习一下高分文章中的图是如何绘制的。因此呢,今天呢小云在这里手把手的带小伙伴去复现一些高分文章中的图,这次小云看的一篇文章是来自影响因子8.8的《Cellpress》期刊中的《Single-cell RNA sequencing identifies a population of human liver-type ILC1s》的单细胞数据相关,里面作者用了一些图很好看,而且文章中许多的单细胞数据都用一种类型的图去展示,我们来看一下是怎样的图
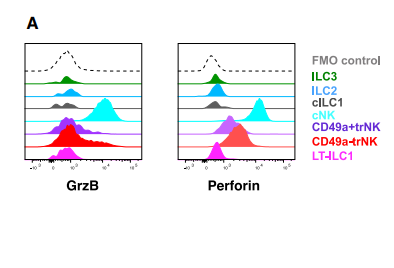
我们从上图可以看出来这是基因与单细胞的关系,横坐标是基因,纵坐标单细胞类型,那我们是不是也可以这样学着绘制一下呢,
有的小伙伴还不知道这是什么类型的图,我们先简单了解一下,小云告诉大家这是山脊图,但是这有很多个“山脊”,也可以叫做堆叠山脊图,原理其实和我们平时简单堆叠小提琴图类似,横坐标一个基因与多个单细胞类型多个关系,那么我们就可以用山脊图去可视化出来,我们利用一下单细胞的数据去学习这个山脊图,好了下面我们去学习一下。

这里呢小云还要说明一下,我们做单细胞分析的时候,操作占用内存可能比较大,所以建议各位小伙伴可以使用服务器去运行,如果没有自己的服务器也没有关系,可以欢迎联系我们使用服务器租赁~
我们先把要需要的包加载一下
#相关R包载入:
library(ggridges)
library(ggplot2)
library(cols4all)
然后导入我们的数据
#数据读入:
df <- read.csv(‘data.csv’,header = T)
head(df)
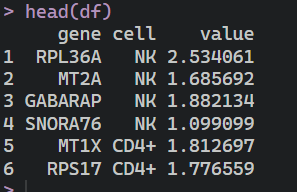
小伙伴可以按照这样的数据格式去自行设置,
这里小云就用一点数据去复现,
我们先看看基础的山脊图
#外轮廓半开放
p <- ggplot(data = df,
aes(x = value, y = cell, fill = cell)) +
geom_density_ridges() +
theme_classic()
p
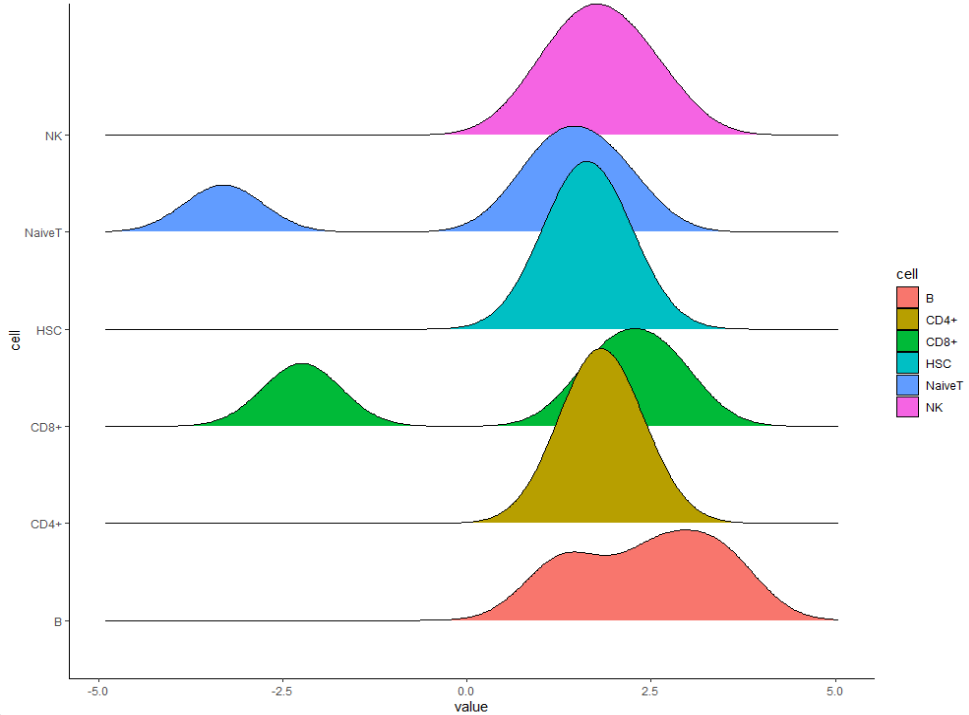
这样的图其实就是基础的绘制,我们需要修改很多的细节,来继续往下走
还有一种展示形式
#外轮廓封闭
pp <- ggplot(data = df,
aes(x = value, y = cell, fill = cell)) +
geom_density_ridges2() +
theme_classic()
pp #
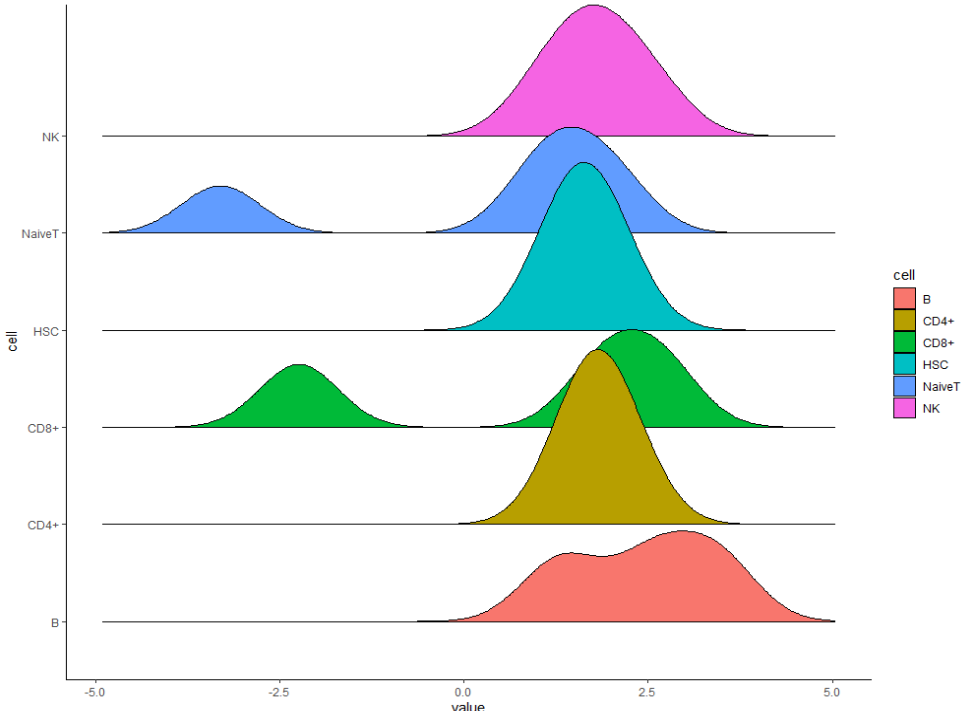
感觉就是函数不同,其他的细节我们就需要用到我们的老朋友ggplot2了
来看一下简单的图形
p1 <- ggplot(data = df,
aes(x = value, y = cell, fill = cell)) +
geom_density_ridges(alpha = 0.8,
color = ‘white’,
rel_min_height = 0.01, #尾部修剪,数值越大修剪程度越高
scale = 1.8, #山脊重叠程度调整,scale = 1时刚好触及基线,数值越大重叠度越高
#quantile_lines = TRUE, #显示分位数线
quantiles = 2 #仅显示中位数线
) +
theme_classic() +
theme(legend.position = ‘none’)
p1

这样一来和文章中有点相似,我们一步一步的修改,保持和文章中类似,我们可以修改一下坐标轴的限制
在这个图的基础上:
#坐标轴限制:
p2 <- p1 +
scale_x_continuous(limits = c(-4, 4),
breaks = seq(-4, 4, by = 2)) #x轴限制
p2
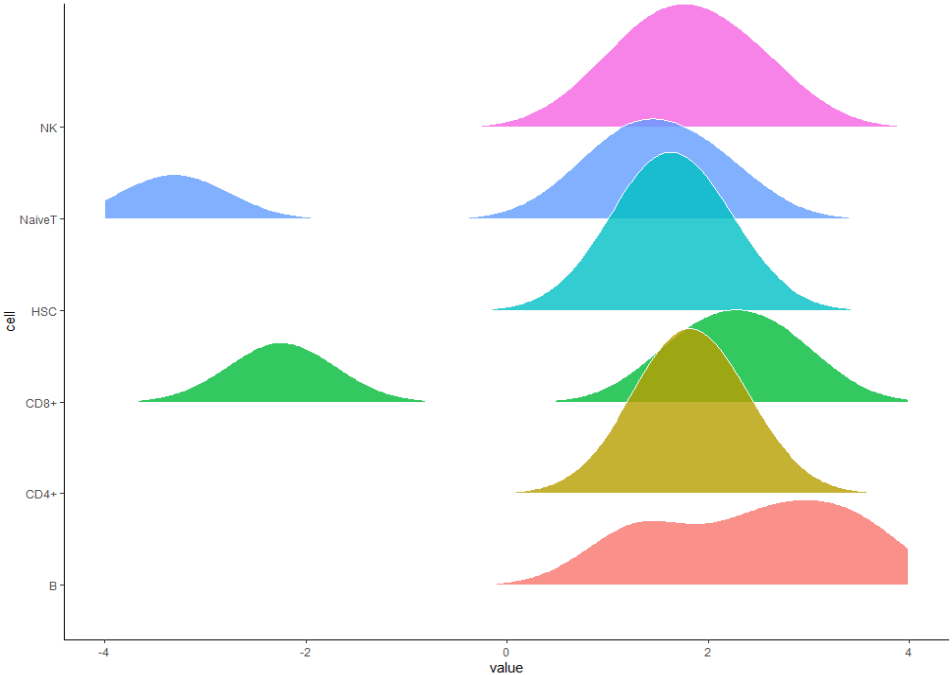
然后把我们一般需要的阈值线再加上去,
#阈值线添加:
p3 <- p2 +
geom_vline(xintercept = c(-3, 3),
size = 0.5,
color = ‘grey’,
lty = ‘dashed’)
p3
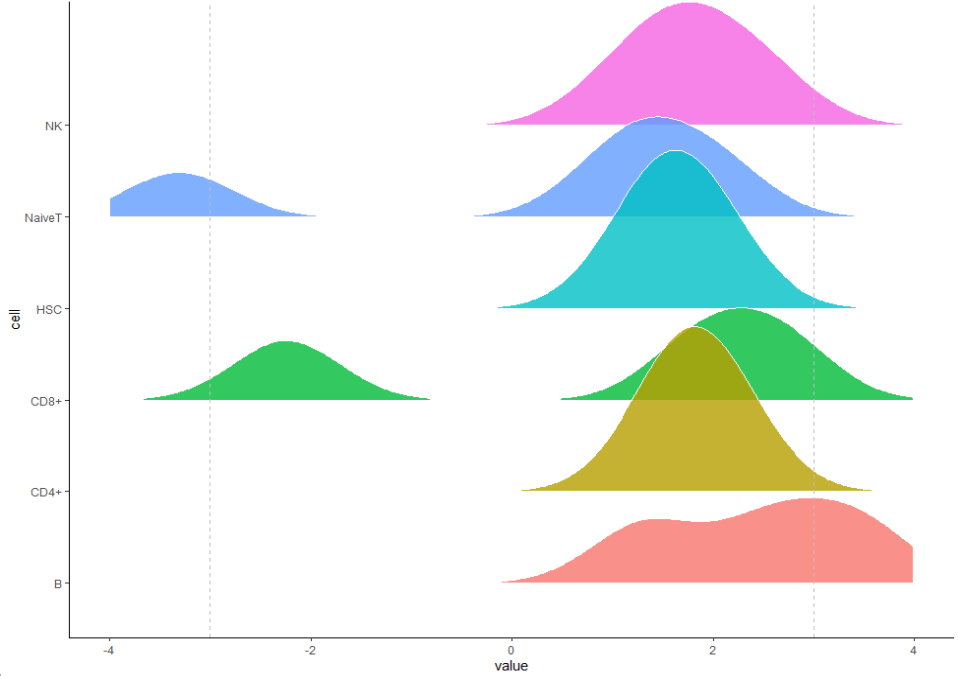
这里小云随机设置一下阈值范围
到这里还有一些细节没修改,我们再去调整一下颜色
这里小云给小伙伴介绍一个调色板
c4a_gui()
我们输入这个调色板
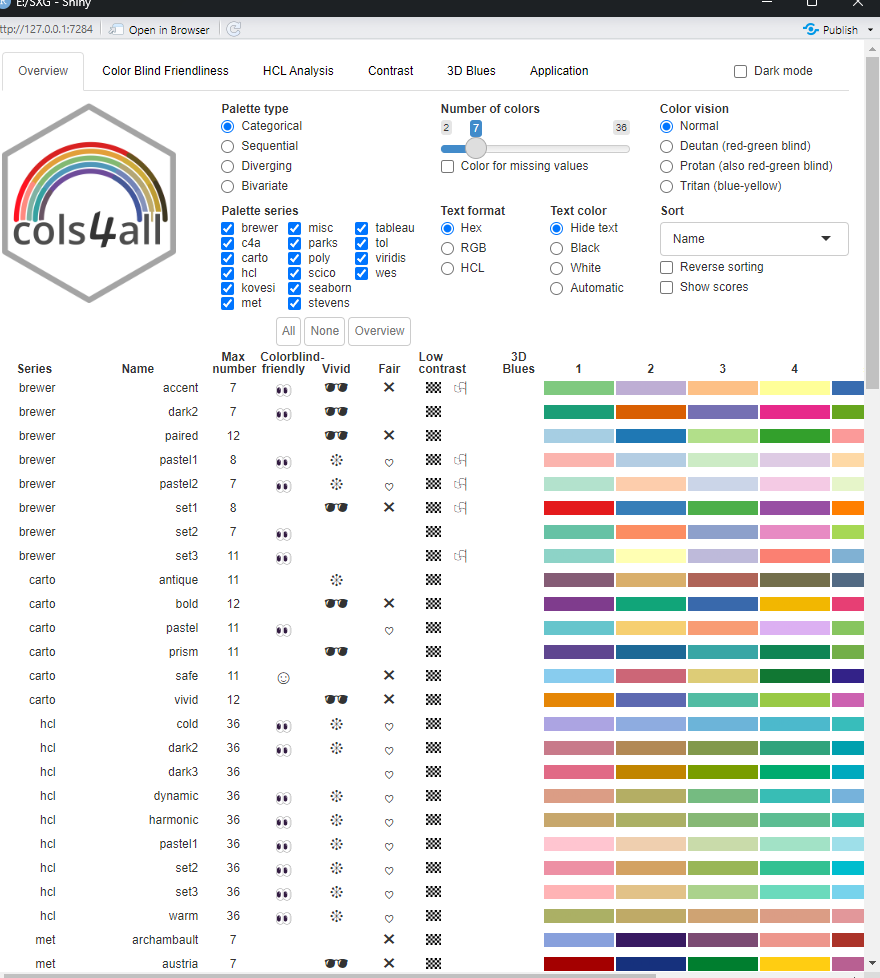
就会跳出来这个页面,我们可以自行去选择调色板的颜色
这里小云选择一个和文章中类似的颜色
mycol <- c4a(‘prgn’,6)
然后我们添加上去,小伙伴可能还有主要到,文章中Y轴字体大小还有颜色也是有变化的,我们把颜色保持和山脊图中颜色一致,在把字体调大,来看一下,
p4 <- p3 +
scale_fill_manual(values = mycol)+
theme(axis.text.y = element_text(colour=mycol))+#y轴文本设置
theme(axis.text.y=element_text(vjust=1,size=16,face = “bold”))#y轴字体大小设置
p4
这些都是在p1的基础上加上去的,
 到这里已经差不多了,看看还有那些细节没有改,还有我们的X轴坐标的字体
到这里已经差不多了,看看还有那些细节没有改,还有我们的X轴坐标的字体
我们在主题设置中加上去 theme(axis.title.x = element_text(
size = 16, lineheight = .9,
family = “Times”
))
来看下
p4 <- p3 +
scale_fill_manual(values = mycol)+
theme(axis.text.y = element_text(colour=mycol))+#y轴字体颜色设置
theme(axis.text.y=element_text(vjust=1,size=16,face = “bold”))+y轴字体大小设置
theme(axis.title.x = element_text(
size = 16, lineheight = .9,
family = “Times”
))
p4
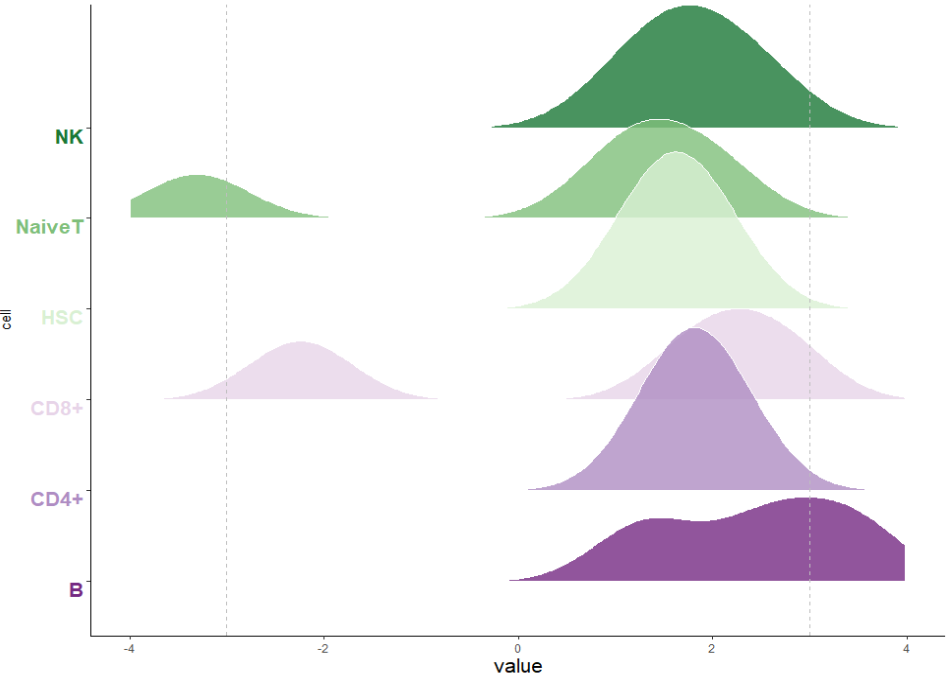
好了,我们到这里就绘制差不多了,但是文章中是多个山脊图绘制在一起的,这就需要我们添加几个山脊图我们可以使用拼图的函数去组合
这里我们在绘制一个和上述一样的山脊图来作为演示,需要注意的是我们需要把左侧的图注删除,文章中是使用一个图注作为演示来看一下,这次我们一步到位
p6 <- ggplot(data = df,
aes(x = value, y = cell, fill = cell)) +
geom_density_ridges(alpha = 0.8,
color = ‘white’,
rel_min_height = 0.01, #尾部修剪,数值越大修剪程度越高
scale = 1.8, #山脊重叠程度调整,scale = 1时刚好触及基线,数值越大重叠度越高
#quantile_lines = TRUE, #显示分位数线
quantiles = 2 #仅显示中位数线
) +
scale_fill_manual(values = mycol)+
theme_classic() +
theme(legend.position = ‘none’)+#图注信息注释
theme(axis.text.y = element_text(colour=mycol))+
theme(axis.ticks = element_blank(), axis.text.y = element_blank())+
theme(axis.title.x = element_text(
size = 16, lineheight = .9,
family = “Times”
))#修改X轴文本信息
p6

下面我们就去组合一下
使用
cowplot::plot_grid(p4, p6,ncol = 2)就可以
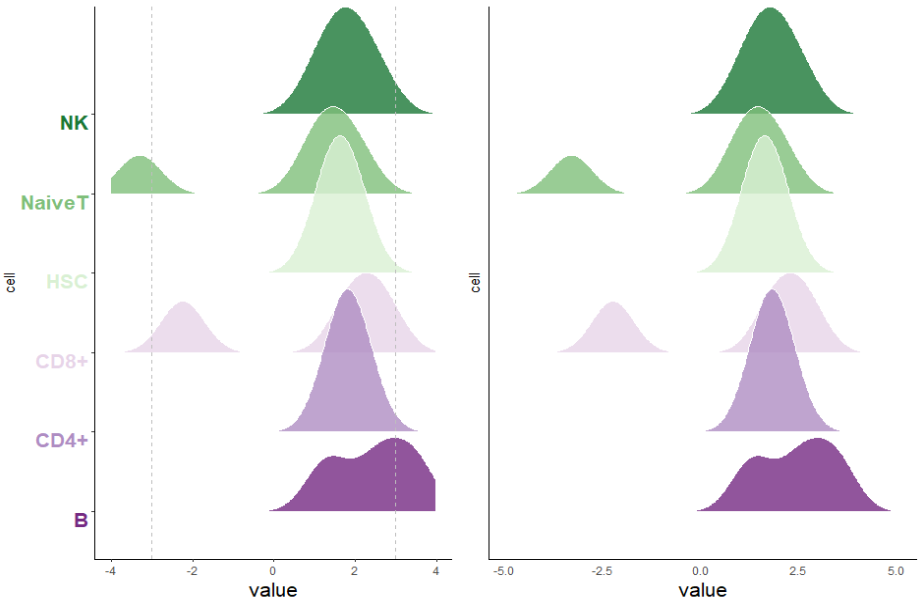
这里小云数据不是很多,所以颜色的区分没有文章中那么多,小伙伴学会了之后可以用自己的数据去复现一下,好了到这里小云就教完了,小伙伴要多多理解代码的意思,这样才能绘制出自己图形,快去动手试试吧!
最后呢,小云给小伙伴带个小福利,如果小伙伴觉得自己运行代码太麻烦,可以试试我们的云生信小工具哟,里面有成熟的代码,只要输入合适的数据就可以直接出想要的图呢。
多多关注我们公众号,云生信!
