毋庸置疑,线粒体是近年的研究热点,特别是和各种细胞死亡相整合,都快杀疯了。这么大个IP,小云也不遗余力的给大家进行了多次推荐。
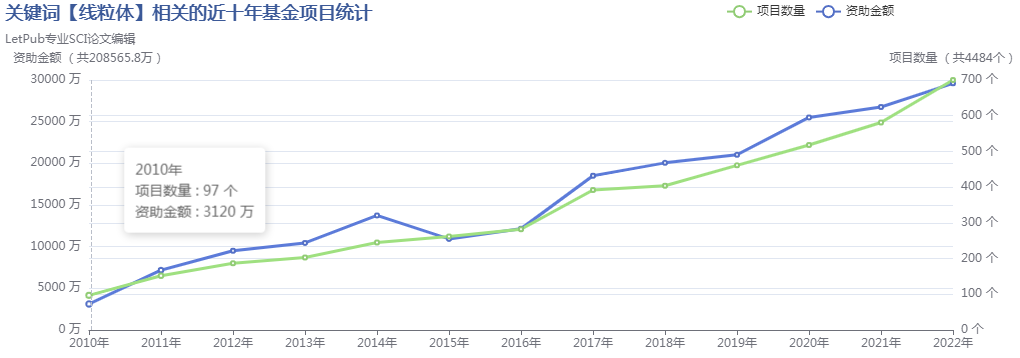
对于尚有迟疑的人,其最大的担心点必然是—我研究的方向是否和线粒体有关?或者说,我这个方向能否挖掘到与线粒体相关的显著差异基因了?
针对这个问题,小云的想法分为2方面:
- 理论层面: 线粒体是细胞中生产能量的主要场所,参与多种细胞生命活动,线粒体稳态对维持正常的细胞功能至关重要。绝大部分疾病的发病和治疗过程均涉及到细胞功能的变化,因此,与线粒体直接或者间接相关的研究数量自然很多。
- 技术层面:线粒体功能复杂,涉及基因数量巨大,能获得显著差异基因概率高。
小云今天找到1篇8分+分析线粒体基因与胃癌的文章,该文章并未将线粒体基因与细胞死亡进行关联,而是从肿瘤微环境、免疫浸润等角度来讨论线粒体基因的功能;同时,该文章列举出了2030个线粒体基因,可谓是最丰富的版本,为大家的分析提供了参考(有需要基因集的粉丝,可以关注公众号回复获取)。下面,我们一起看看这篇文章吧(如果没有分析思路或者文献复现有困难,可以找小云,超多创新性高的分析思路和分析服务供你选择!)。


题目:构建线粒体相关基因特征以评估肿瘤免疫微环境并进行胃腺癌的生存预测
杂志:Journal of translational medicine
影响因子:8.44
发表时间:2023年3月
![]() 数据信息
数据信息 ![]()
| 数据集/队列 | 数据库 | 数据类型 | 样本信息 |
| TCGA-STAD | TCGA | RNA-seq和临床信息 | 317 例胃腺癌和29个健康对照 |
| GSE15459 | GEO | 芯片数据和临床信息 | 182 例胃腺癌患者 |
| GSE66229 | GEO | 芯片数据和临床信息 | 150对胃腺癌和癌旁组织 |
![]() 研究思路
研究思路 ![]()
基于TCGA和GEO数据库中的STAD表达谱及芯片数据,利用“limma”包获得差异表达基因(DEGs);将DEGs与线粒体相关基因取交集,获得2031个差异表达的线粒体基因;通过单变量Cox回归、Lasso回归分析和多变量Cox回归分析筛选线粒体相关基因,构建新的预后相关基因集,利用基因集将患者分为高风险组和低风险组;对高/低风险组患者的差异基因进行GO、KEGG和GSEA富集分析;基于“ESTIMATE”包计算高/低风险组患者的肿瘤微环境分数和免疫评分;使用R包的CIBERSORT算法计算高/低风险组患者中22种肿瘤浸润免疫细胞(TIIC)的丰度;使用R包“pRRophetic”计算患者对138种化疗/靶向疗法药物反应的能力;最后利用qPCR法检测4种线粒体相关特征基因在临床患者和细胞系中的表达(图1)。
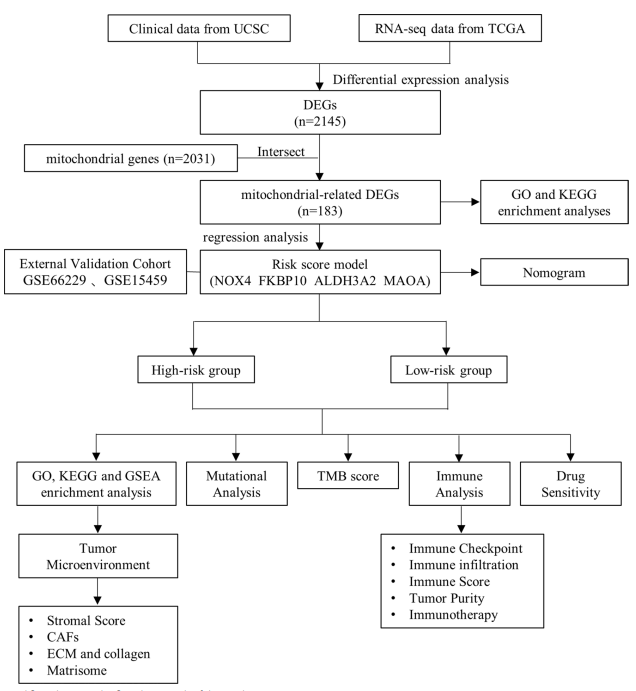
图1,本研究的分析流程图
![]() 主要结果
主要结果 ![]()
- STAD中与线粒体相关的DEGs的鉴定和风险模型构建
对上述3个数据集进行DEG(差异表达基因)分析,共获得2145个可编码的DEGs(图2A);进一步将GSEA中筛选出的线粒体相关基因和上述DEGs进行了联合分析,共筛选出STAD中的183个候选线粒体相关DEGs(图2B);通过单因素Cox回归分析筛选出19个基因作为STAD患者预后的潜在危险因素(图2C)。利用LASSO回归分析、多变量Cox回归分析最终确认出4个备选基因(图2D,E)。与正常组织相比,分别在肿瘤样本中观察到较高的NOX4和FKBP10表达以及较低的ALDH3A2和MAOA表达(图2F)。(ps:差异基因分析和火山图可以用小云新开发的零代码生信分析小工具实现,云生信分析工具平台包含超多零代码分析和绘图小工具,上传数据一键出图,感兴趣的小伙伴欢迎来尝试哟,网址:http://www.biocloudservice.com/home.html)。
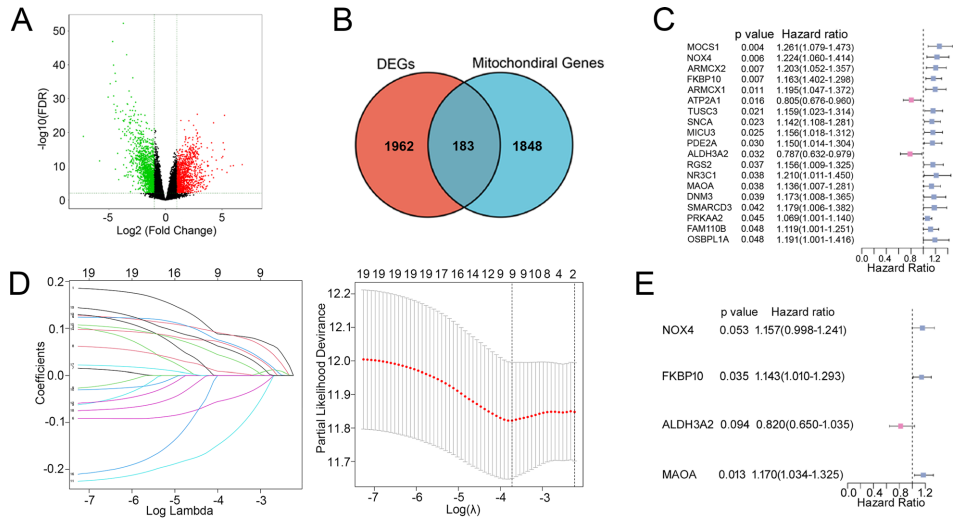
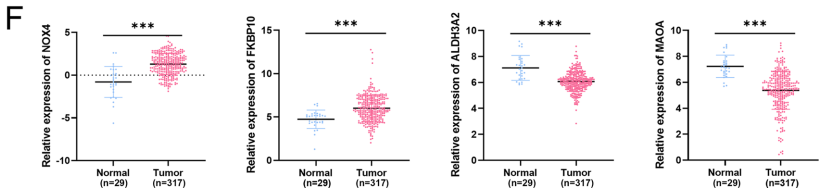
图2.STAD中与线粒体相关的DEGs的鉴定和风险模型构建
利用患者的4个线粒体相关DEGs基因集的中位风险评分,将患者分为高风险和低风险亚组。K-M曲线显示高危组患者的OS较差(P =0.0009,图3A)。ROC曲线证明了预后风险模型的准确性(图3B)。风险评分与生存时间、生存状态和风险等级之间的关系证明了风险模型的预后作用(图3C)。GSE66229和GSE15459数据集进一步验证了预后风险模型的稳健性(图3D)。在GSE66229数据集中,1年、3年和5年生存率的ROC曲线的AUC值分别为0.620、0.625和0.601(图3E)。
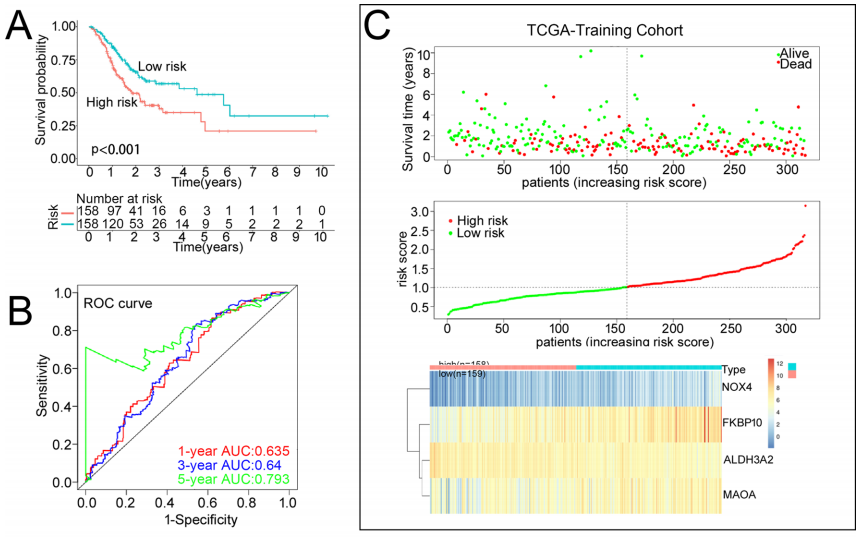
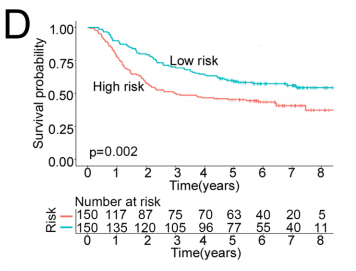

图3.与线粒体相关的DEGs风险模型构建和验证
- 高危和低危人群DEGs功能富集分析
对高风险和低风险患者组的298个DEG进行了GO分析,发现差异基因主要富集在含胶原蛋白的ECM(细胞外基质)和胶原蛋白三聚体中(图4A)。KEGG分析得到的前10条通路依次为:蛋白质消化吸收、癌蛋白多糖、粘着斑、人乳头瘤病毒感染、PI3K-Akt信号通路、ECM-受体相互作用、细胞粘附分子、轴突导向、cAMP信号通路、血管平滑肌收缩(图4B)。GSEA结果表明,风险评分与高风险组的ECM糖蛋白、核心基质体、ECM组织显着相关(图4C)。
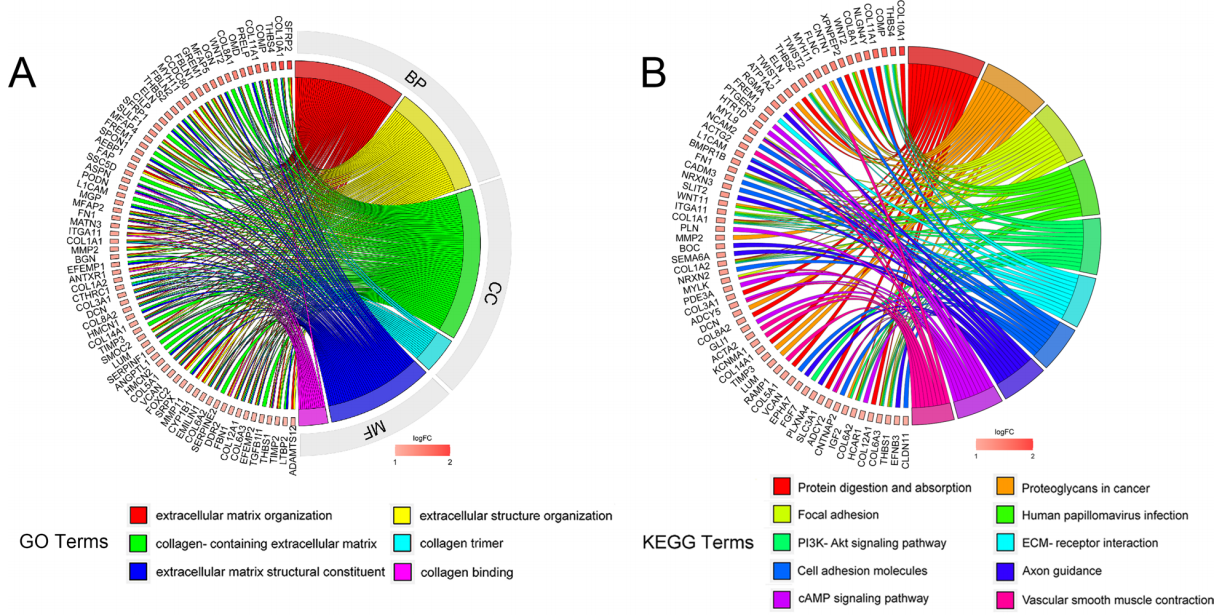

图4.高危和低危人群DEGs功能富集分析
- 线粒体相关风险评分与 STAD 中的免疫特征和免疫治疗反应相关
对高风险和低风险患者组的免疫细胞浸润进行分析,发现高危组幼稚B细胞、调节性T细胞(Tregs)、M0巨噬细胞和M2巨噬细胞含量显着升高(图5A)。ESTIMATE结果显示,与低危组患者相比,高危组患者的免疫评分更高,肿瘤纯度显著降低(P=0.370)(图5C、D)。由此,作者推测激活的CD8+T细胞特征与基质体和CAF特征呈负相关,提示肿瘤免疫抑制微环境可能导致高危组STAD患者预后较差。分析发现,43个免疫检查点在高风险组中受到显着调节(图5E)。
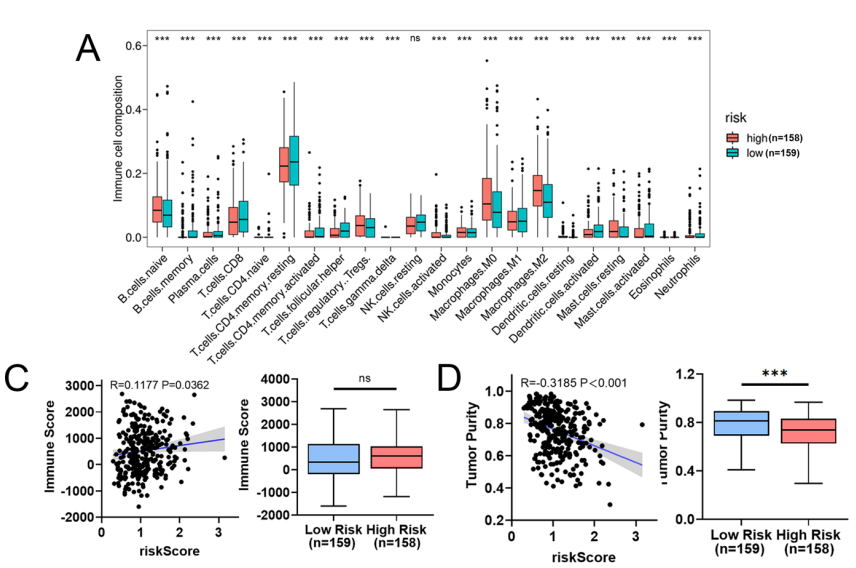
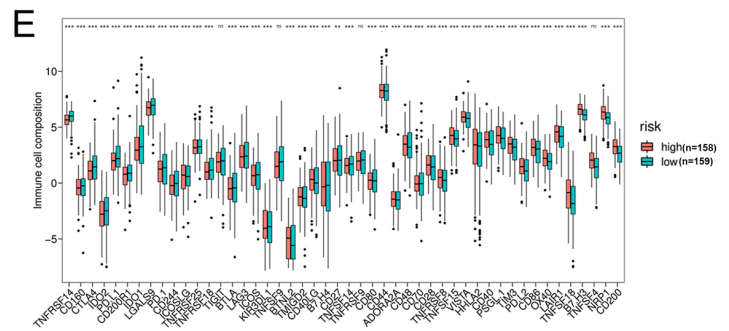
图5线粒体相关风险评分与 STAD 中的免疫特征和免疫治疗反应相关
作者进一步使用TIDE算法来评估基于线粒体相关基因的风险评分在预测STAD免疫治疗反应中的能力。结果表明,高危组的免疫治疗反应率明显低于低危组(图 6A)。应用PRJEB25780队列(PD-L1抑制剂治疗45名晚期胃癌患者)来验证线粒体相关风险特征的结果与TIDE的预测结果一致(图6B)。在免疫评分较低的亚组中,低危组的免疫治疗反应率明显高于高危组,表明将患者的风险评分和免疫评分相结合是一种更有效的预测方法(图6C、D)。
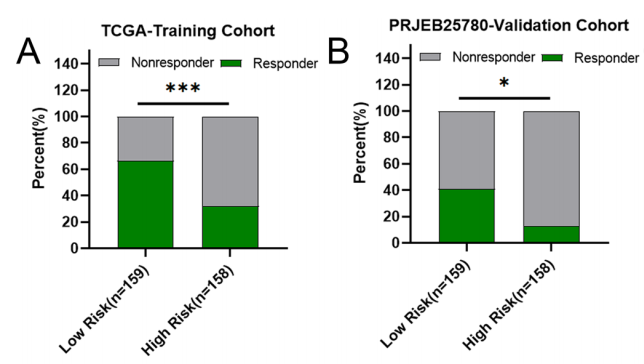

图6线粒体相关风险评分与 STAD 中的免疫特征和免疫治疗反应相关
- 不同风险评分STAD患者对治疗效果的预测
作者预测了138种药物(包括化疗、靶向治疗和免疫治疗)对不同风险评分组STAD患者的效力,发现高风险组患者可能对雷帕霉素、PD-0325901、达沙替尼等更敏感,而低风险组患者可能对AZD7762、CEP-701、甲氨蝶呤等更敏感,可为临床治疗提供可靠参考(图7)。 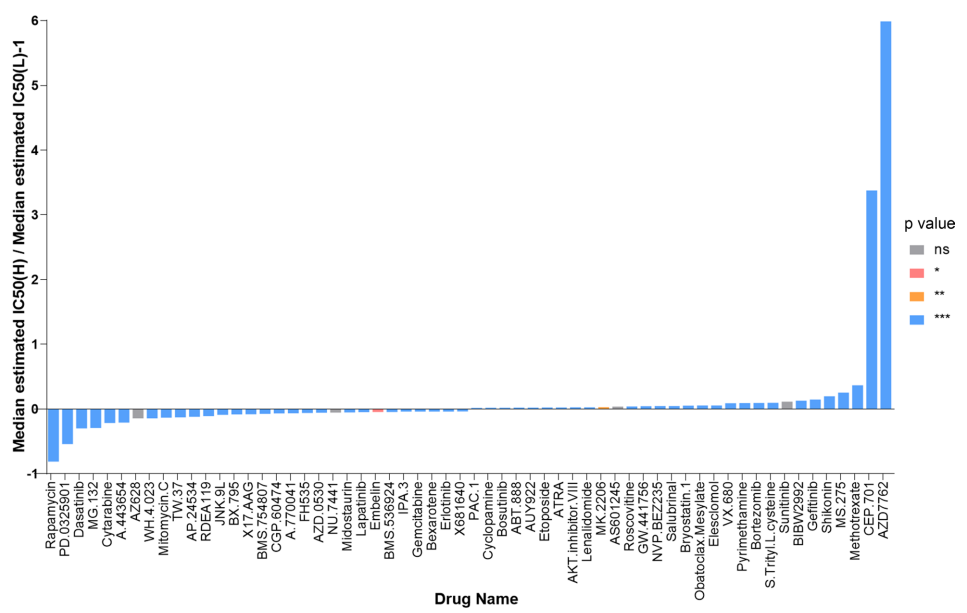
图7. 不同风险评分STAD患者对治疗效果的预测
- 对STAD中线粒体相关hub基因的实验验证
作者收集了10对STAD患者的癌与癌旁组织进行了qRT-PCR。如图8A所示,STAD肿瘤组织中NOX4和FKBP10的表达分别显着高于成对的相邻正常组织;ALDH3A2和MAOA在STAD肿瘤组织中的表达分别显着低于成对的相邻正常组织。相比于正常胃上皮细胞,上述4个基因在胃癌细胞系中的表达量与临床组织样本中均一致(图8B),表明了这四个线粒体相关基因的可靠性。
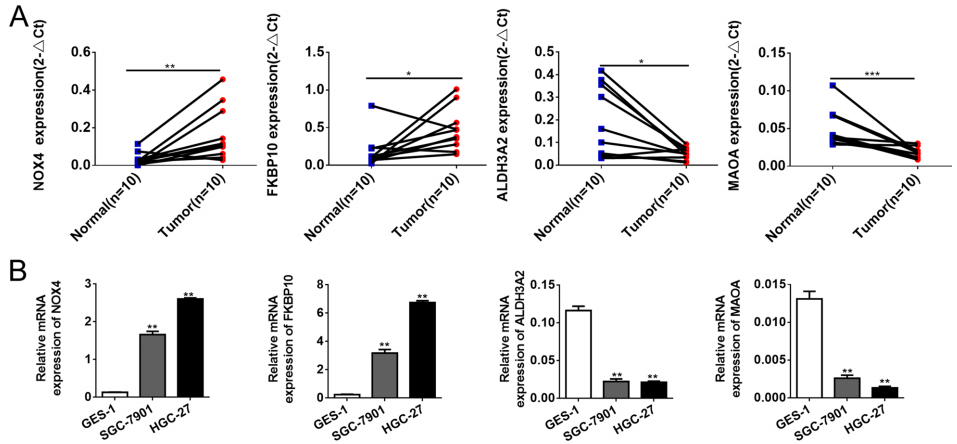
图8. 对STAD中线粒体相关hub基因的实验验证
![]() 总结
总结 ![]()
这篇文章的分析内容比较丰富,主要的特征是在筛选线粒体相关基因后,并未将线粒体基因与细胞死亡(铁死亡、自噬、铜死亡)进行关联,而是从线粒体相关基因与肿瘤微环境、免疫浸润等角度来讨论,且文章给出的2030个线粒体基因相对完整,可用于大家后续的分析。文章最后还加入了验证试验,这种干湿结合的方式可以学习起来,是现在发文的趋势哦!
(https://mp.weixin.qq.com/s/YGCWkrgAJXt-IxiC_s-2IQ)
Nature重磅综述加持,线粒体方向生信依然坚挺,8分+文章一举拿下,妥妥的发文和申课题利器!
一分钟告诉你如何利用机器学习筛选线粒体相关诊断标志物,非肿瘤疾病关联免疫浸润,7分+文章你也能拥有!
还没找到生信分析思路?看这里,线粒体自噬方向,4分+保姆级纯生信分析模板套路,拿走不谢
双热点分析思路再度来袭!铁死亡+EMT强强联合,常规预后模型的纯生信也能轻松入手8分+!速围观~
不用做实验的8分+TNF基因家族纯生信思路!两次聚类+风险评分“老药新用”的玩法!基因集已备好,就等你来复现~

