
云服务器租赁
(加微信备注99领取试用)

题目:耐免疫治疗的微卫星不稳定性高胃肠癌患者肠道微生态组的多组学研究
杂志:Cell Reports Medicine
影响因子:IF=14.3
研究背景
dMMR亚型患者无法识别和修复某些自发突变,导致较高的肿瘤突变负担和微卫星高度不稳定(MSI-H)状态,因此更可能受益于抗PD-1/PD-L1免疫治疗。然而,对于MSI-H/dMMR胃肠道癌症患者,他们对免疫治疗的反应存在很大的异质性,大约30%的患者表现出原发性耐药性。因此,本研究旨在通过多组学方法,包括微生物组、血液代谢物组和细胞因子/趋化因子组的分析,探索微生物组在MSI-H胃肠道癌症患者中的作用,以及其与免疫治疗耐药性的关联。研究结果有助于深入了解微生物组在药物耐药性中的重要作用,并为未来的预防性诊断和治疗提供新的思路。
数据来源
这篇文章使用了多种数据进行多组学分析(表1)。研究人员对患者的肠道微生物进行了宏基因组测序以及对患者样本进行了RNA-seq,还对患者的血浆样本进行了代谢组分析等。
表1 研究使用到的数据信息及来源
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
本研究的研究思路是通过多组学分析来研究MSI-H胃肠道癌症患者的肠道微生物生态系统(图1)。研究人员检测了77名晚期MSI-H/dMMR胃肠道癌症患者在治疗前和免疫治疗期间的肠道微生物组、血液代谢组以及细胞因子和趋化因子,并对多组学数据进行了相关分析。此外,还在小鼠模型以及另一个独立队列数据集中进行验证。
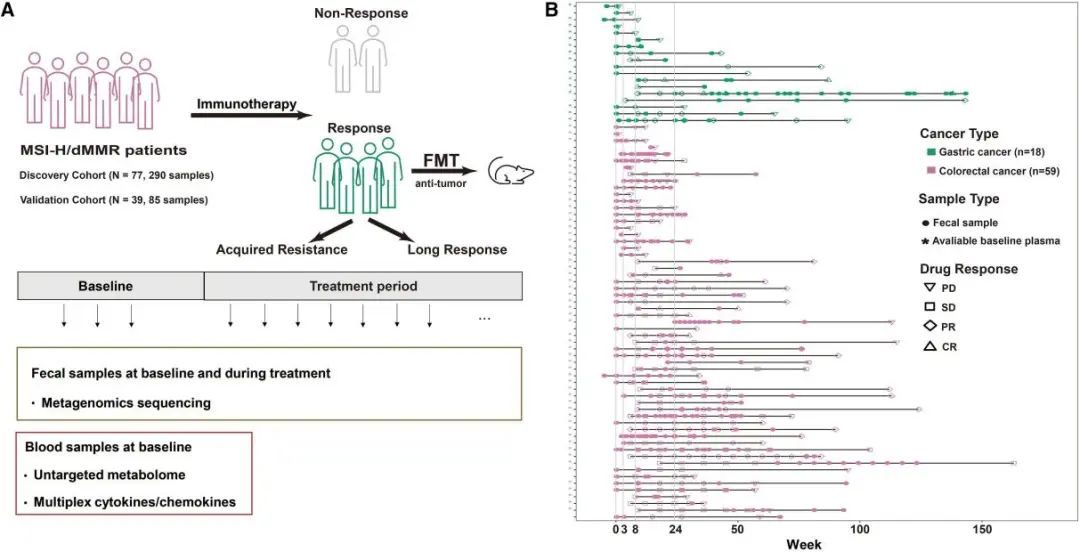
图1 研究摘要
主要结果
研究人员对98例MSI-H/dMMR的胃肠癌患者进行数据收集和分析,包括30例胃癌患者和68例结直肠癌患者。其中,由于治疗方法和没有足够样本进行宏基因组测序而去除21例患者。最终的队列包括77名患者。研究人员观察了这些患者的治疗反应和临床特征,包括治疗前后的粪便样本和血液样本分析,用以评估抗PD-1/PD-L1治疗在这些患者中的疗效和影响因素。
研究人员对基线(治疗前或免疫治疗后不超过3周)样本进行分析已探究免疫治疗原发性耐药性相关的全局特征。研究人员分析了对免疫治疗有响应组(R)和无响应组(NR)的α多样性和β多样性,结果显示没有显著差异(图2A)。接着,研究人员进行了MaAsLin2分析,显示一些细菌物种在两组患者中存在显著差异(图2B)。上述结果在纵向样本的时间序列分析和对基线样本的独立验证队列的分析得到了验证。
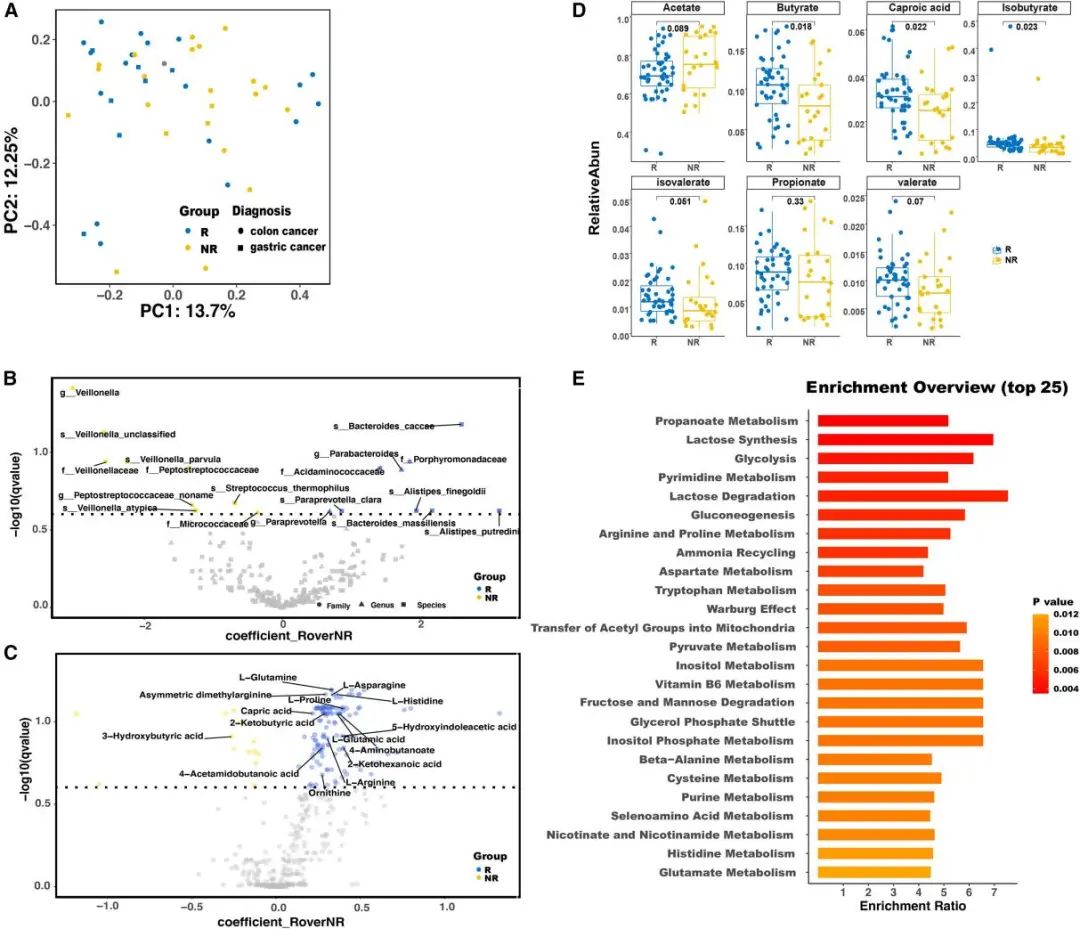
图2 与药物反应相关的肠道微生物组组成、功能途径和血液代谢物
研究人员利用液相色谱-质谱法(LC-MS)对不同免疫治疗响应的患者的血浆样本进行代谢组学分析。结果共鉴定出143种代谢物在在两组患者间有差异(图2C)。其中,与治疗反应性相关的代谢途径主要涉及精氨酸代谢和短链脂肪酸代谢(SCFA)。精氨酸代谢相关的代谢物与肠道微生物存在关联。在对免疫疗法具有原发性耐药性的患者中,丁酸、己酸和异丁酸等短链脂肪酸富集,而丙酸代谢通路也显示出显著性(图2C)。
研究人员通过将六名免疫治疗患者的粪便样本移植到小鼠模型中,验证肠道菌群在免疫治疗反应中的作用(图3A)(PS:干湿结合是现在发文的最佳方法,大家要学会给自己的课题项目设计这样的套路。)。结果显示,接受对免疫治疗有响应患者粪便移植的小鼠肿瘤大小显著减小,并且肿瘤微环境中的T细胞亚群比例增加(图3B-D)。研究人员接着对小鼠血液样本进行代谢组学分析以及对肿瘤组织进行RNA-seq分析以揭示有响应组和无响应组之间的代谢和基因表达差异。结果共鉴定出97种差异代谢,并发现精氨酸代谢可能影响免疫检查点抑制剂的反应,而精氨酸代谢与Treg细胞的关联提示了其调节免疫功能的潜在作用(图3E-G)。
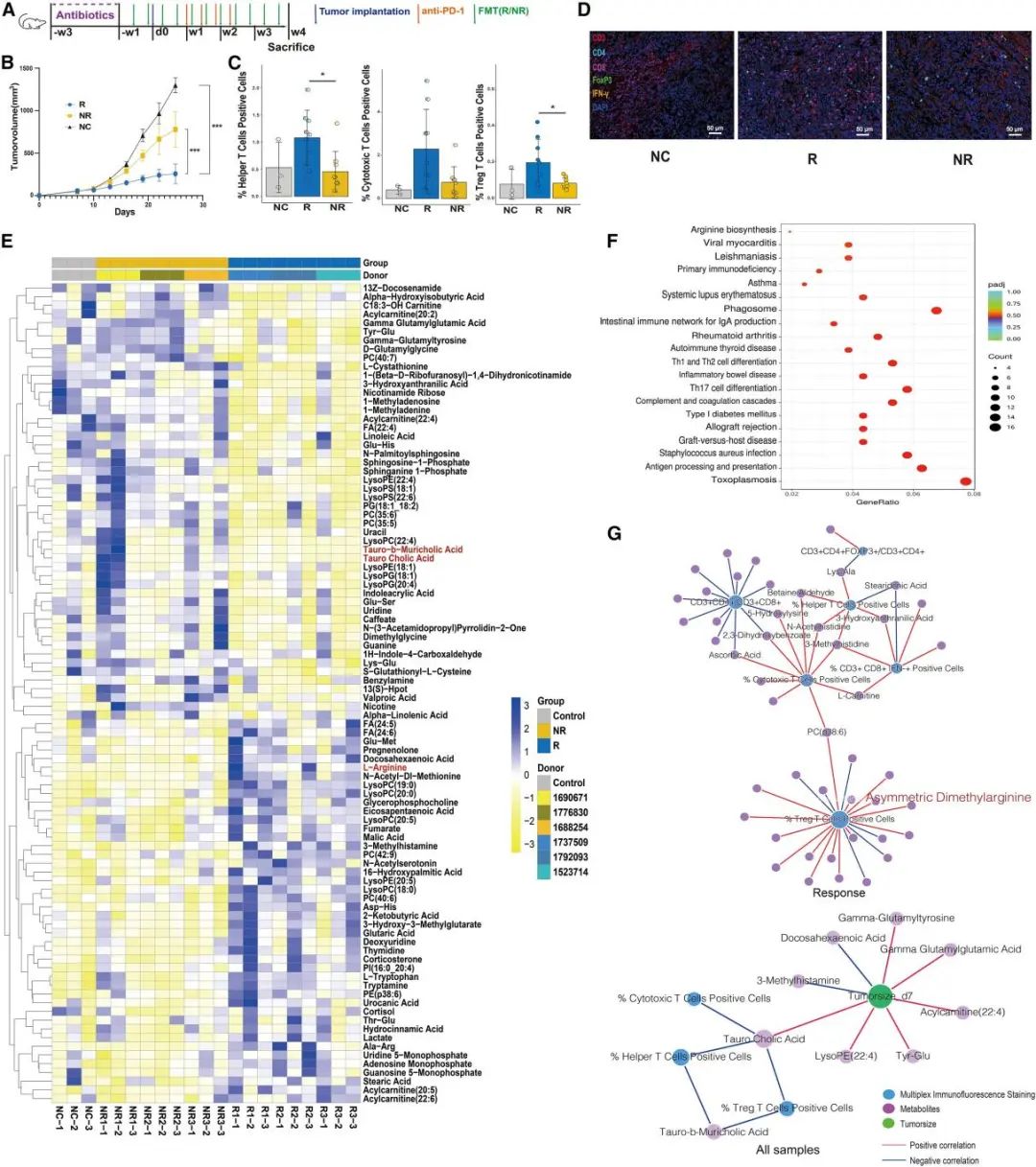
图3 用来自Rs的粪便进行粪便微生物群移植(FMT)后,小鼠中的抗肿瘤作用增强
研究人员建立了一个机器学习模型,利用肠道菌群数据预测免疫治疗的效果。该模型在训练集、测试集和独立验证集上表现良好,预测曲线下面积分别为0.9、0.88和0.79(图4A)。通过分析特征的贡献,研究人员确定B. caccae、V. parvula、V. atypica和Clostridiales bacterium等肠道菌群可以作为预测免疫治疗反应的特征(图4B)。这些结果表明肠道菌群可能成为预测免疫治疗效果的潜在指标,并且选定的微生物物种可能是预测治疗反应的生物标志物。
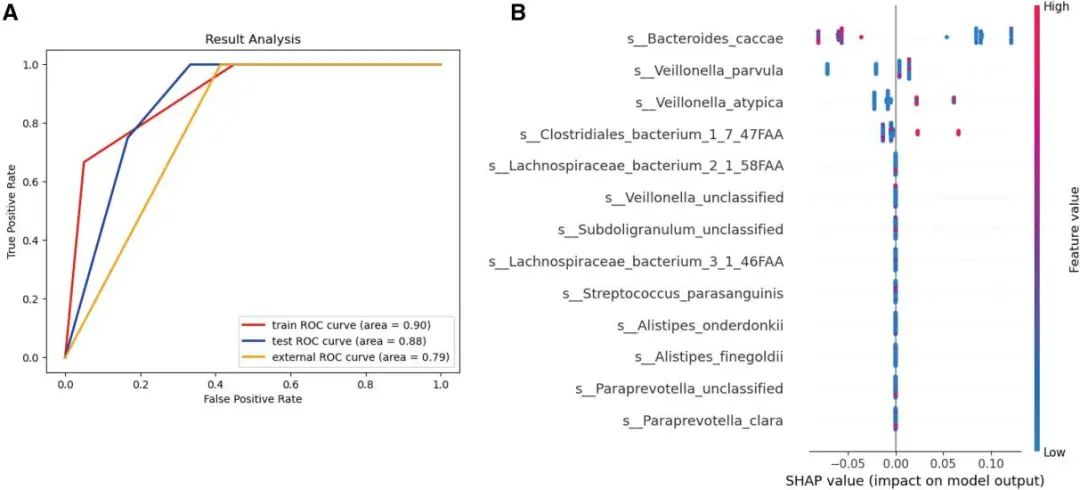
图4 基于微生物生物特征的机器学习模型
在临床实践中,研究人员发现一些患者出现了对免疫治疗的获得性耐药(AR)以及在治疗后持续受益1年以上且没有疾病进展的长期受益患者(LRs)。研究人员对两组患者进行了基线多组学分析(图5)。研究发现,与药物耐药相关的18种菌种,其中包括两种Alistipes菌种在AR样本中减少,同时也在原发性耐药组(NRs)中减少。在耐药过程中,其他一些菌种也出现了显著变化。对比NR和AR基线时的微生物组成差异以及治疗过程中的动态变化,研究人员还发现一些微生物的数量变化与治疗反应相关。
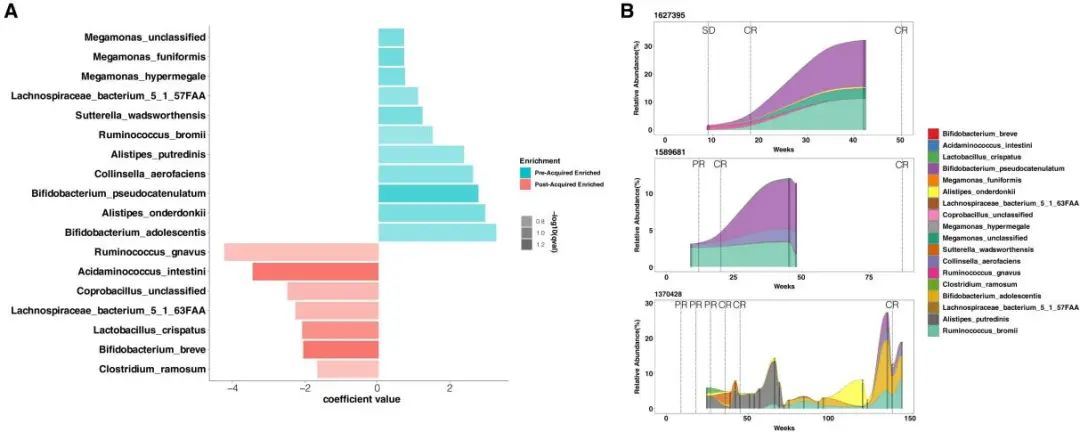
图5 与AR相关的肠道微生物组
文章小结
本篇文章通过对具有微卫星不稳定性高的胃肠癌患者的肠道微生物组、血液代谢组和肿瘤组织的多组学分析,探索了与免疫治疗抵抗相关的生物标志物和机制,为深入理解免疫治疗抵抗机制提供了重要的线索。同时,文章采用了多种先进的统计分析方法和机器学习模型,值得学习和借鉴。相信看完这篇文章后,大家对多组学联合分析的分析方法和内容会有更深的认识和理解。如果有想法,那小编还是觉得大家应该早日行动,毕竟单组学能卷成多组学,以后多组学卷成什么样可就不好说了。当然,如果你对其中的分析方法和统计方法有疑问,可以随时来和江江包沟通交流。
参考文献doi:https://doi.org/10.1016/j.xcrm.2023.101355
原文始发于微信公众号(云生信学生物信息学):秀出实力!北京大学肿瘤医院沈琳、彭智团队:肠道菌群中的多组学分析一举斩获14+Cell子刊