
各位小伙伴们,深度学习火到生信领域不是没有理由的呀,基础模型拿过来就能发文章!再加上小途平日分享的加分小点子,高分成果还不是手到擒来?(上分第一步!关注小云,打开新视界!)今天小云给大家带来一篇深度学习结合机器学习方法,并以生信分析做点缀,结合单细胞数据挖掘来实现候选基因筛选和药物发现的新思路,数据来源于公共数据库。文章的思路打开,而且做到了很好的效果。更重要的是,作者从投稿到见刊只需要一个多月!(新点子新技术还不快收入囊中?让我们一起跟随小云,探索这篇文章的精彩之处吧~)
1,本文中采用GEO公共数据库构建了一个全面的透明细胞肾癌的肿瘤微环境景观,涵盖了其肿瘤相关巨噬细胞、内皮细胞、肿瘤相关成纤维细胞和T细胞的异质亚群;
2,本文中通过结合分子特征模型DMPNN和最优机器学习分类器GBDT等方法,来预测特定于EPAS1蛋白的潜在化合物,实现了最优性能;
3,本文中通过模型集合单细胞数据挖掘分析,识别出关键转录因子蛋白EPAS1,该因子调节涉及调整缺氧机制的基因表达;
4,本研究最终筛选出五种候选化合物,为旧药新用和开发新的ccRCC治疗药物提供了重要的理论基础。想要发表高分SCI的小伙伴们也可以借鉴一下本文的研究思路哦~ ps:如何更好的将深度学习完美复现并应用到临床医学药学相关领域呢?有兴趣的小伙伴快扫码交流吧!

云服务器租赁
(加微信备注99领取试用)

题目:使用深度学习算法和单细胞分析促进透明细胞肾癌特异性药物发现
杂志:Int J Mol Sci
影响因子:IF=5.6
发表时间:2024年4月
研究背景
肾细胞癌(肾癌)是泌尿系统中常见且恶性程度高的肿瘤,主要起源于肾实质尿管上皮,被称为肾腺癌,占肾脏恶性肿瘤的80%至90%。透明细胞肾癌(ccRCC)是最常见的肾癌类型,治疗包括肾切除和消融,但早期免疫治疗和化疗的效果有限。近年来,新的靶向治疗和免疫治疗方法已被开发,但面临药物抗性和肿瘤异质性的挑战。肿瘤的发生、生长和转移与其肿瘤微环境(TME)的复杂网络密切相关,系统地研究ccRCC的TME特性对开发新的治疗策略至关重要。同时,药物开发是一个复杂且成本高昂的过程。为此,作者使用公共数据库中的scRNA-seq数据基于深度学习和机器学习算法进行了候选基因筛选和靶向药物发现研究。
数据来源
|
数据集 |
数据库 |
详细信息 |
|
6个ccRCC样本 |
GEO |
31625个单细胞转录组数据:GSM4735364、GSM4735366、GSM4735368、GSM4735370、GSM4735372和GSM4735374, |
|
化合物数据 |
ChEMBL、MCE |
用于选择临床靶点和深度学习-机器学习集成算法 |
研究思路
作者使用了GEO数据库中六名患者的单细胞RNA测序(scRNA-seq)数据,描述了TME的特征。通过与TME组分(如T细胞、肿瘤相关巨噬细胞和内皮细胞)相关的不同转录组模式,作者识别并筛选了150个与ccRCC发展和伪分化相关的候选基因。随后,通过与转录因子(TF)数据库对比,作者确定了三个与肿瘤功能状态和免疫微环境相关的关键转录因子作为ccRCC抑制剂的候选靶点。经过对它们的蛋白结构分析,选择EPAS1/HIF-2α进行虚拟筛选,作为抗ccRCC治疗的药物靶点,使用一种基于神经网络的分子特征模型(DMPNN)和一个最优机器学习分类器(GBDT)来预测特定于EPAS1蛋白的潜在化合物。使用Schrodinger Maestro 11.4软件进行虚拟筛选和PyMol进行三维映射后,作者发现了五种潜在的ccRCC特异性化合物,包括两种FDA批准的药物。最终,作者进行了分子动力学分析,以进一步证明EPAS1蛋白与这两种药物之间的相互作用机制。
主要结果
1. scRNA-seq分析ccRCC中细胞类型聚类
从GEO数据库下载的转录组数据经过了碱基调用、比对、过滤和标准化处理。经过进一步的质量控制,仅保留每个指标中的高质量细胞(图1)。
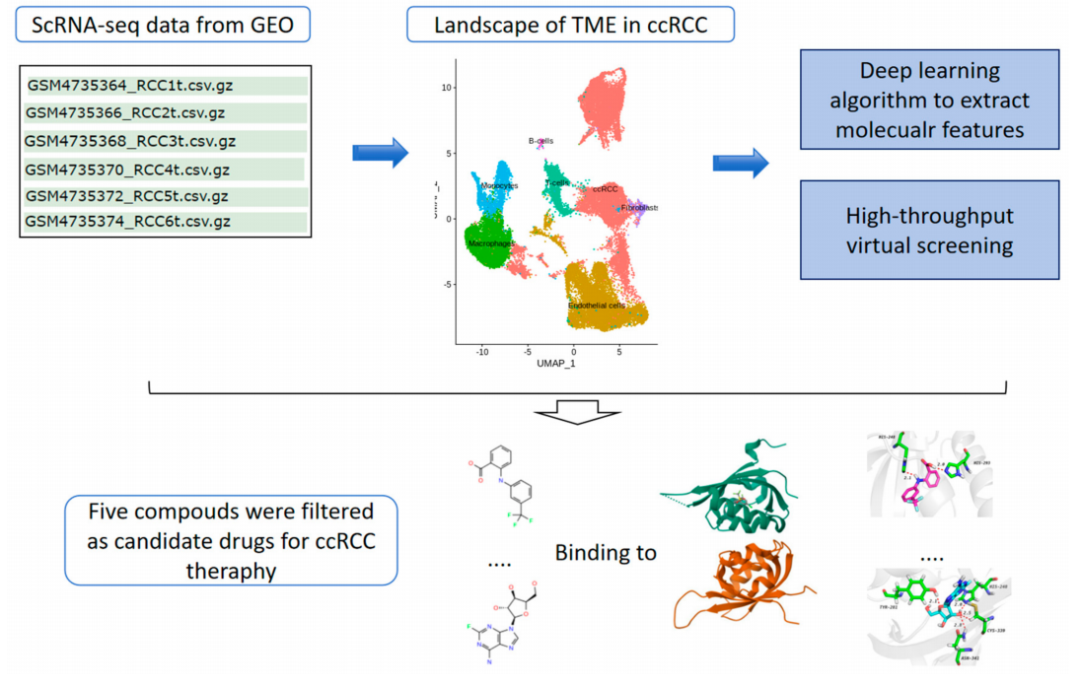
图1 本研究中使用的方法和过程的工作流程
经过Seurat(R包)处理后,从ccRCC患者中获得了31,625个高质量的单细胞转录组数据点。基于标记基因的表达水平反映,这些细胞被分类为五大亚型:ccRCC肿瘤细胞、与TME相关的细胞、T细胞、内皮细胞(EC)、癌相关成纤维细胞(CAF)和肿瘤相关巨噬细胞(TAM)。在探索ccRCC中TME时,发现恶性细胞与免疫和非免疫细胞相互作用,形成这一复杂的生态系统(图2)。作者随后将这些细胞分离出来进行进一步研究。总之,作者通过高质量的单细胞转录组数据,深入分析了ccRCC肿瘤微环境中的细胞异质性和细胞相互作用。

图2 细胞聚类分布和不同细胞亚群的标记基因热图
2. 在巨噬细胞中发现的四种亚群
巨噬细胞是先天免疫的关键组成部分,能应对病原体并通过抗原呈递参与适应性免疫。在肿瘤发展中,它们以TAM的形式存在。本研究发现了两种TAM亚型,并通过Monocle2重建了它们的细胞分化演化轨迹。结果表明,M1型TAM具有抗肿瘤特性,而M2型TAM则可能促进肿瘤生长和免疫抑制。此外,研究还识别了影响TAM分化的关键基因ACTB、CAPG和STXBP2,并进一步分析了相关基因的表达模式(图3)。这些发现为理解TAM在肿瘤微环境中的角色提供了深入见解,为开发针对性治疗策略提供了可能。
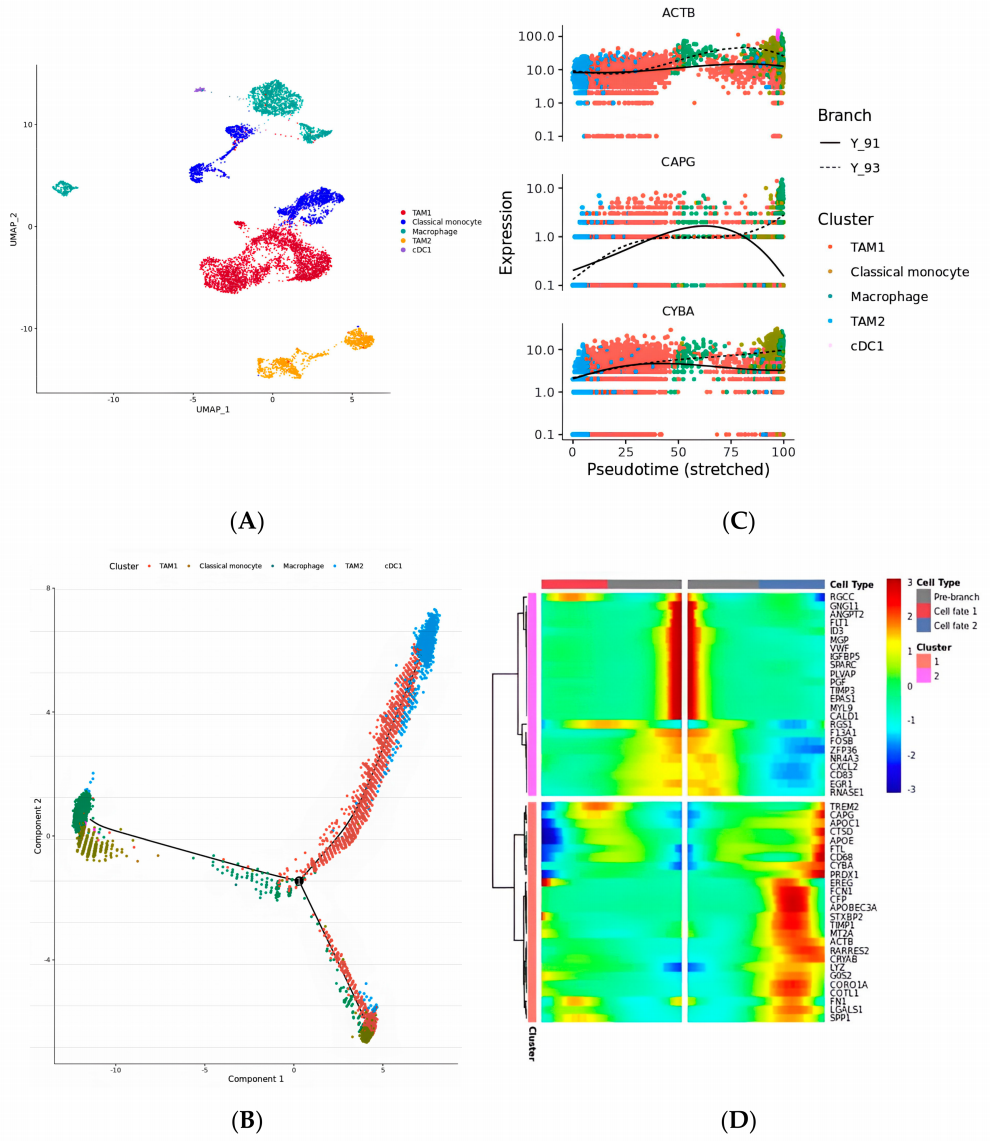
图3 UMAP、进化轨迹、表达水平和随时间变化的热图
3. ccRCC中内皮细胞的六种亚型
肿瘤微环境中的EC群体被证实可以通过调节免疫细胞的迁移、激活状态和功能来调控炎症。ccRCC是高度血管化的肿瘤,具有无序的血管结构。本研究识别了五种EC亚型和一个pro-EC亚型,重建了这些亚型的演化轨迹。在EC发展的末期,RND1和ENPP2基因高度表达,参与血管发展和重塑。特别是pro-EC和EC3在EC分化的早期阶段发挥重要作用,SPP1和CCL5基因在调控免疫、细胞生存和肿瘤进展中具有重要作用(图4)。这些发现揭示了EC亚型在ccRCC血管环境建立和病理进程中的关键角色。
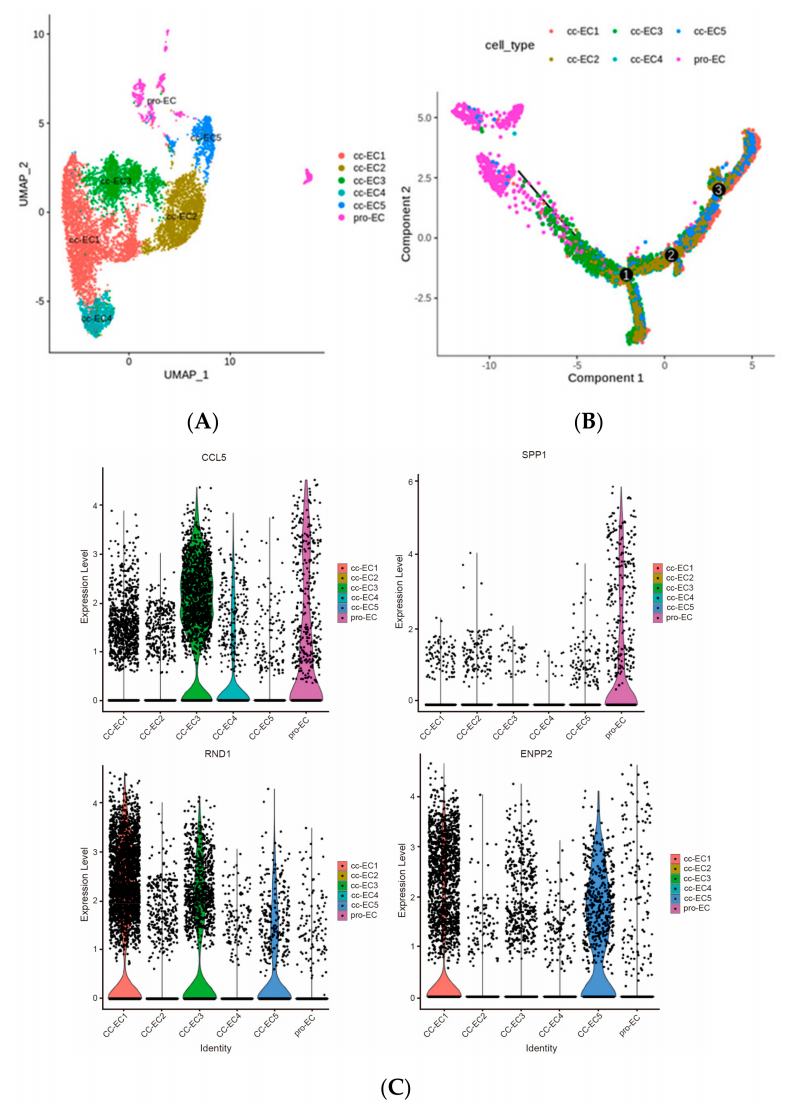
图4 EC的聚类、进化轨迹和显著表达基因的表达水平分布
4. scRNA-seq揭示了ccRCC中成纤维细胞的多样性
CAF广泛存在于肿瘤细胞和超过50%的基质细胞中,是一类能分泌多种活性因子以调节肿瘤的发生、发展和转移的激活成纤维细胞。在ccRCC中,作者鉴定出四种CAF亚型:三个不同的CAF亚群和一个成纤维细胞群。轨迹分析显示,成纤维细胞处于发展的早期阶段,而CAF-3和CAF-2细胞则分化为具有不同转录特征的两组。CAF-3亚群高度表达Lumican(LUM)和COL1A1基因,这些基因与胶原蛋白纤维组织、细胞迁移和组织修复等过程相关,而CAF-2细胞高度表达的CCL5和NKG7基因则与慢性炎症环境和免疫反应有关(图5)。这些结果揭示了不同CAF亚型在肿瘤进展和免疫调节中的关键作用,为肿瘤治疗提供了潜在的新靶点。
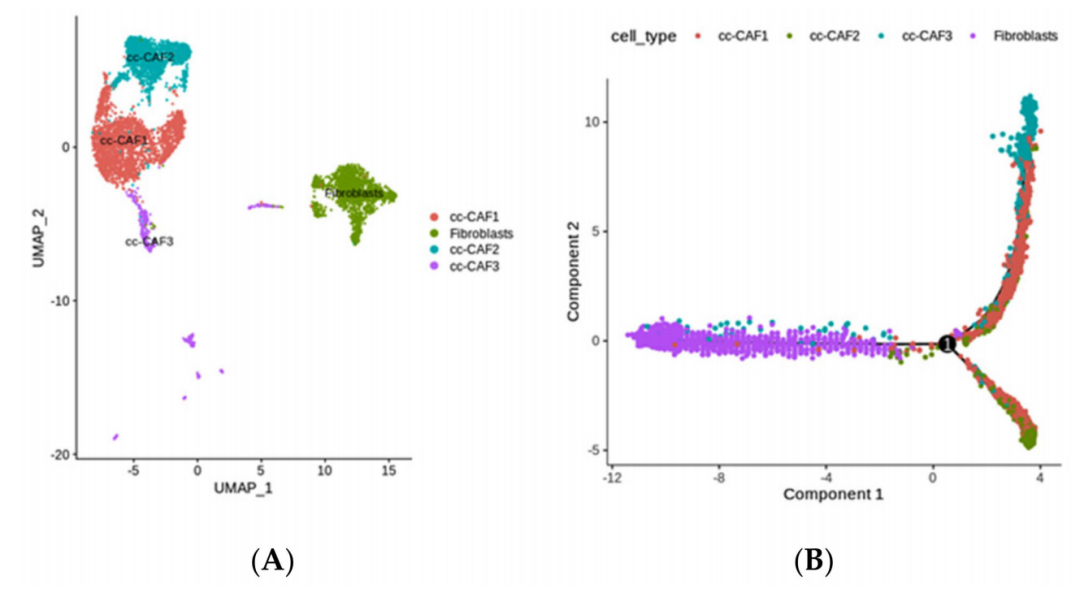
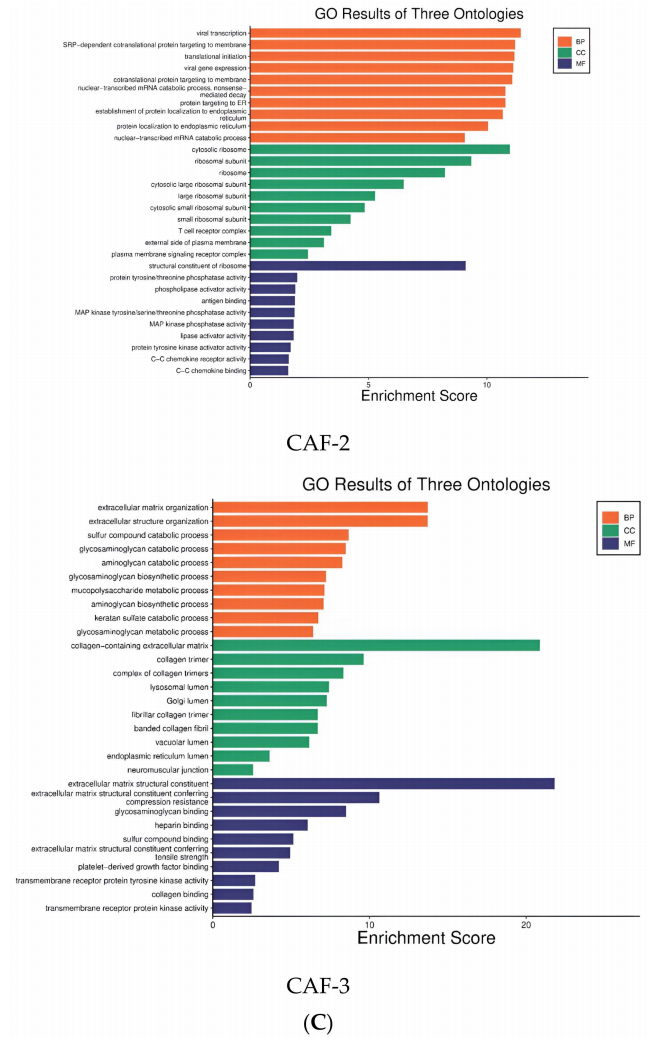
图5成纤维细胞亚簇的分析
5. ccRCC免疫微环境中T细胞特性的多样性
T细胞是具有肿瘤杀伤特性的免疫细胞,在细胞介导的主动免疫和一定程度的体液免疫中发挥重要作用。在本研究中,ccRCC细胞群中的T细胞被识别为CD8+ T细胞1、CD8+ T细胞2、CD8+ T细胞3和CD4+ T细胞。然而,在六个ccRCC样本中,除了一个来自3期肿瘤的样本外,T细胞的表达远低于其他细胞。调节性T细胞作为CD4+ T细胞的主要免疫抑制亚群,在多种实体瘤中高度浸润,与许多类型肿瘤的较差临床结果密切相关。因此,作者考虑了调节性T细胞和CD4+ T细胞的主要特征基因作为下一步选择TF的候选基因(图6)。总之,这些发现揭示了不同T细胞亚型在肿瘤免疫微环境中的作用及其对临床结果的潜在影响。
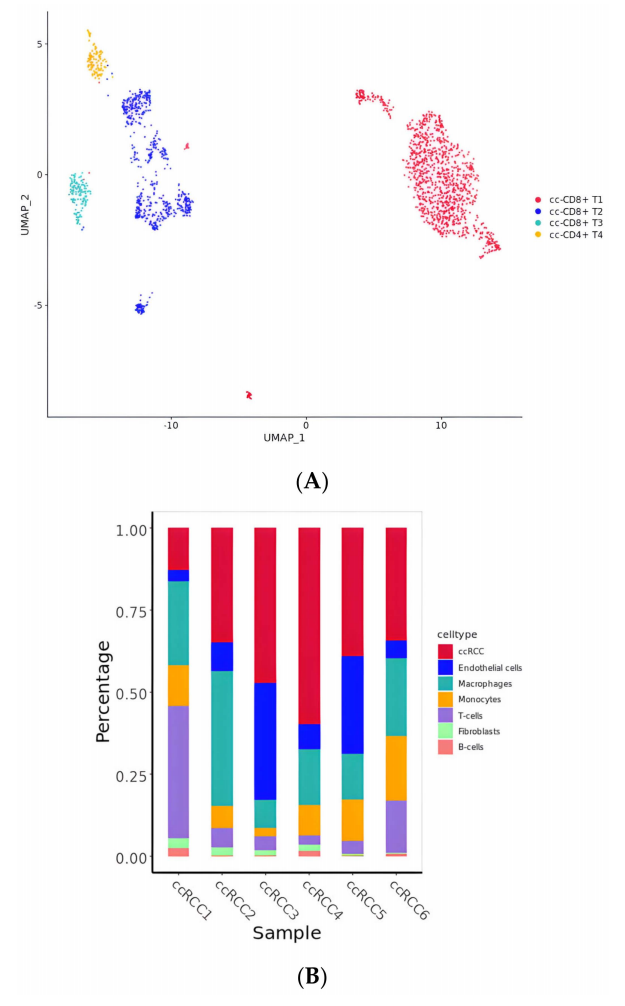
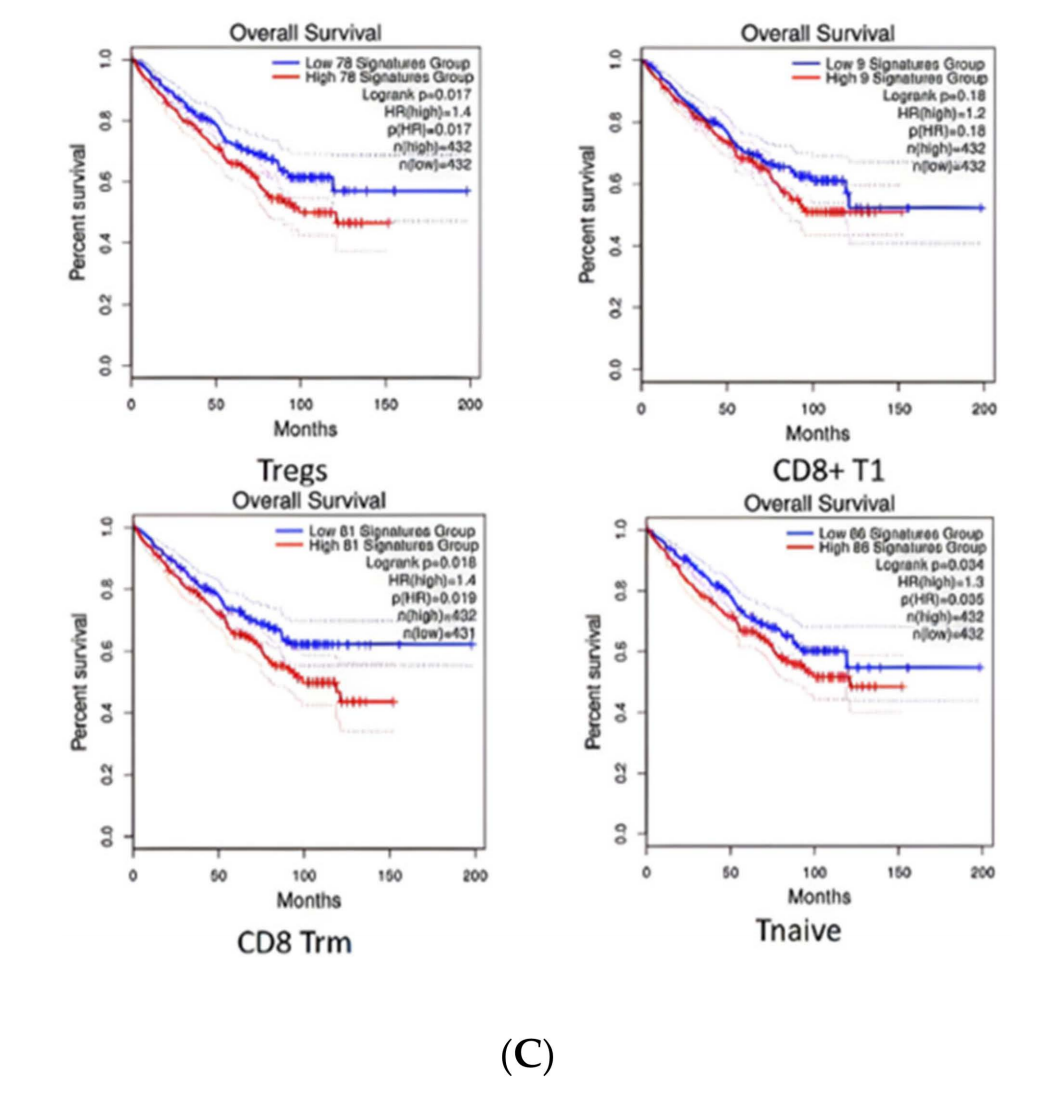
图6T细胞亚簇的分析以及TCGA标记基因表达和生存分析
6. EPAS1/HIF-2α与ccRCC高度相关
内皮PAS结构域蛋白1(EPAS1),亦称为缺氧诱导转录因子2(HIF-2α),是一种基础螺旋-环-螺旋/PAS结构域转录因子,在高度血管化的器官中高度表达。作为维持体内稳态的主要调控因子,HIF通过激活包括红细胞生成素(EPO)基因和血管内皮生长因子(VEGF)基因在内的多个基因来调节对缺氧的响应。特别是在ccRCC中,VHL基因突变失活是最早的遗传事件之一,而HIF可以被这种肿瘤抑制蛋白(VHL)泛素化,导致HIF-1α和HIF-2α的累积,并调控ccRCC的发展和炎症。此外,研究发现HIF-2α在M2型巨噬细胞极化过程中被T辅助细胞2型(Th2)细胞因子诱导,缺乏HIF-2α的骨髓系将导致肿瘤相关巨噬细胞(TAM)浸润减少和肿瘤进展缓解。HIF-2α的缺失还会增加中性粒细胞的凋亡并减少中性粒细胞炎症。总之,EPAS1/HIF-2α与ccRCC的发生和发展密切相关,是肾癌中表达最高的预后标志物之一。
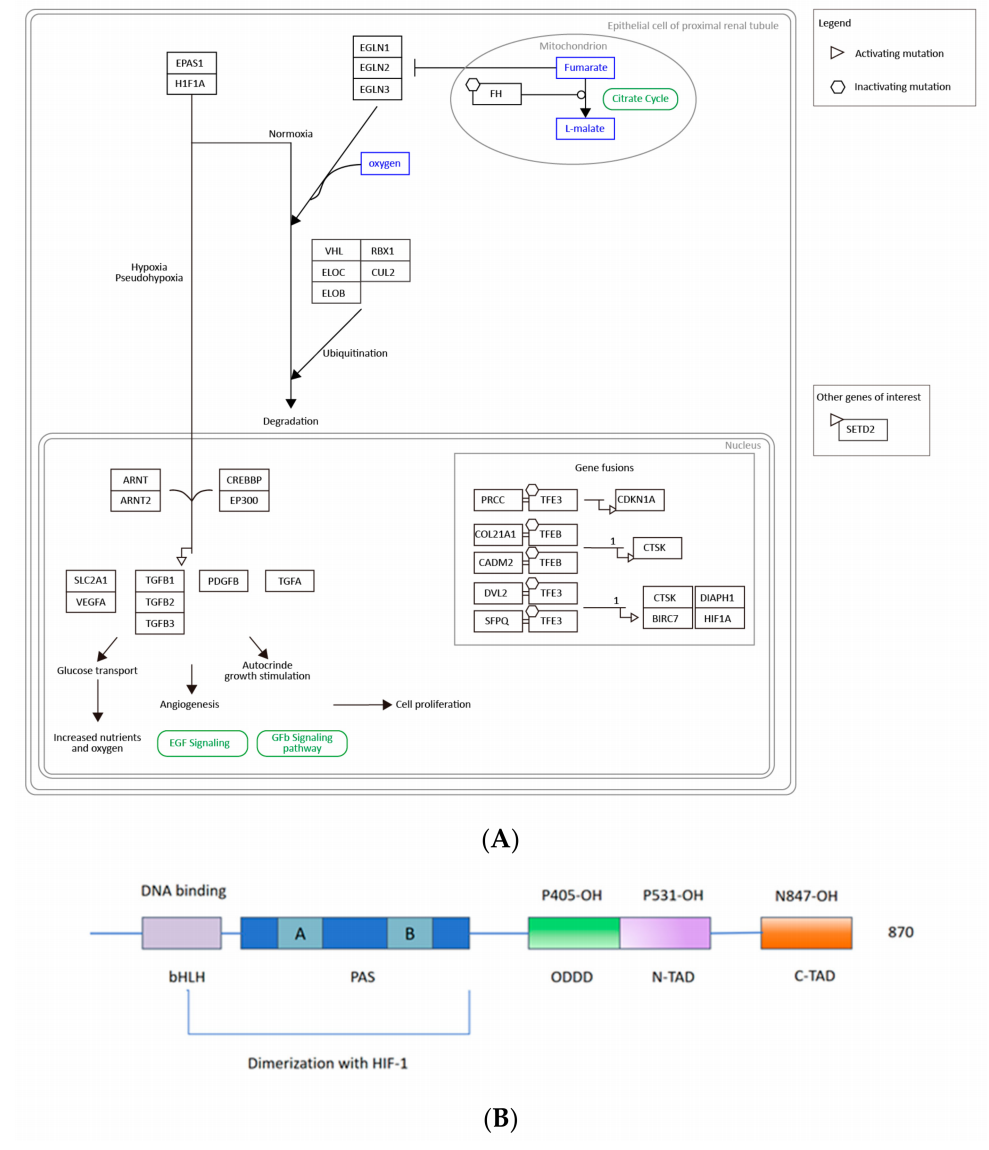
图7EPAS1的通路和蛋白的结构域
7. 鉴定出5种针对TF EPAS1的化合物
在应用深度学习算法DMPNN + XGBoost提取分子特征并预测目标蛋白“EPAS1”的分子对接分数后,筛选出了五种顶级对接分数的化合物。其中,化合物1能与EPAS1蛋白形成氢键。化合物2能抑制一氧化氮的产生、COX-2的表达及NF-kB的核转移,并与EPAS1形成氢键。化合物3和化合物4分别是两种FDA批准的药物:氟芬酸和氟达拉滨。氟芬酸是一种非甾体抗炎药,可抑制COX活性、调节离子通道并阻断氯和L型Ca2+通道,同时抑制TEAD功能及相关的细胞迁移和增殖过程。氟达拉滨是一种抗肿瘤活性的氟化嘌呤类似物,可抑制由细胞因子诱导的STAT1活化和依赖的基因转录。最后一个化合物是胞嘧啶核苷类似物,具有抑制DNA甲基转移酶的作用及潜在的抗代谢和抗肿瘤活性。这些化合物不仅具有氢键生成能力,还能产生π-π相互作用(图8)。这部分结果揭示了通过分子对接筛选出的化合物对EPAS1的潜在调控机制。
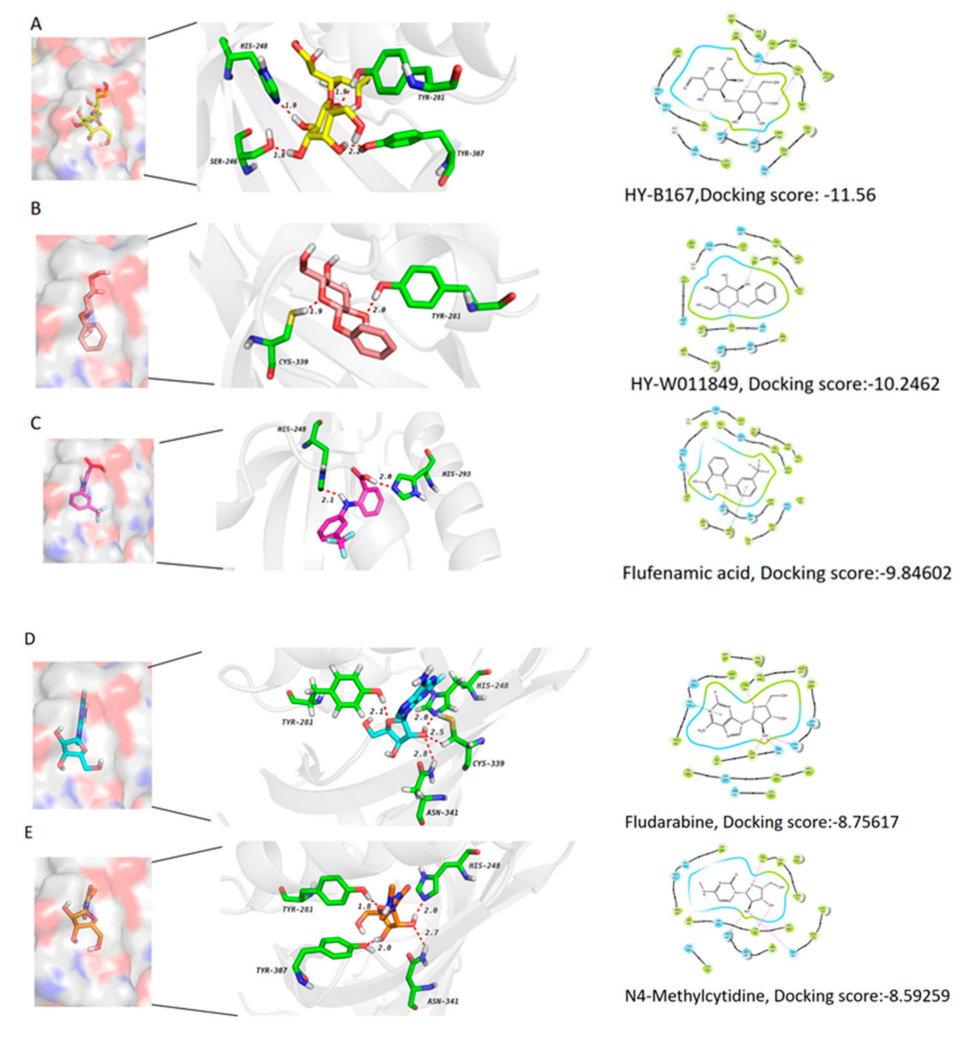
图8五种筛选出的化合物的对接模型和化学结构
为了进一步验证化合物与蛋白质结合的程度和稳定性,作者对EPAS1蛋白和两种FDA批准的药物进行了25纳秒的分子动力学模拟。通过RMSD曲线来评估蛋白质配体复合物的稳定性,整个过程中RMSD曲线的波动范围控制在3Å以内。此外,为了研究小分子与蛋白质口袋中每个残基的相互作用,作者统计了模拟过程中的相互作用。在氟芬酸复合物中,蛋白质残基HIS-248、SER-249、Ph-254、TYR-281和HIS-293对结合自由能的贡献较大。在氟达拉滨复合物中,蛋白质残基ASN_341、TYR_307和CYS_339与小分子形成稳定的氢键相互作用。此外,SER_304和TYR_281在约40%的轨迹中与小分子有氢键相互作用(图9)。这些分子动力学模拟结果证实了氟芬酸和氟达拉滨与EPAS1蛋白之间具有高度的结合稳定性和重要的相互作用。
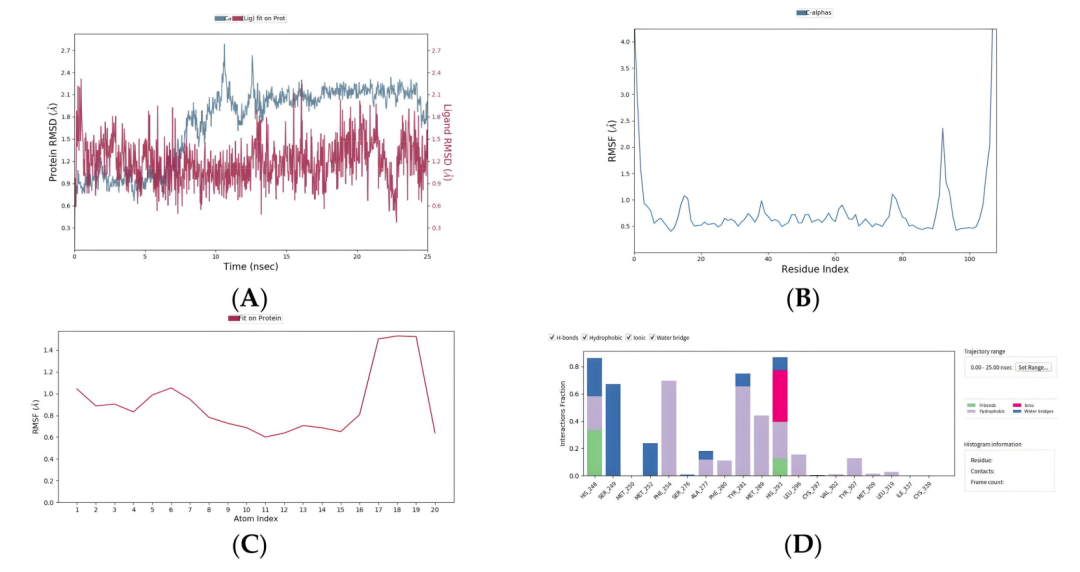
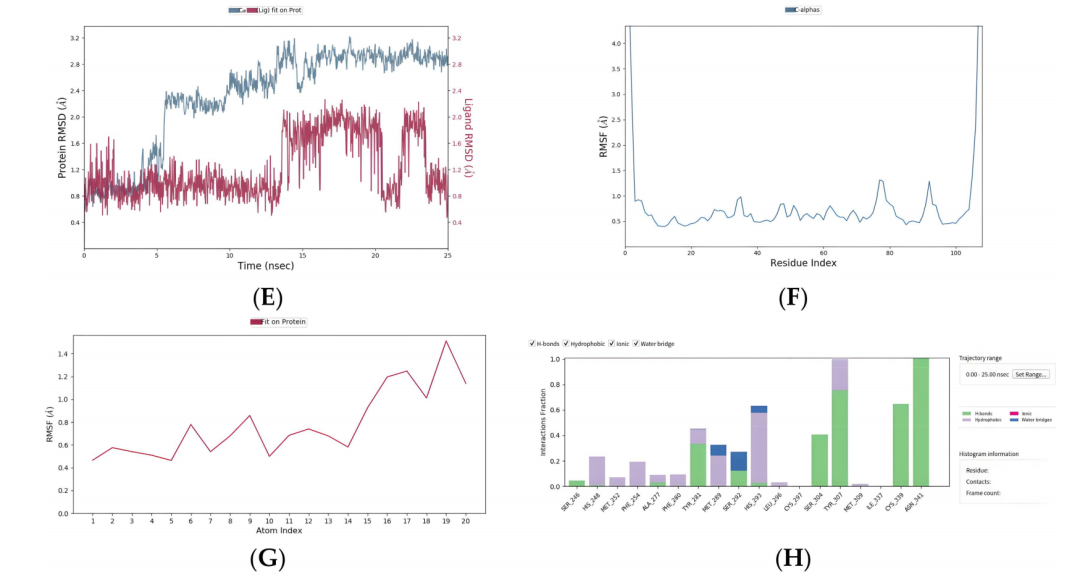
图9FDA药物氟灭灭酸和氟达拉滨的分子动力学模拟结果
文章小结
在本研究中,作者构建了一个全面的ccRCC肿瘤微环境景观,涵盖了其肿瘤相关巨噬细胞、内皮细胞、肿瘤相关成纤维细胞和T细胞的异质亚群。通过这些关键TME特征的差异性分型和综合深度学习模型的计算,识别出关键转录因子蛋白EPAS1/HIF-2α,该因子调节涉及调整缺氧机制(如血管生成或凋亡以及肿瘤生长和侵袭)的基因表达。最终,作者筛选出五种候选化合物,包括两种FDA批准的抗癌药物和一种DNA甲基转移酶抑制剂,这些都为旧药新用和开发新的ccRCC治疗药物提供了重要的理论基础。深度学习结合机器学习分析单细胞和巨噬细胞数据,非常新颖的数据挖掘思路!如果还在传统生信分析中无法很好解决问题的话,不妨就来尝试一下深度学习和机器学习的融合吧,一定能碰撞出新的火花!对于相关研究的创新思路还在困惑吗?或者在进行深度学习分析时无从下手?快向小云发起交流,一起找到新领域新思路新方法吧~

定制生信分析
云服务器租赁
热点推荐
代码合集(点击查看)
原文始发于微信公众号(云生信学生物信息学):投稿到见刊不到两个月?北科大艾冬梅团队零实验5.6分:单细胞挖掘分析+机器学习+公共数据库+药物发现,顶级思路来了!