
昨天有小伙伴私信小云,觉得公共数据库中数据集往往经过大量分析,难以创新。有一定道理,但只要用对思路,综合利用公共数据库,一样可以轻松发文章!今天小云就给大家分享一篇。本文综合利用了两个数据库,采用多种生物信息学方法,分析了糖尿病足溃疡和细胞衰老相关的基因和通路。这篇论文是如何做的呢?小云带小伙伴们一起揭秘一下吧~
1,公共数据库的综合利用:文章通过整合基因表达数据集和细胞衰老数据库,研究识别了一组差异表达基因(DEGs),并进一步对这些基因进行功能富集和通路分析,揭示了与糖尿病足溃疡发病机制相关的生物学过程和信号通路;
2,综合利用多种生物信息方法分析:文章文章利用蛋白质相互作用网络构建和模块分析揭示了核心的差异基因,并通过通路富集分析和mRNA调控网络分析深入探讨了这些基因在细胞衰老和糖尿病足溃疡中的功能特征。找到了糖尿病足溃疡和细胞衰老相关的基因和通路。想发SCI的小伙伴们可以借鉴一下本文的研究思路~~(ps:感兴趣的同学欢迎来扫码私聊噢!小云作为深耕生信领域多年的老生信人,熟悉各种生信分析思路和相关数据库哦,总有适合你的哦!)。

云服务器租赁
(加微信备注99领取试用)

题目:通过综合转录组分析鉴定与糖尿病足溃疡细胞衰老相关的中心基因和通路
杂志:Journal of Cellular and Molecular Medicine
影响因子:IF=5.295
发表时间:2023年4月
研究背景
随着全球糖尿病患者数量的增加,糖尿病足并发症的发生也在增加。据统计,大约80%的下肢截肢患者在手术前都会患有糖尿病足。糖尿病足并发症的病理机制涉及内源性因素,如神经、血管、免疫、代谢和慢性感染的改变,以及过度炎症和表皮或真皮细胞对修复信号的不响应。近年来,越来越多的证据表明细胞衰老(CS)与糖尿病足溃疡的发生和进展密切相关。最近的研究表明,高胰岛素血症导致成熟脂肪细胞中早期衰老的转录组和分泌物质轮廓,同时导致神经元中的胰岛素抵抗,从而产生衰老样表型。因此研究细胞衰老与与糖尿病足溃疡的发生关系,能够在基因水平上提供对糖尿病足溃疡发展的新见解,能够为糖尿病足溃疡的新治疗策略。
数据来源
|
数据集/队列 |
数据库 |
数据类型 |
详细信息 |
|
GSE80178 |
GEO |
基因表达数据 |
包含了糖尿病足溃疡(DFU)和糖尿病足皮肤(DFS)样本 |
研究利用了CellAge数据库。CellAge数据库是一个专注于细胞衰老相关基因的资源库,其中收集了各种人类细胞类型的基因操纵实验得到的相关基因信息。通过CellAge数据库,研究人员获取了与细胞衰老相关的279个基因。
研究思路
研究团队通过整合生物信息学分析,探索糖尿病足溃疡(DFU)中细胞衰老(CS)相关的基因和通路。首先,收集并分析公共数据库中DFU和正常皮肤样本的基因表达数据,识别出与CS相关的差异表达基因(DEGs)。然后,构建这些DEGs的蛋白质相互作用(PPI)网络,并识别出核心的CS-DEGs。接下来,通过功能富集分析和路径通路分析揭示这些基因在生物学过程和信号通路中的功能和作用机制。最后,结合miRNA-TF-mRNA调控网络分析,探究这些CS-DEGs的调控网络及其在DFU中的调控作用。这一研究思路旨在揭示CS与DFU发病机制之间的关系。
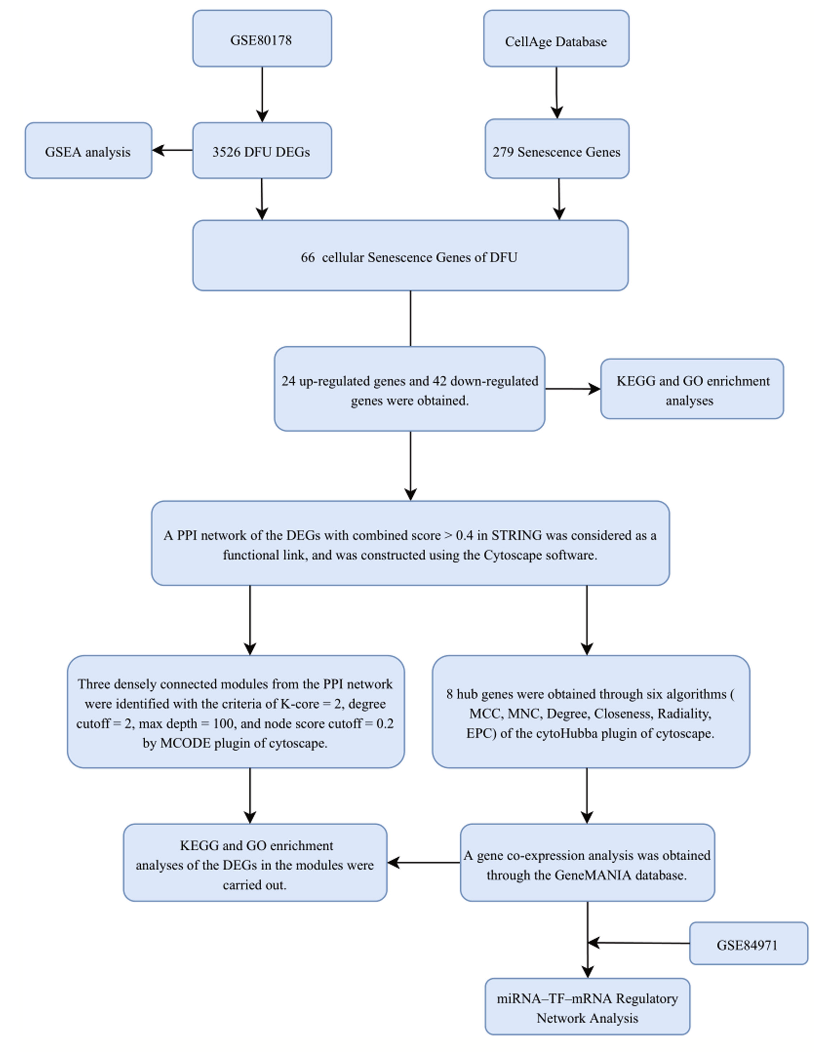
研究结果
1.差异表达基因(DEGs)的识别
研究人员使用了R语言中的limma包进行了差异表达基因(DEGs)的识别。研究人员首先进行了数据的归一化处理,然后对GSE80178数据集进行分析。在归一化后的数据中,共检测到了3526个差异表达基因(DEGs),这些基因在糖尿病足溃疡(DFU)和非糖尿病足皮肤(DFS)样本中显示出显著的表达差异。为了进一步探索与细胞衰老(CS)相关的差异基因,研究人员将GSE80178中的DEGs与CellAge数据库中的CS相关基因进行了比对(图1)。通过Venn图的分析,确定了66个同时存在于GSE80178和CellAge数据库中的重叠的CS-DEGs。这些CS-DEGs的表达热图展示了它们在DFU和DFS样本之间的表达模式差异,能够有效区分这两种样本。表S1中提供了这些重叠的CS-DEGs的详细信息,包括基因名、表达模式等。这些结果表明,这些CS-DEGs可能在糖尿病足溃疡的发生和发展中发挥着重要的作用。
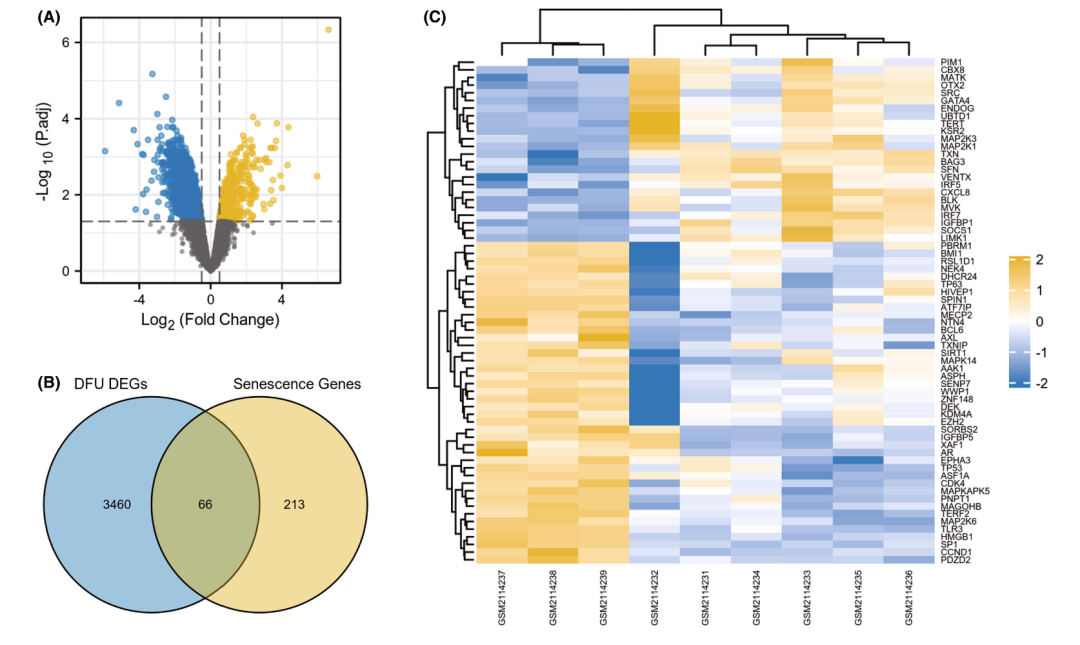
图1 GSE80178数据集的火山图与差异性分析
2.功能性分析
研究人员使用了基因集富集分析(GSEA)来识别与糖尿病足溃疡(DFU)相关的生物学过程和通路。GSEA分析显示,前五个富集项为RNA代谢、细胞周期、有丝分裂细胞周期、含有帽的内含子预mRNA的加工以及细胞器的生成和维持(图2A)。这些结果提示了在DFU的发生和发展过程中,RNA代谢、细胞周期调控和细胞器功能维持等生物学过程可能起着重要作用。为了进一步分析涉及细胞衰老相关差异表达基因(CS-DEGs)的生物功能和通路,研究人员进行了GO(Gene Ontology)和KEGG(Kyoto Encyclopedia of Genes and Genomes)通路富集分析。GO分析结果显示,CS-DEGs在生物过程(BP)方面显著富集于细胞衰老、细胞老化和衰老等过程。在细胞组分(CC)方面,这些差异基因主要与核染色质、PcG蛋白复合物和转录抑制复合物相关,暗示了这些基因在细胞核内的功能活动。在分子功能(MF)方面,CS-DEGs主要与MAP激酶激酶活性、蛋白酪氨酸激酶活性和蛋白丝氨酸/苏氨酸/酪氨酸激酶活性相关,表明它们可能参与信号传导和调节细胞的生物学功能(图3)。KEGG通路分析显示,这些CS-DEGs主要参与细胞衰老、卡波西肉瘤相关的单纯疱疹病毒感染以及Toll样受体信号通路等通路。这些结果为进一步研究糖尿病足溃疡的发病机制和治疗提供了重要线索,提示细胞衰老可能在DFU的发生和发展中起着关键作用。
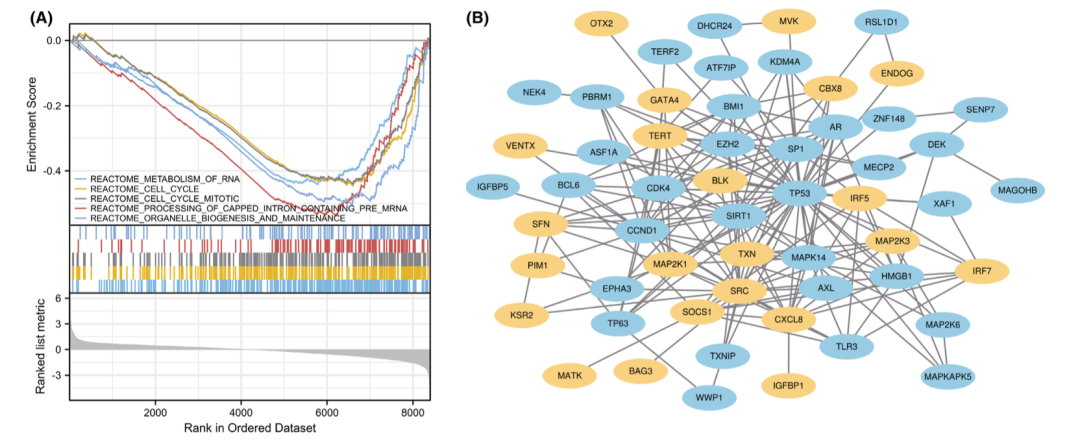
图2 数据富集项和蛋白质相互作用网络可视化分析
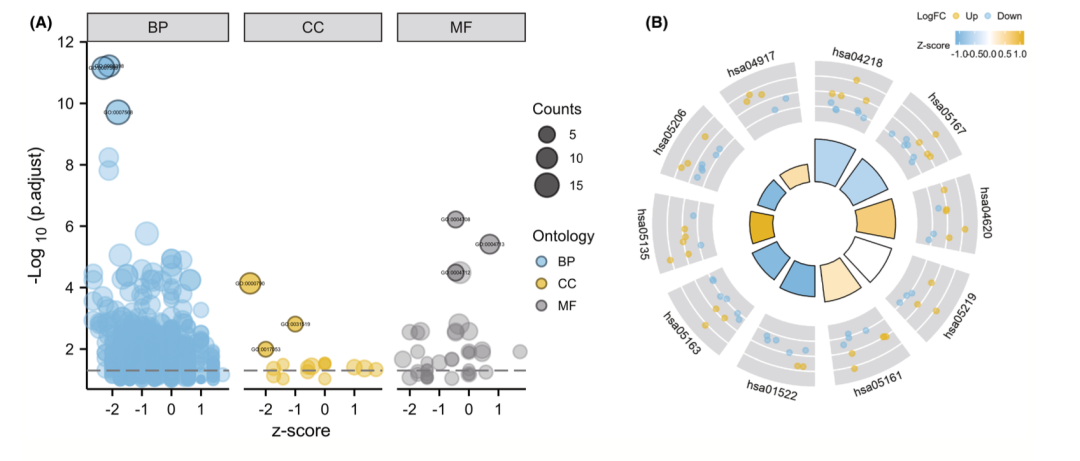
图3细胞衰老差异表达基因富集结果分析
3. 蛋白质相互作用(PPI)网络分析
研究团队利用通过Cytoscape构建了与细胞衰老相关的差异表达基因(CS-DEGs)的蛋白质相互作用(PPI)网络,并进行了模块分析。首先研究人员利用Cytoscape构建了CS-DEGs的PPI网络。网络显示了CS-DEGs之间的复杂相互作用模式,揭示了它们在糖尿病足溃疡(DFU)发生和发展中可能扮演的角色。接着,研究人员利用Cytoscape的MCODE插件进行了模块分析,发现了三个密切相关的基因模块,包括18个CS-DEGs和41个相互作用对(图4A–C)。这些模块可能代表了在DFU病理生理过程中功能紧密相关的基因集合。GO(Gene Ontology)分析显示这些基因参与了多个生物学过程,包括节律过程、胰岛素受体信号通路的调节、转移酶复合物、含磷基团的转移、PcG蛋白复合物、特异性启动子染色质结合以及蛋白C-末端结合等过程(见图5D)。这些结果提示了这些CS-DEGs可能通过多种途径影响DFU的发生和发展。
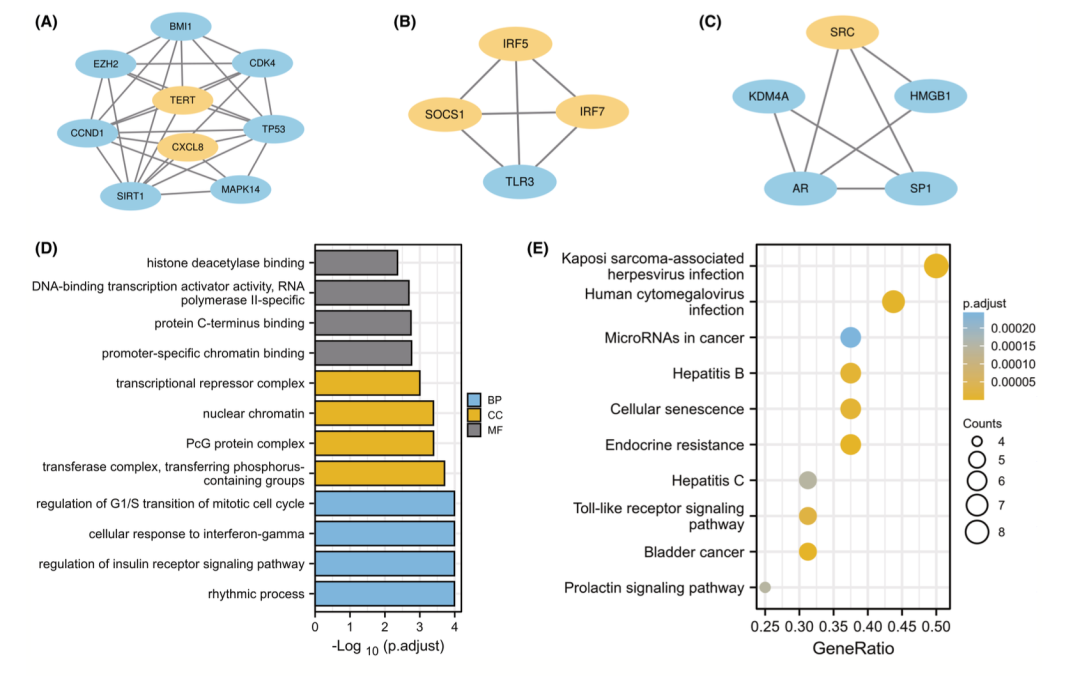
图4 显著基因聚类模块及其富集分析
4.核心CS-DEGs的选择和分析
研究人员计算了前10个枢纽基因,发现了8个重叠的枢纽基因,分别是TP53、SRC、SIRT1、CCND1、EZH2、CXCL8、AR和CDK4(图5A)。接着,研究人员利用GeneMANIA数据库进行了这些基因的共表达网络和相关功能的分析。这些基因展示了一个复杂的蛋白质相互作用网络(PPI网络),其中物理相互作用占44.88%,预测相互作用占19.39%,共定位占12.51%,通路占11.63%,遗传相互作用占6.28%,共表达占5.32%(图5B)。这些基因与调节细胞周期G1/S期转换、有丝分裂细胞周期G1/S转换以及有丝分裂细胞周期G1/S转换相关。GO分析显示,这些基因与对酮的响应、细胞对酮的反应、核染色质、ESC/E(Z)复合物、RNA聚合酶II基础转录因子(TF)结合和转录共抑制活性相关。KEGG通路分析发现,这些基因主要与膀胱癌、细胞衰老以及与卡波西肉瘤相关的单纯疱疹病毒感染相关。
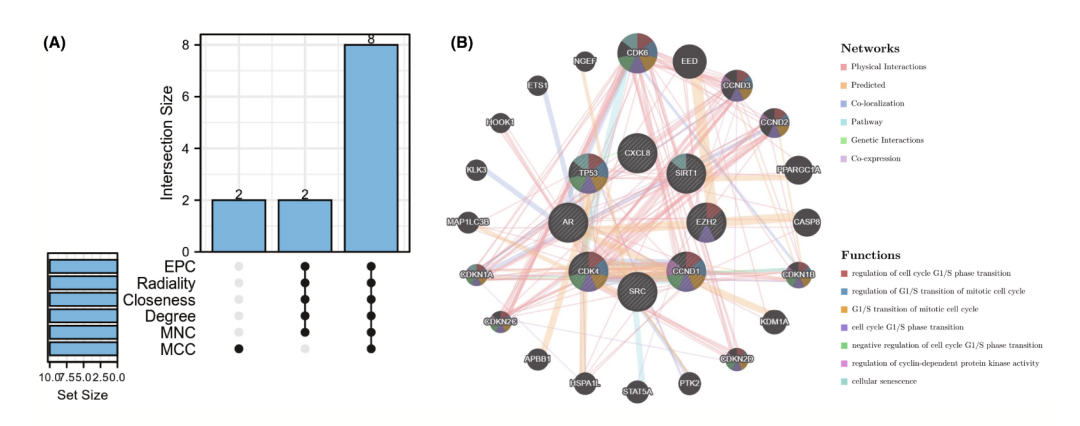
图5 细胞衰老差异表达基因及核心基因的关联分析
5. miRNA-TF-mRNA调控网络分析
研究团队进行了miRNA-TF-mRNA调控网络的分析。利用TRRUST数据库研究人员发现了3个CS-DEGs充当转录因子,以调控其他基因的表达。这些CS-DEGs包括TP53(调控EZH2、TP53、CDK4、CCND1)、SIRT1(调控CCND1、TP53、EZH2)和EZH2(调控CCND1、TP53)。研究人员使用GSE84971数据集获得了31个差异表达的miRNA。然后,我们利用miRWalk数据库分别从TargetScan、miRDB和miRTarBase预测了这8个基因的上游miRNA,预测得到的上游miRNA分别为127、1070和928。经过Venn图的交集处理,获得了3个重叠的miRNA,包括hsa-miR-93-5p(作用于CCND1)、hsa-miR-15a-5p(作用于CCND1)和hsa-miR-132-3p(作用于SIRT1)。最后,研究人员利用Cytoscape构建了miRNA-TF-mRNA调控网络。SIRT1作为转录因子受到上游hsa-miR-132-3p的调控,并同时调控下游的CS-DEGs的表达。
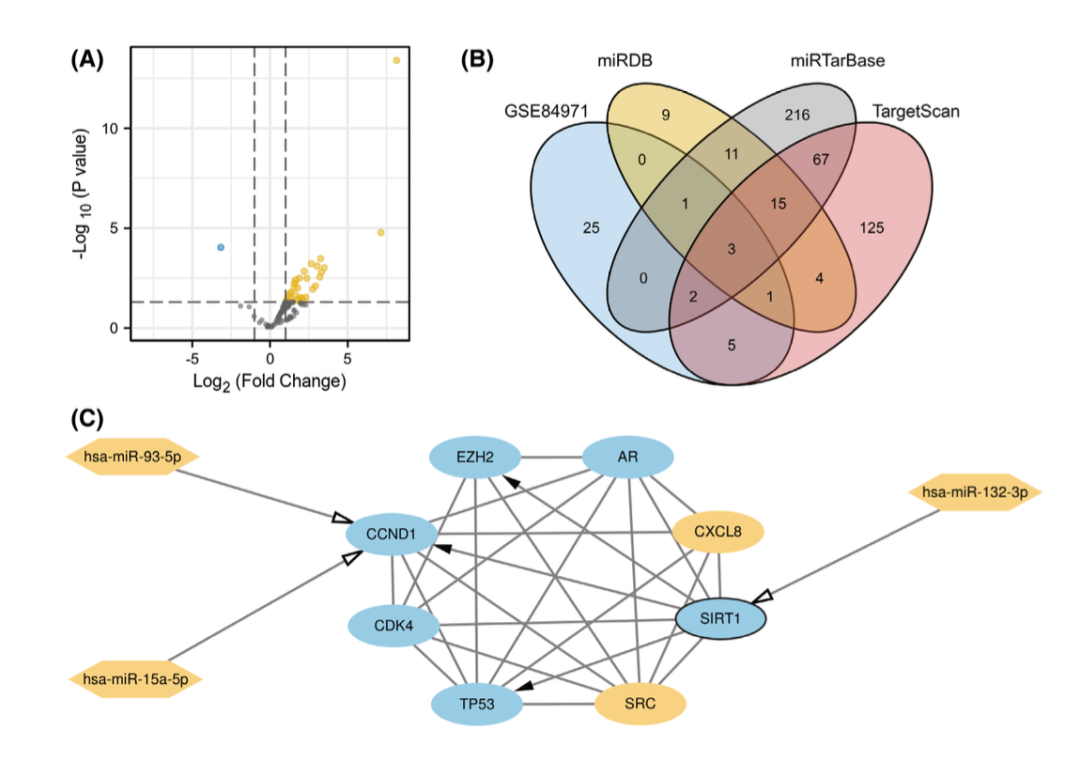
图6 miRNA-TF-mRNA调控网络分析
文章小结
这篇文章这篇文章深度结合了两个公开数据库,发现了糖尿病足溃疡和细胞衰老相关的基因和通路。并通过通路富集分析和mRNA调控网络分析深入探讨了这些基因在细胞衰老和糖尿病足溃疡中的功能特征,揭示了与糖尿病足溃疡发病机制相关的生物学过程和信号通路。看到这不知道小伙伴们有没有想法?把握住生信分析思路流程,文章快快发起来吧!感兴趣的话,有需要的小伙伴可以扫码来找小云做设计哟~

定制生信分析
云服务器租赁
热点推荐
代码合集(点击查看)
原文始发于微信公众号(云生信学生物信息学):天呐!公共数据库还能这么用?成都医学院曾明强团队1区新作!GEO数据库+CellAge数据库+综合转录组分析,快来学呀!