GSEA进行富集分析(图形界面)
 GSEA,即Gene Set Enrichment Analysis (基因集富集分析)用来评估一个预先定义的基因集的基因在与表型相关度排序的基因表中的分布趋势,从而判断其对表型的贡献。
GSEA,即Gene Set Enrichment Analysis (基因集富集分析)用来评估一个预先定义的基因集的基因在与表型相关度排序的基因表中的分布趋势,从而判断其对表型的贡献。
GSEA软件有java和R语言两个版本,其中java版具有图形界面,操作更直观,本次小云就介绍一下java版的GSEA
软件下载
软件下载网址如下
http://www.gsea-msigdb.org/gsea/downloads.jsp
Mac、windows、linux系统均支持
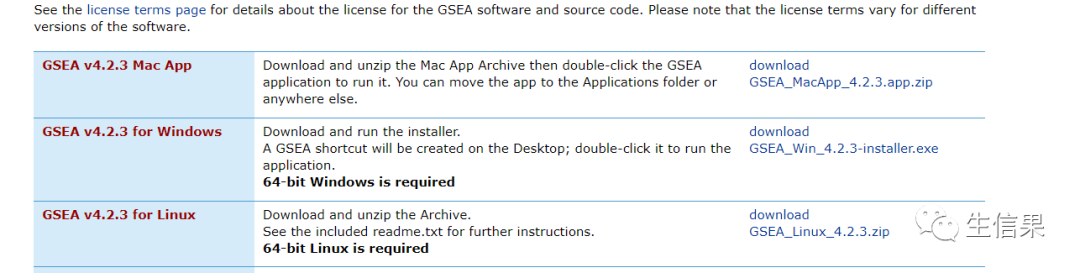
下载后按照默认设置安装即可
需提前配置java环境
输入文件准备
输入文件有三个,基因集文件、表达谱文件、表型文件
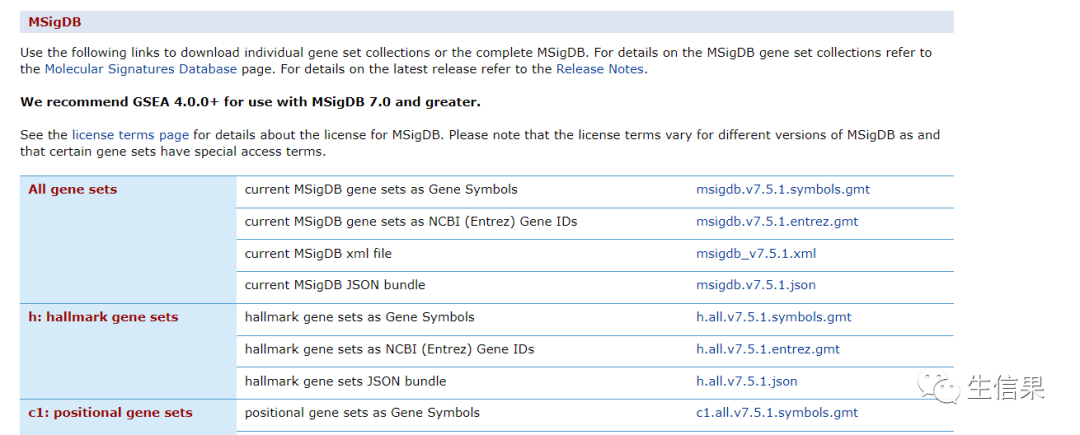
1、基因集文件下载
http://www.gsea-msigdb.org/gsea/downloads.jsp
如上网址下拉,会看到MsigDB数据库,我们依照研究目的选择合适的基因集进行下载
2、表达谱文件
我们自己准备,行名基因,列名样本号即可

3、表型文件
后缀名为CLS,分隔符为换挡符/t 。我们建个TXT文件,改后缀名即可
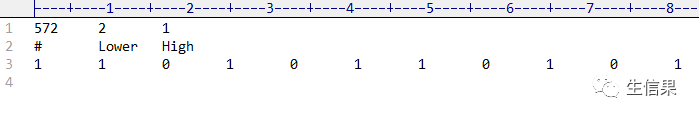
第一行为 样本数 表型分类数 固定数值1
第二行是具体的表型分类,这里是两种,对应0的Lower,对应1的High
第三行是每个样本对应的表型
三软件运行
点击 Load data进入加载数据界面
将输入数据直接拖入输入框

点击Load these..
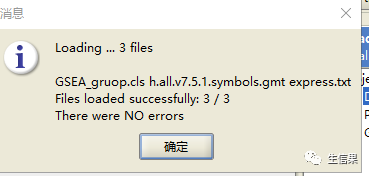
载入成功
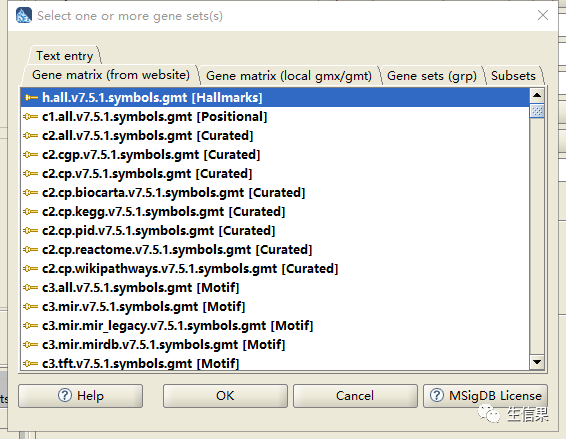
选择基因集
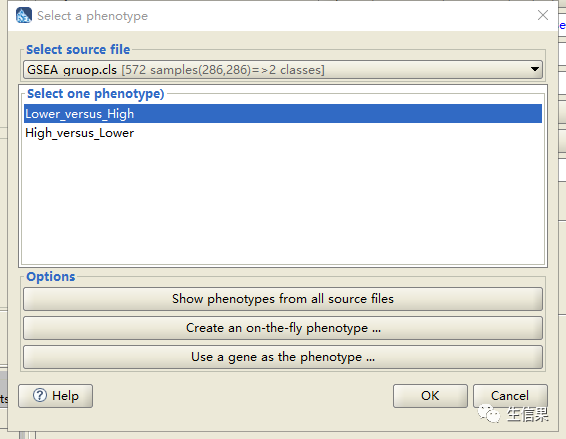
选择分组
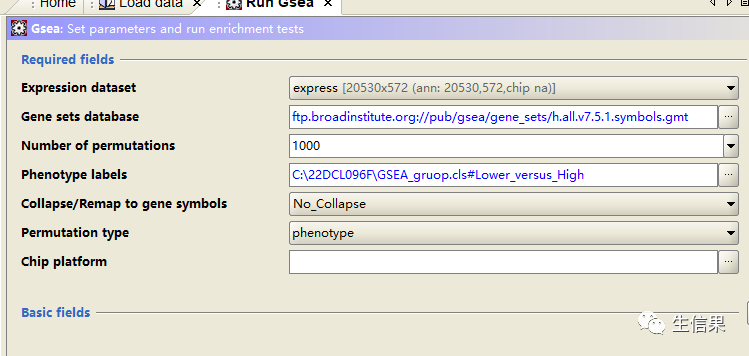
设置完成
点击Run运行
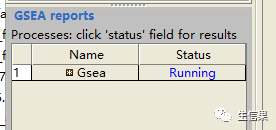
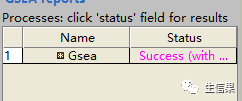
运行成功
进入结果文件夹
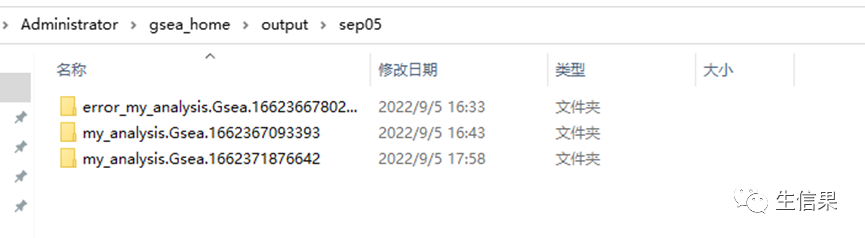
默认文件夹在如上路径
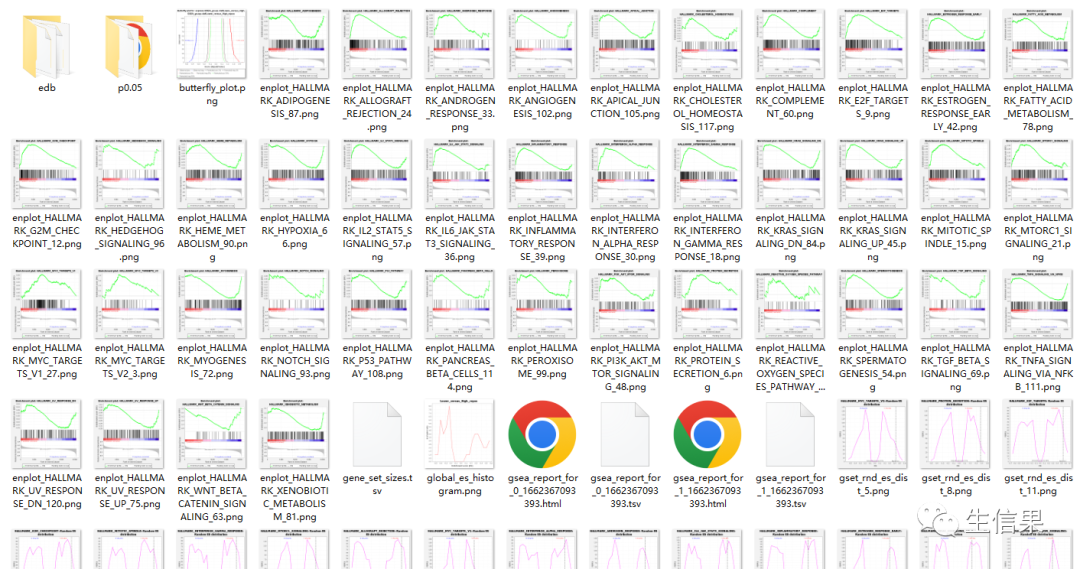
查看分析结果
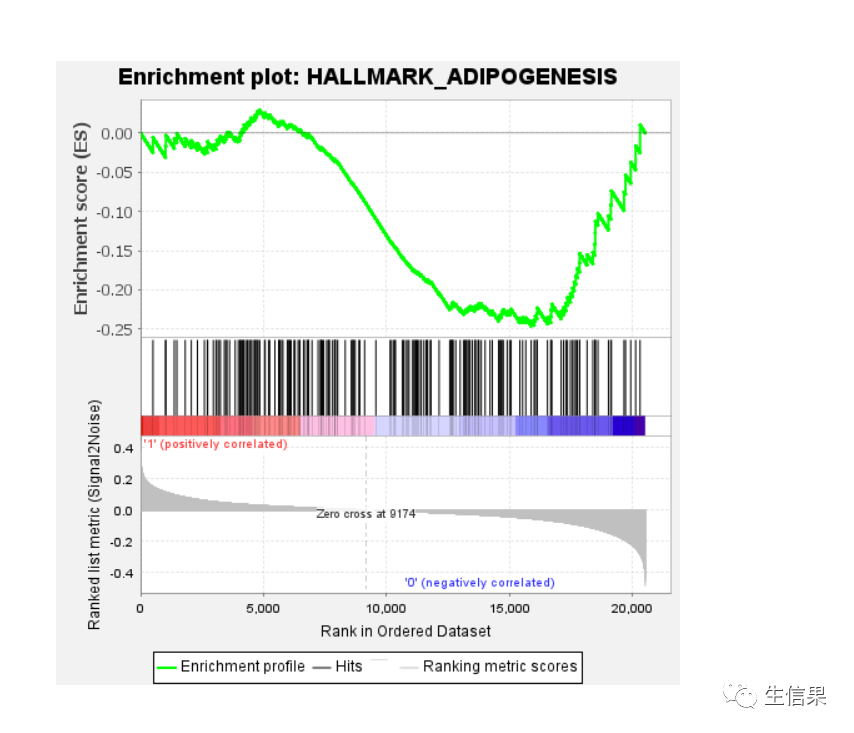
至此结束
推荐阅读