用MEGA构建进化树,不会还有人不会吧!

通过进化树我们可以揭示生物进化历史、推断分类学关系、预测生物学特征等,在生物学中很常用,今天小果来分享一下用MEGA构建进化树的方法。
构建进化树的过程可以分为三个主要步骤:收集序列数据、进行多序列比对、利用比对结果建立进化树。以下是基于MEGA11.0.13软件的详细步骤:
1.收集序列数据
本文以SOCS1基因为例,通过NCBIhttps://www.ncbi.nlm.nih.gov/获取尼罗罗非鱼(Oreochromis niloticus)、斑点雀鳝(Lepisosteus oculatus)、青鳉(Oryzias latipes)、红鳍东方鲀(Takifugu rubripes)、斑马鱼(Danio rerio)、小鼠(Mus musculus)和人(Homo sapiens)的SOCS1基因fasta序列来进行进化树的构建。
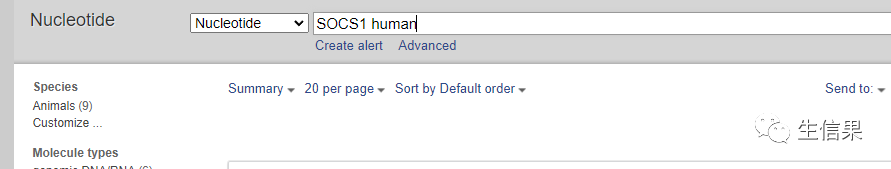
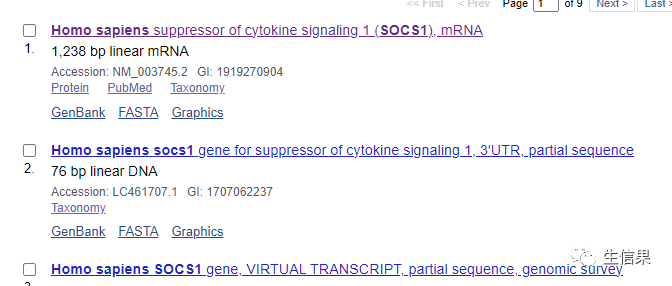

点击fasta加载出序列
此时可以从>开始直接选中所有序列复制粘贴到文本文档中,其他物种的序列也同样操作,粘贴到同一个文本文档,保存后然后修改文档后缀为.fasta就可以了。
另一种方式可以点击右上方的send to,直接从网页下载fasta格式的文件,最后将下载的序列放到同一个文件里就可以了。
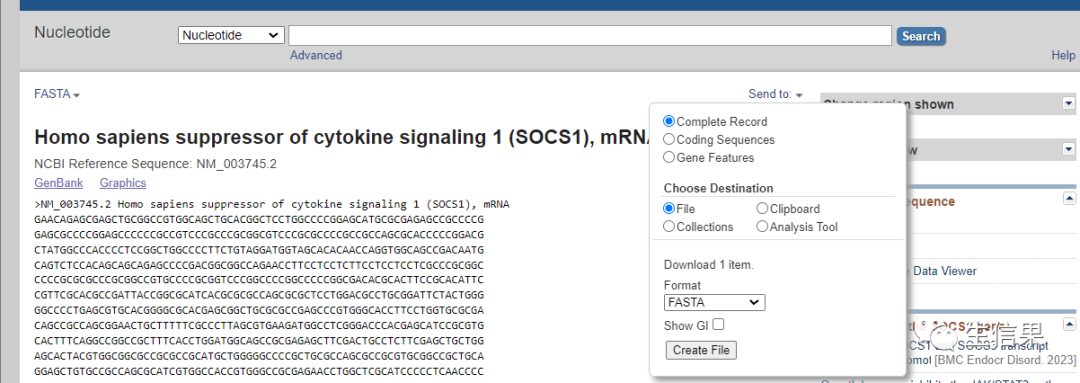
获取所需序列后可以直接在文件中修改序列的名称,不要删除>,也可以在树的构建完成后在MEGA中修改,如下,小果改为物种名加基因名的形式。
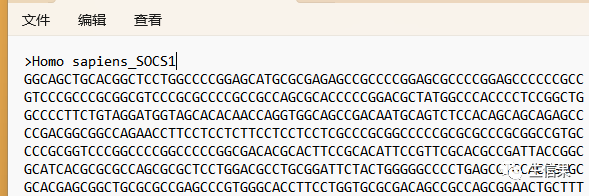
2.多序列比对
打开MEGA➡Open A File/Session➡Align

选择Align后会弹出新的窗口,我们比对的序列就加载进来了
接下来进行序列比对
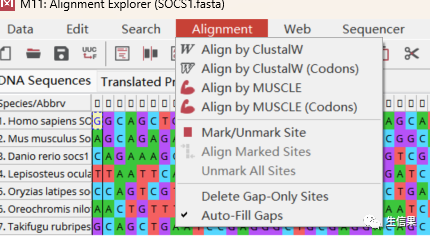
看到这么多选择不要慌,如果比对的序列比较少,直接选择第一项就可以了,如果序列很多可以用MUSCL,比对速度比较快。
这里小果选择Align by ClustalW
弹出对话框选择select all即可或者也可以手动选择要比对的序列
选择OK默认即可

等一会后序列就比对完了,将结果保存下来。
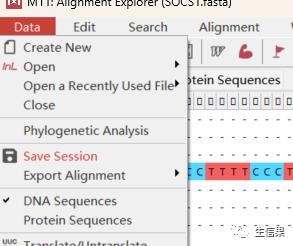
内容有限,未完待续,小果下期继续分享~
如果有绘制进化树的需求,小果强烈安利本公司的云生信平台,打开链接,在云生信·迎新春一栏找到“进化树”,动动手指直接上传自己的数据就出图啦!下图是用平台示例数据做的图哟~
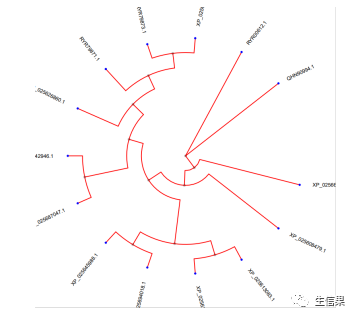
怎么样是不是很简单呢!快来注册账号吧~
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

