两分钟完成从数据下载到免疫浸润ssGSEA分析

今天小果分享一下免疫浸润ssGESA分析,从数据下载到分析,代码如下:
安装所需的R包
install.packages(“ggplot2”)Install.packages(“tidyverse”)install.packages(“GSVA”)install.packages(“pheatmap”)
导入所需的R包
library(ggplot2)library(tidyverse)library(GSVA)library(pheatmap)
数据下载
在Xena数据库下载表达矩阵和ID对应表格#表达矩阵下载wget https://gdc-hub.s3.us-east-1.amazonaws.com/download/TCGA-KIRC.htseq_fpkm.tsv.gz#基因ID转化列表wget https://gdc-hub.s3.us-east-1.amazonaws.com/download/gencode.v22.annotation.gene.probeMap
代码展示
#基因ID转换expr<-read.table("TCGA-KIRC.tsv",header=T,sep="t",row.names=1)geneann<-read.table("gencode.v22.annotation.gene.probeMap",header=T,sep="t",row.names=1)expr$gsym <- geneann[rownames(expr),]$geneexpr<-distinct(expr,gsym,.keep_all=T)row.names(expr)<-expr$gsymexpr<-slect(expr,-gsym)write.table(expr,"TCGA-KIRC-expr.txt",col.names=T,row.names=T,sep="t")#ssGSEA分析exp<-read.table("TCGA-KIRC-expr.txt",header=T,row.names=1,sep="t")geneset<-read.table("genelist.txt",header=T,sep="t")构建免疫细胞Marker基因集:geneset2 <- split(geneset$Metagene,geneset$Cell.type)#ssGSEA:res <- gsva(as.matrix(exp),geneset2,method = "ssgsea",kcdf = "Gaussian",mx.diff = F,verbose = F)#给热图添加样本注释信息:group_list <- ifelse(str_sub(colnames(res),14,15) < 10,"tumor","normal")annotation <- data.frame(group_list)rownames(annotation) <- colnames(res)pheatmap(res,show_colnames = F,annotation_col = annotation,fontsize = 10,filename=”ssGSEA.pdf”)
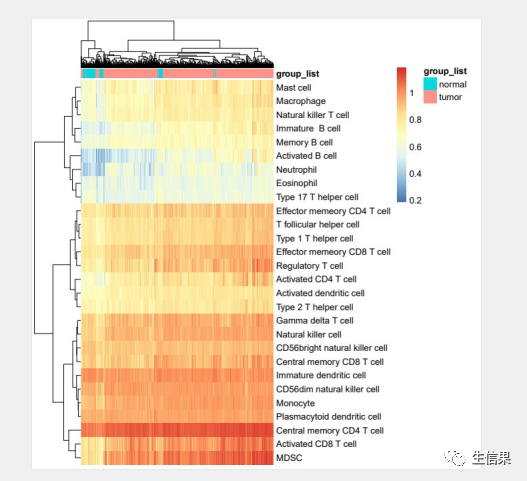
今天小果的分享就到这里,有需要的可以借鉴学习。
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)
