有我在,别害怕!利用DNAMAN进行核酸比对的教程来啦~

DNAman是一款专业的分子生物学软件,可以用于DNA序列分析、比对、修剪、重组、克隆等操作。
首先导入比对序列,小果还是用上次做进化树用到的序列:通过NCBIhttps://www.ncbi.nlm.nih.gov/获取尼罗罗非鱼(Oreochromis niloticus)、斑点雀鳝(Lepisosteus oculatus)、青鳉(Oryzias latipes)、红鳍东方鲀(Takifugu rubripes)、斑马鱼(Danio rerio)、小鼠(Mus musculus)和人(Homo sapiens)的SOCS1基因fasta序列。
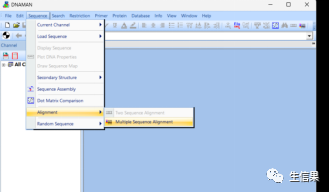
打开DNAman9软件,选择“Sequence”菜单下的“Align sequences”选项。
在“Align sequences”窗口中,点击“Mutiple Sequence Alignment”按钮
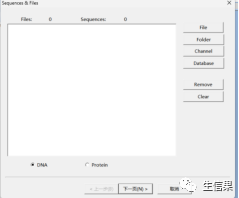
注意最下方导入的序列是DNA序列还是蛋白序列,勾选相应的即可,点击右侧File导入fasta序列,下一步
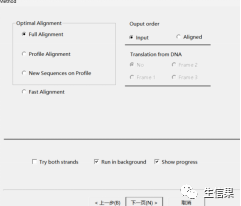
继续下一页,在这个界面一般默认即可
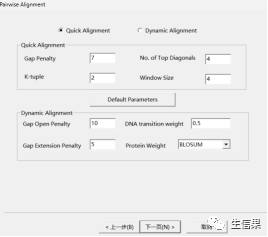
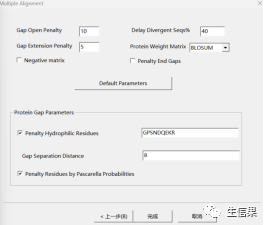
* K-tuple的大小一般设置在2-6之间,具体取决于序列的长度和变异性。通常情况下,较短的K-tuple能够更好地捕捉序列中的局部相似性,但可能会在比对过程中产生误配。而较长的K-tuple则能够更好地区分序列的差异,但可能会错过一些相似性区域。
*Gap penalty和gap extension penalty:这两个参数控制空位的罚分,即空位出现的惩罚程度。一般情况下,gap penalty的值应该比gap extension penalty高,因为在实际生物序列中,空位出现的概率较低。建议初始值分别设置为10和0。
* delay divergent seqs%”是多序列比对中的一个参数,其作用是控制在比对过程中跳过高度分歧的序列,以减少这些序列对比对结果的影响。
完成以后等待结果,在这个窗口中,可以查看序列的比对结果,包括比对得分、相似性矩阵、序列的同源性等信息。
可以使用“File”菜单下的“Save as”选项将比对结果保存为文本文件,也可以选中结果后使用“Edit”菜单下的“Copy”选项将比对结果复制到剪贴板中,以便于在其他软件中进行分析和处理。
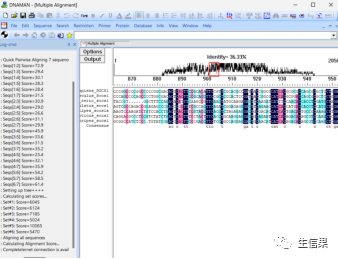
到这里核酸比对就结束啦,可以看到保守性比较较高的区域都用相同的色彩标记出来了,移动上方的红色框线可以查看不同区域。
如果你还在为生信分析苦恼,苦苦学习却不得要领,一定要试一下这个线上平台,什么circos图、小提琴图、曼哈顿图只要上传数据统统不在话下!
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

