序列比对金标准BWA·真保姆级教程
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

随着illumina为主的NGS测序技术的流行,测序成本在短短十年内飞速下降,现在测序的成本仅仅是十年前的零头,越来越多课题组选择基因组学开展研究。
其中,短序列比对(mapping)是NGS分析的重中之重,目前流行的外显子、WGS、GBS都需要用到短序列比对。在众多短序列比对中,BWA和bowtie2是其中的佼佼者,随着三代测序long read的兴起,BWA也及时跟进了算法,增加了split reads和RNAseq的比对,让BWA隐隐有成为金标准的趋势。
目前BWA支持三种算法,分别是bwa-backtrack,bwa-sw和bwa-mem,其中backtrack主要用于100bp以下的reads,目前较少应用。sw和mem算法都支持long read和split比对,支持70bp到1M的reads,其中mem算法更新、更快、更强,可用于illumina、454、sanger等多个平台,是官方流程推荐的算法,很明显,跟着官方workflow走就对了。接下来跟着小果进入实操环节吧。
首先当然是软件安装环节,小果作为资深的生信狗,不要跟我提conda,什么bioconda,什么一键安装。不可能,绝对不可能,我果小果就是累死,所有头发都薅秃,也不会用你一次conda。
咳咳,首先让我们打开conda,安装bwa
$ conda install -c bioconda bwa
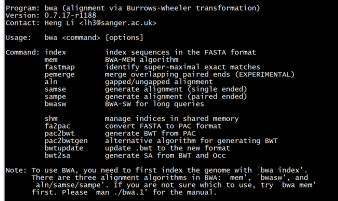
参数如图所示,在bwa工作流程中,序列比对分为两步,首先需要使用bwa index建立索引,其中-a可以指定索引的算法,其中包括bwtsw、is和rb2,is适合用于参考基因组较小的物种,如细菌,人类基因组则需要使用bwtsw。位于中间的基因组选哪个都行。
$bwa index human_glk_v37.fasta人家说可以加-a,也没说一定要加呀。
构建完index后就可以进行序列比对了,bwa mem同时支持单端测序和双端测序序列比对代码如下所示。
$ bwa mem -R '@RGtID:E1602061tSM:bartLB:library1' -t 4 reference.fnareads.1.fq.gz reads.2.fq.gz >result.sam
其中-R里的内容非常重要,不同lane、文库、样本依赖RG进行区分后续gatk进行snp calling时会检查-R是否缺失,-t是指计算的线程数,reads1和reads2指双端测序的两条reads,单端测序也可以只填一个,输出的result支持sam和bam两种格式。
至此,fastq已经转化为bam文件了,欲知后续分析如何进行,欢迎关注微信公众号。

点击“阅读原文”立刻拥有
↓↓↓