复杂热图绘制秘籍:用ComplexHeatmap创造数据的视觉奇迹!

hello,相信大家已经对Heatmap这个R包轻车熟路啦,那么你知道如何在热图中展示不同样本的相对丰度嘛?今天小果就来教大家如何用ComplexHeatmap包绘制复杂热图,感兴趣的话就和小果一起看下去吧!!
Heatmap和ComplexHeatmap的区别
在使用之前,小果先来带大家区分一下这两个R包吧!
·功能和灵活性:
Heatmap是R语言中基本的绘制热图的函数,它可以用于简单的热图绘制,但功能相对较为有限。它主要用于展示数值矩阵的颜色编码,不支持添加分组注释、行列聚类等高级功能。ComplexHeatmap是一个由Zuguang Gu开发的R包,它在Heatmap基础上提供了更多高级功能和灵活性。通过 ComplexHeatmap,你可以自定义热图的各个方面,如添加分组注释、自定义颜色映射、行列聚类、调整注释等。它还提供了一系列的辅助函数和参数,使得用户可以进行更复杂、更精细的热图绘制。
·代码复杂度:
由于ComplexHeatmap提供了更多的功能和选项,其使用相对复杂一些,需要更多的代码来完成定制化的热图绘制。Heatmap则相对简单,适用于简单的热图需求或快速实验。
·社区支持:
ComplexHeatmap作为一个专门的R包,拥有更大的社区支持和更广泛的用户群体。你可以在其官方文档、GitHub上找到大量的示例、文档和交流资源,以便在使用时获取帮助和解决问题。相比之下,Heatmap作为R语言的基本函数,其文档和示例相对较少。
综上所述,ComplexHeatmap相对于Heatmap提供了更多的高级功能和灵活性,但也更复杂一些。如果你需要进行复杂的热图绘制,并添加分组注释、调整颜色映射等高级功能,推荐使用ComplexHeatmap。如果你只需要简单的热图展示,或者对R语言不太熟悉,可以使用Heatmap函数完成基本的热图绘制哦。
好啦,现在你了解二者的区别了嘛?那接下来进入我们今天的正题!
ComplexHeatmap绘制复杂热图
·安装与导入R包:
首先,大家可以通过以下代码安装R包哦:
if (!require(“BiocManager”))
install.packages(‘BiocManager’)
if (!require(“ComplexHeatmap”))
BiocManager::install(‘ComplexHeatmap’)
library(ComplexHeatmap)
·准备数据
1.为了绘制热图,我们需要准备相对丰度矩阵及其对应的分组数据表格。小果已经帮你们准备好这两个文件啦,在此将它们命名为”test.csv”和”group.csv”。确保这两个文件要与R脚本位于同一目录下哦:
df <- read.csv(“test.csv”, header = T)
group_df <- read.csv(“group.csv”, header = F)
·数据处理
在开始绘制热图之前,我们需要对数据进行一些处理。首先,我们将按”Class”和”Family”列对矩阵的所有行进行排序。
df <- df[order(df$Class, df$Family), ]
然后,我们将从数据框中提取相对丰度数据,并将其转换为矩阵格式:
mat <- df[-c(1, 2, 3)]
mat <- as.matrix(mat)
row.names(mat) <- df[, 3]
最后,我们获得 class-family-species 对应表格:
df_annotation <- df[c(1, 2, 3)]
·绘制一个简单的热图
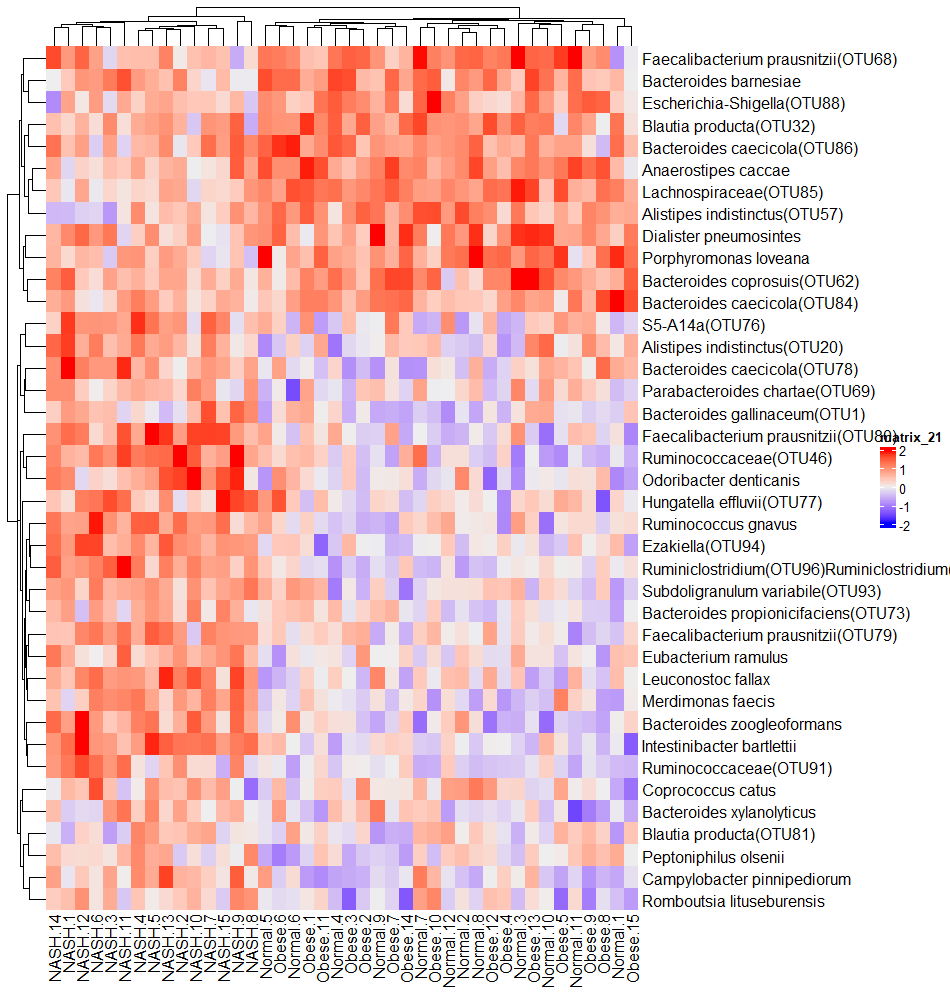
为了与后面的热图做比较,我们先来绘制一个简单的热图:
ht <- Heatmap(mat)
绘制的热图如下所示:
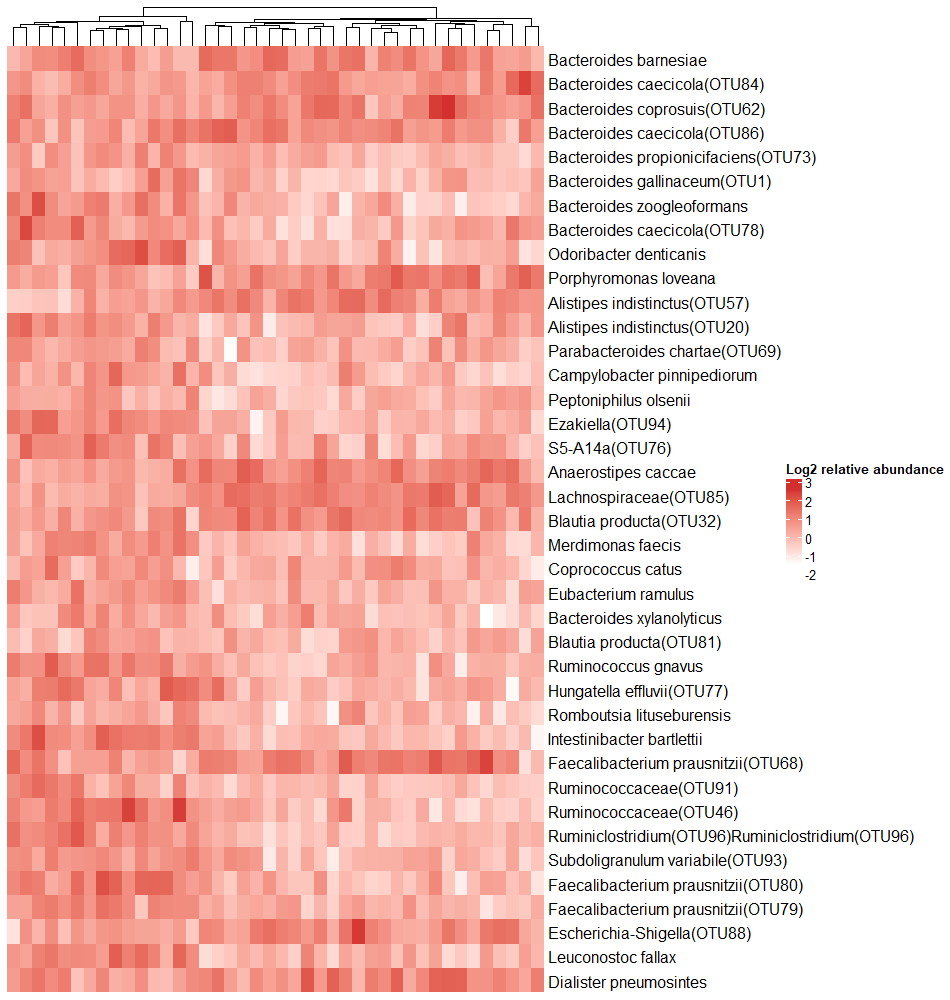
然后,我们可以调整参数来简单的美化一下热图。例如,我们可以设置不按行聚类、不展示列名,并设置
热图图例的名称和颜色:
ht <- Heatmap(mat,
cluster_rows = F,
show_column_names = F,
heatmap_legend_param = list(title = “Log2 relative abundance”),
col = c(“#FFFFFF”, “#D32F2F”))
·添加行分组注释和列分组注释
接下来,我们可以生成列分组注释。为此,我们需要设置列分组的颜色和图例顺序:
top_col <- c(“Normal”=”#2196F3”, “Obese”=”#FFEB3B”, “NASH”=”#E91E63”)
top_anno <- HeatmapAnnotation(State = group_df[, 2],
col = list(State = top_col),
annotation_legend_param = list(State = list(at=c(“Normal”, “Obese”, “NASH”))))
同样地,我们也可以生成行分组注释。设置行分组的颜色和图例顺序:
left_col <- c(“Bacteroidaceae”=”#1976D2”, “Marinifilaceae”=”#2196F3”, “Porphyromonadaceae”=”#03A9F4”, “Rikenellaceae”=”#00BCD4”, “Tannerellaceae”=”#B3E5FC”,
“Campylobacteraceae”=”#F8BBD0”,
“Family XI”=”#D32F2F”, “Family XIII”=”#F44336”, “Lachnospiraceae”=”#E64A19”, “Peptostreptococcaceae”=”#FF5722”, “Ruminococcaceae”=”#FFCCBC”,
“Enterobacteriaceae”=”#4CAF50”,
“Leuconostocaceae”=”#FFEB3B”,
“Veillonellaceae”=”#673AB7”)
left_anno <- rowAnnotation(Family = df_annotation[, 2],
show_annotation_name = F,
col = list(Family = left_col),
annotation_legend_param = list(
Family = list(at=c(“Bacteroidaceae”, “Marinifilaceae”,
“Porphyromonadaceae”, “Rikenellaceae”,
“Tannerellaceae”, “Campylobacteraceae”,
“Family XI”, “Family XIII”, “Lachnospiraceae”,
“Peptostreptococcaceae”, “Ruminococcaceae”,
“Enterobacteriaceae”, “Leuconostocaceae”,
“Veillonellaceae”))))
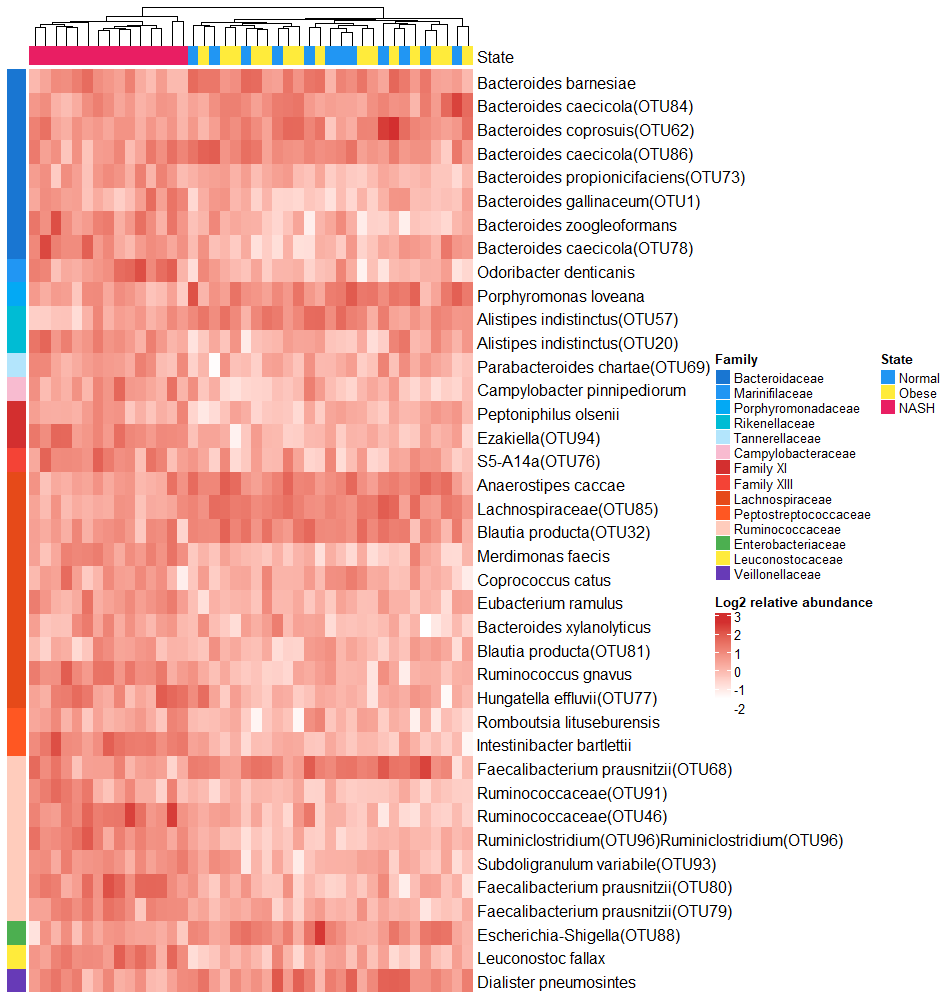
最后,我们可以将之前的设置与热图对象合并,并绘制出最终的热图:
ht <- Heatmap(mat,
cluster_rows = F,
show_column_names = F,
heatmap_legend_param = list(title = “Log2 relative abundance”),
col = c(“#FFFFFF”, “#D32F2F”),
top_annotation = top_anno,
left_annotation = left_anno)
优化后的热图展示如下:
你以为这就是最终结果了嘛?No!!我们还可以进一步细化:
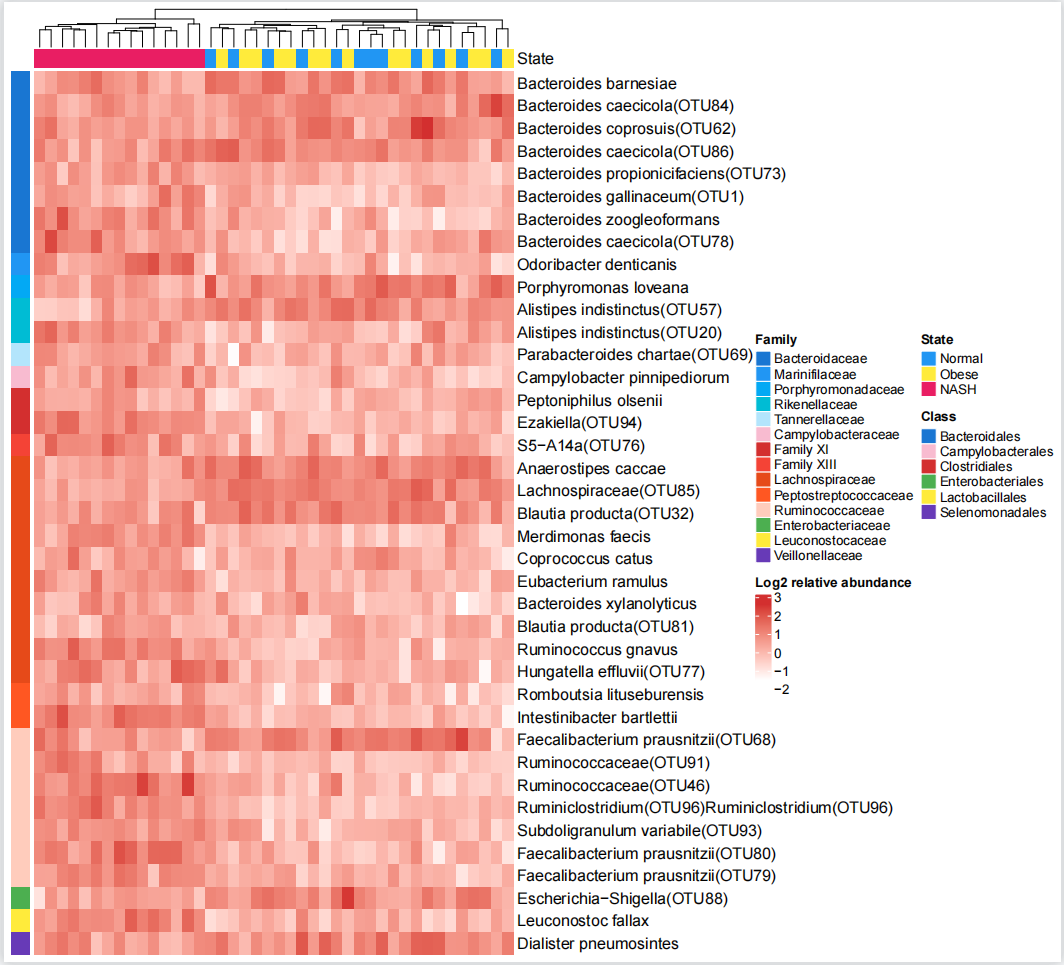
·给Class单独添加图例
如果需要单独绘制一个图例,可以按照以下步骤进行操作:
class <- unique(df_annotation$Class)
然后,我们创建一个 Legend 对象,并设置标签、标题和图例颜色:
lgd <- Legend(labels = class, title = “Class”,
legend_gp = gpar(fill = c(“#1976D2”, “#F8BBD0”, “#D32F2F”, “#4CAF50”, “#FFEB3B”, “#673AB7”)))
pdf(“plot.pdf”, width = 11, height = 10)
draw(ht, ht_gap = unit(7, “mm”), annotation_legend_list = lgd)
dev.off()
好啦,让我们来一起看看绘制的最终结果吧!!
怎么样,你学会怎么使用ComplexHeatmap包了嘛?更多学习干活请多多关注小果哦!
往期推荐