「付费」【代码分享-17】一文拿捏,如何挖掘TCGA肿瘤数据,跟免疫建立联系

今天小果给小伙伴带来的分享非常实用,首先通过下载TCGA表达矩阵,临床信息和突变信息文件,然后提取自己感兴趣癌症的样本和临床信息以及感兴趣基因的表达量和突变信息,最后利用MCPcounter算法计算免疫细胞含量,通过pheatmap包绘制热图,有需要的小伙伴可以跟着小果开始今天的实操学习。
1.准备需要的R包
#安装需要的R包install.package("devtools")library(devtools)install_github("ebecht/MCPcounter",ref="master", subdir="Source")install.packages("stringr")install.packages("data.table")install.packages("pheatmap")#加载需要的R包library(MCPcounter)library(stringr)library(data.table)library(pheatmap)
2.输入数据
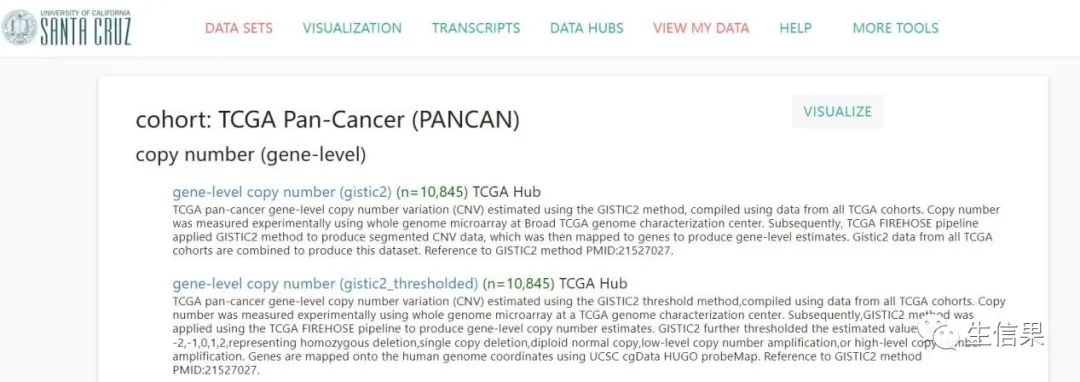
输入数据都通过该网站下载:https://xenabrowser.net/datapages/?cohort=TCGA%20Pan-Cancer%20(PANCAN)&removeHub=https%3A%2F%2Fxena.treehouse.gi.ucsc.edu%3A443
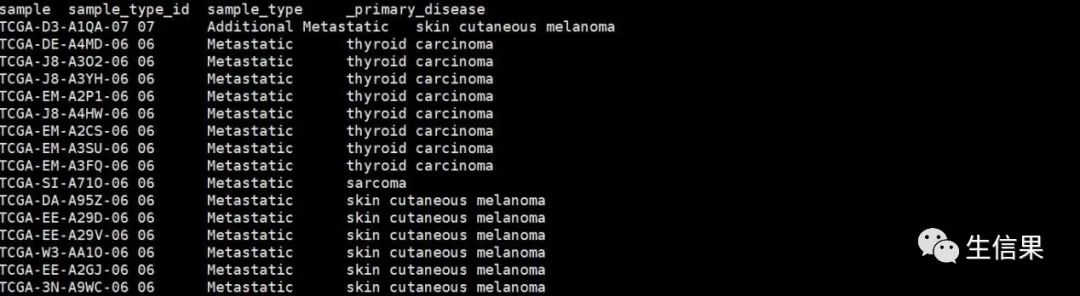
#读取表型数据TCGA_phenotype_denseDataOnlyDownload.tsv
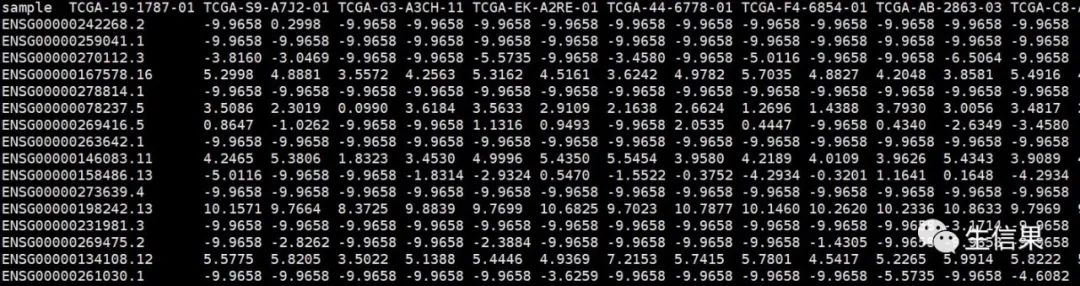
#基因表达矩阵文件tcga_RSEM_gene_tpm
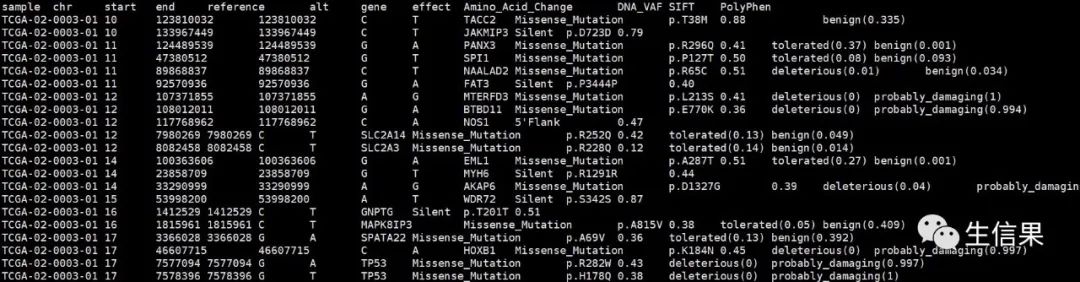
#突变信息文件
#临床信息文件,包含样本信息,癌症类型等信息
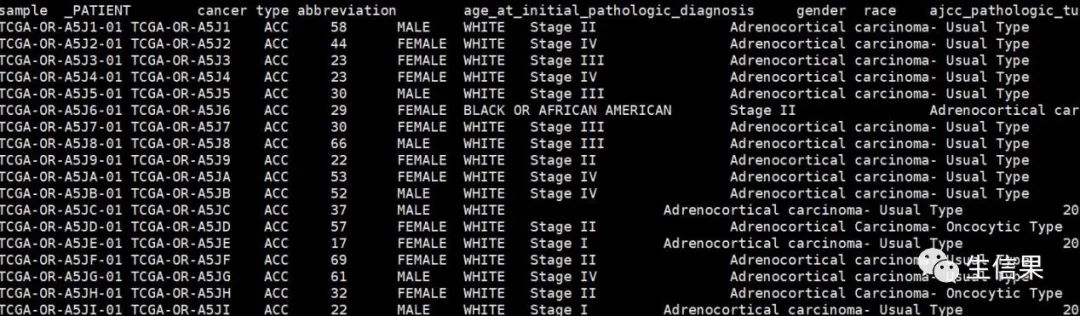
#基因注释文件,第一列为ENSEMBL ID,第二列为Gene symbol。
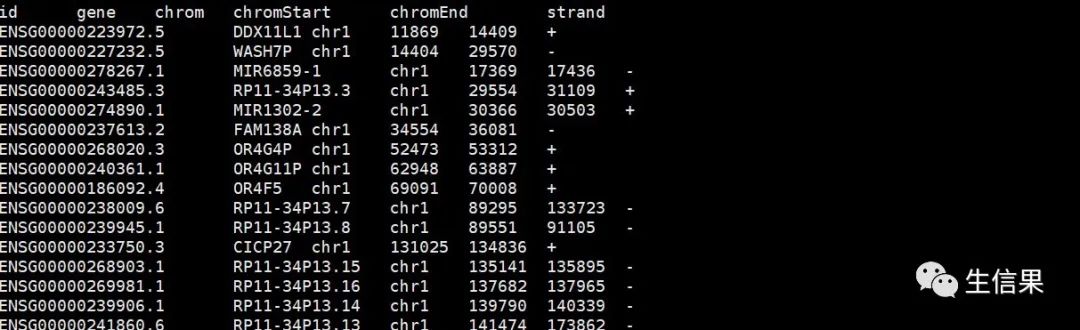
3.结果文件
3.1MCPcounter.pdf
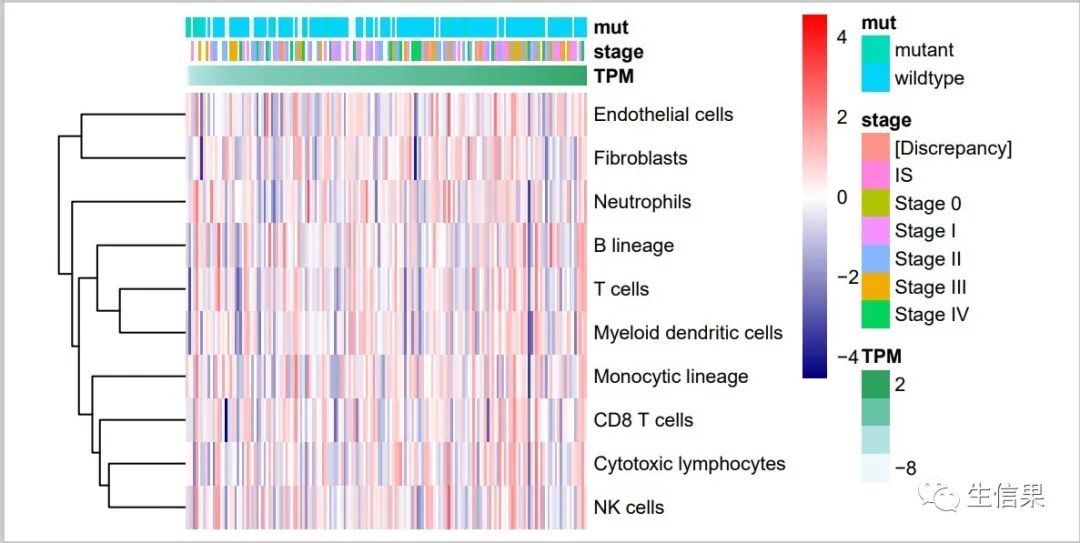
3.2MCPcounter_output.csv
该结果文件为通过MCPcounter方法计算的目标癌症所有样本免疫细胞含量结果文件;行名为免疫细胞类型,列名为样本名。
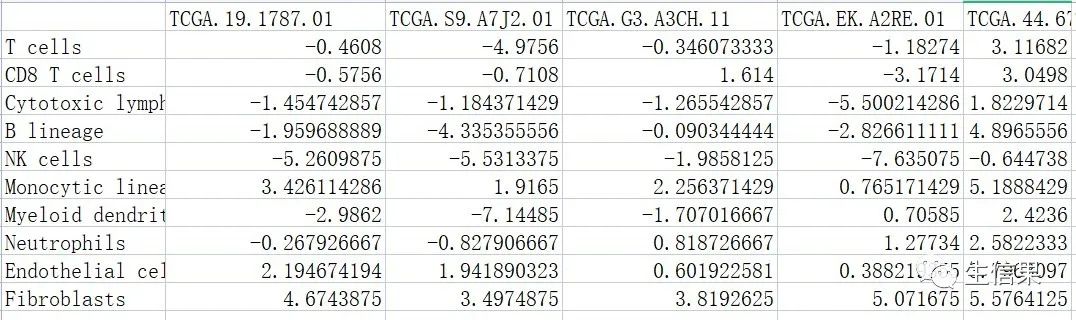
3.3easy_input_cli.csv
该结果文件为整理的临床和突变信息文件。
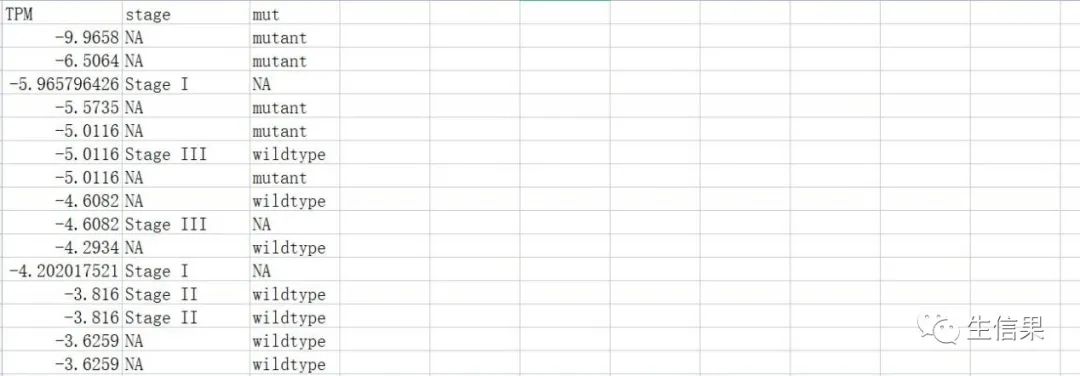
最终小果通过TCGA数据下载,下载了基因表达矩阵文件,临床信息,表型信息和突变信息文件,然后进行免疫浸润分析,绘制了免疫细胞含量,临床信息和突变信息的热图,欢迎大家和小果一起讨论学习呀!

往期推荐
如果需要完整的代码可以点击付费获取哦!今天小果的分享就到这里,如果小伙伴有其他数据分析需求,可以尝试本公司新开发的生信分析小工具云平台,季代码完成分析,非常方便奥!
(扫码领取正好的输入文件,代码文件及示例结果)