肿瘤生信文章必备分析内容-亚型生存与临床相关性分析
今天小果为小伙伴带来的分享内容为肿瘤亚型生存与临床相关性分析,首先利用 survival包中的 Kaplan–Meier曲线方法评估不同亚型之间生存预后相关性,分析不同亚型之间的生存预后信息是具有显著差别,之后对基因表达水平在亚型样本中的分布进行热图绘制,最后整理样本临床信息(年龄、性别等),对亚型与样本的临床因素进行相关性分析;这就是小果今天带来的内容,在肿瘤生信文章中亚型相关分析出现的频率非常高,灰常值得小伙伴学习哈,接下来跟着小果开始今天的学习吧!
1.如何进行亚型生存与临床相关性分析?
如何进行亚型生存与临床相关性分析?小果来为小伙伴做一哈简单介绍,在进行该分析之前,首先利用ConsensusClusterPlus对肿瘤样本进行无监督聚类分析,获得亚型分组信息,然后进行不同亚型之间预后相关性分析,亚型与样本的临床因素相关性分析,这就是大概的分析步骤,其实很简单,非常适合小白,有需要的小伙伴马上跟着小果开始今天的实操吧!

3.读取输入数据
#基因表达矩阵,行名为基因名,列名为样本信息。rt=read.table("热图1.txt",header=T,sep="t",row.names=1,check.names=F)
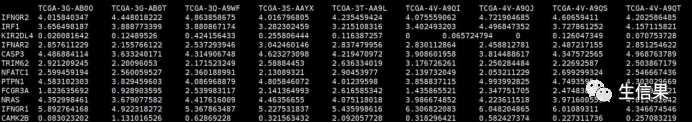
#临床信息和亚型分组信息文件,行名为样本信息文件,第一列为亚型分组信息,其他列为临床因子信息。ann=read.table("热图2.txt",header=T,sep="t",row.names=1,check.names=F)
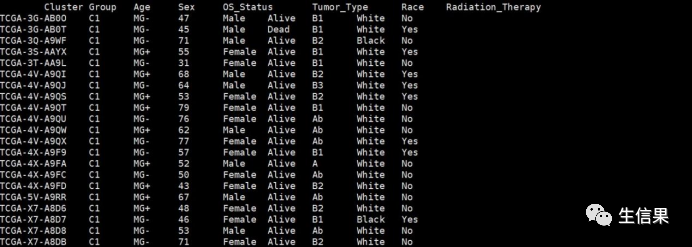
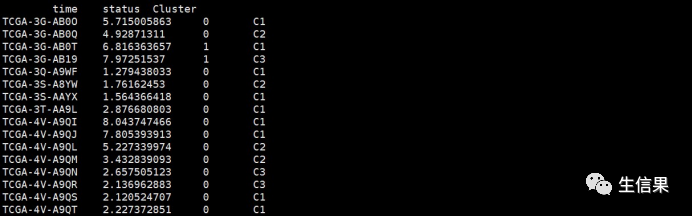
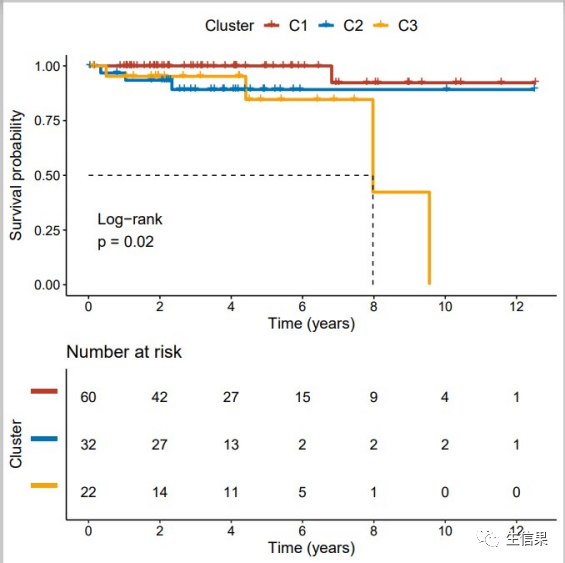
4.不同亚型之间生存预后相关性分析,以及绘制KM曲线
#行名为样本名,列名为生存时间,生存状态和亚型分组信息tmp <- read.table(file = "km.txt",#工作目录下的文件名header = T,#设置列名sep = "t",#分隔符row.names = 1)#设置行名Cluster
fit <- survfit(Surv(time,status) ~ Cluster,data = tmp)#设置主题mytheme <- theme_survminer(font.legend = c(14,"plain", "black"),font.x = c(14,"plain", "black"),font.y = c(14,"plain", "black"))#绘制KM曲线pdf("KM.pdf",height=7,width=7)ggsurvplot(fit,palette= c(pal_nejm()(2),'#F7A20B'),#pal_nejm()(2)数字是2代表3组conf.int=FALSE,size=1.3,pval=T,pval.method = T,legend.labs=c("C1","C2","C3"),legend.title="Cluster",xlab="Time (years)",ylab='Survival probability',risk.table=TRUE,break.time.by = 2,risk.table.title="Number at risk",risk.table.height=.4,risk.table.y.text = FALSE,surv.median.line = "hv",ggtheme = mytheme)dev.off()
5.亚型与样本的临床因素进行了相关性分析
#制作基线表,以Cluster亚型进行分组table1<-gaze(Cluster~.,ann)%>%myft()#导出word文件,在当前目录生成Report.doctable2docx(table1)

6.基因表达水平在亚型样本中的分布进行热图绘制
pdf(file="heatmap.pdf",width=8,height=7)pheatmap(rt,annotation=ann,cluster_cols = F,cluster_rows = T,color = colorRampPalette(c("CornflowerBlue", "white", "Salmon"))(50),show_colnames = F,scale="row",fontsize = 8,fontsize_row=7.5,fontsize_col=6)dev.off()
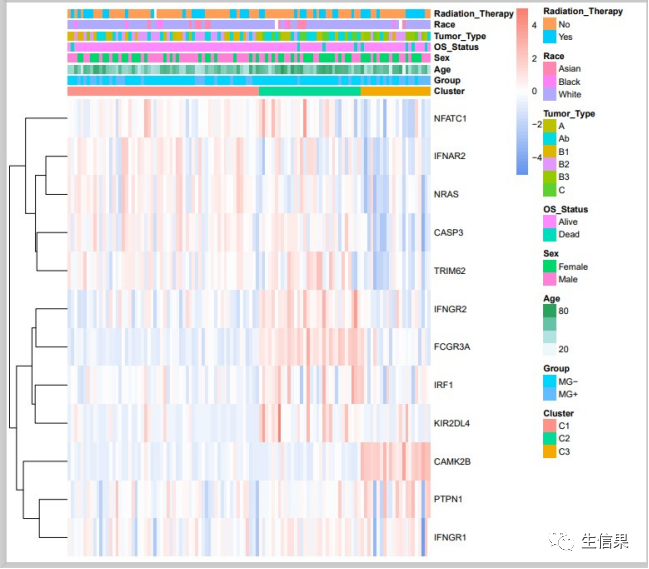
今天小果顺利的完成了亚型的生存与临床相关性分析,想要继续了解其他内容的可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~
往期推荐

