肿瘤生信文章必备分析内容,预测对免疫疗法的反应(TIDE评分)

今天小果为小伙伴带来的分享为使用肿瘤免疫功能障碍和排斥(TIDE)预测算法评估两种亚型的免疫治疗反应
今天小果为小伙伴带来的分享为使用肿瘤免疫功能障碍和排斥(TIDE)预测算法评估两种亚型的免疫治疗反应,该分析在肿瘤相关生信文章中出现的频率非常高,是一个值得小伙伴学习的分析内容,接下来马上和小果一起开始今天的分享学习吧!
准备需要的R包
#安装需要的R包install.packages("ggplot2")install.packages("tidyverse")install.packages("cowplot")install.packages("ggsignif")install.packages("ggsci")#加载需要的R包library(tidyverse)library(ggplot2)library(ggsci)library(ggpubr)library(ggsignif)
如果需要完整的代码可以点击付费获取哦!今天小果的分享就到这里,如果小伙伴有其他数据分析需求,可以尝试本公司新开发的生信分析小工具云平台,季代码完成分析,非常方便奥!

输入数据
#EXP.txt基因表达矩阵,行名为基因名,列名为样本名dat <- read.table("EXP.txt",sep = "t",row.names = 1,header = T,stringsAsFactors = F,check.names = F)
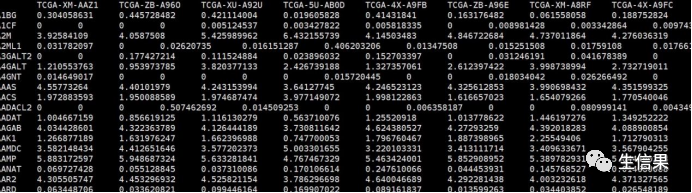
#将样本进行标准化,将样本按均值标准化,表达量减去每个基因所在样本的均值(按行计算均值,在用每个表达量-均值)exp<-t(apply(dat,1,function(x){x-(mean(x))}))#group.txt,风险评分高低分组样本信息文件,第一列为样本名,第二列为高低分组信息ann <- read.table("group.txt",sep = "t",header = T,stringsAsFactors = F,check.names = F)

TIDE评分计算
#TIDE的计算痛过该网址:http://tide.dfci.harvard.edu/login/
在利用该网站计算之前,首先要利用邮箱进行注册,如下图:
#点击Predictresponse就可以进行分析,得到最后分析结果,点击Export to CSV下载TIDE分析结果。
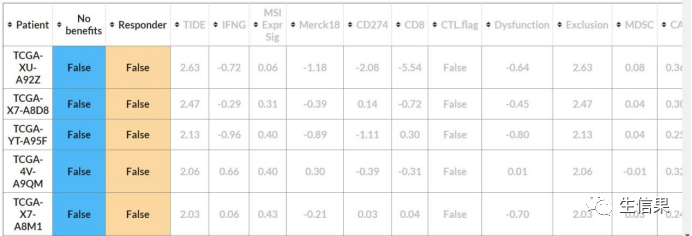
TIDE评分在高低风险组中的显著差异分析
#读取TIDE结果文件,包括样本名,TIDE,Exclusion等列信息。TIDE<-read.csv("export.csv",header=T)
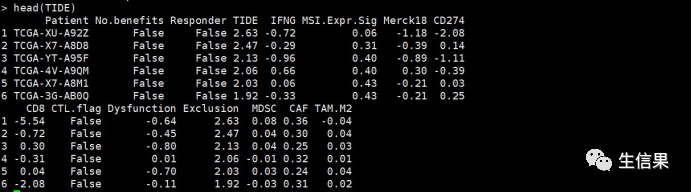
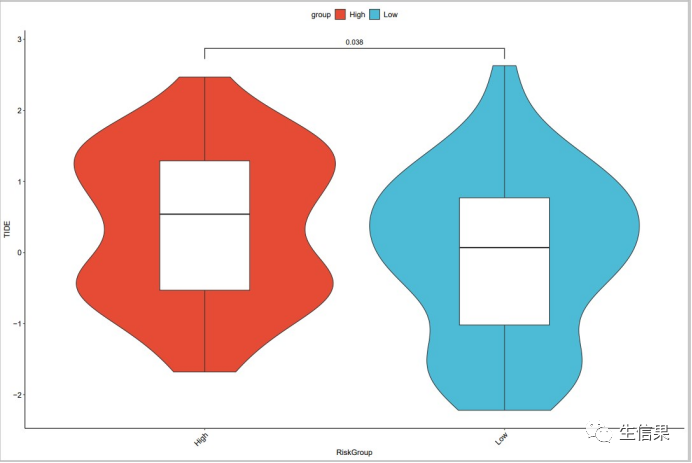
#修改列名TIDE<-rename(TIDE,sample=Patient)#连接TIDE结果文件和高低风险组信息文件final<-left_join(TIDE,ann,by="sample")#绘制TIDE评分显著差异小提琴图+箱线图boxplot_TIDE<- ggplot(final, aes(x = group, y = TIDE))+labs(y="TIDE",x= "RiskGroup")+geom_violin(aes(fill=group))+geom_boxplot(width=0.3)+scale_fill_npg()+#修改主题theme_classic() +theme(axis.title = element_text(size = 12,color ="black"),axis.text = element_text(size= 12,color = "black"),panel.grid.minor.y = element_blank(),panel.grid.minor.x = element_blank(),axis.text.x = element_text(angle = 45, hjust = 1 ),panel.grid=element_blank(),legend.position = "top",legend.text = element_text(size= 12),legend.title= element_text(size= 12)) +#显著差异分析geom_signif(comparisons=list(c("High","Low")),test=t.test,step_increase=0.2,map_signif_level=F)ggsave(file="TIDE.pdf",boxplot_TIDE,height=10,width=15)

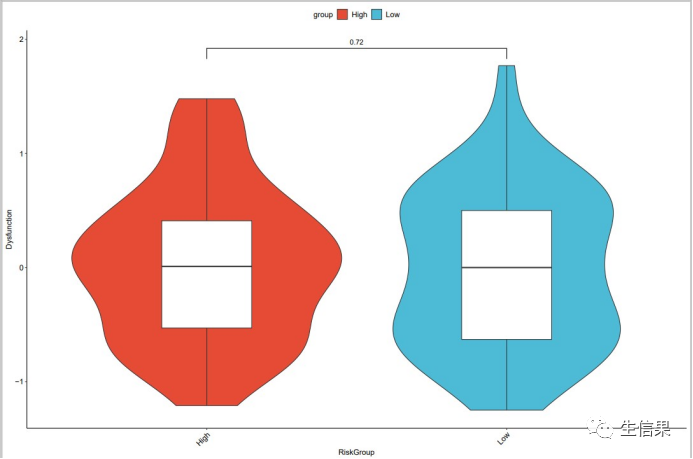
##绘制Dysfunction显著差异小提琴图+箱线图boxplot_Dysfunction<- ggplot(final, aes(x = group, y =Dysfunction ))+labs(y="Dysfunction",x= "RiskGroup")+geom_violin(aes(fill=group))+geom_boxplot(width=0.3)+scale_fill_npg()+#修改主题theme_classic() +theme(axis.title = element_text(size = 12,color ="black"),axis.text = element_text(size= 12,color = "black"),panel.grid.minor.y = element_blank(),panel.grid.minor.x = element_blank(),axis.text.x = element_text(angle = 45, hjust = 1 ),panel.grid=element_blank(),legend.position = "top",legend.text = element_text(size= 12),legend.title= element_text(size= 12)) +geom_signif(comparisons=list(c("High","Low")),test=t.test,step_increase=0.2,map_signif_level=F)ggsave(file="Dysfunction.pdf",boxplot_Dysfunction,height=10,width=15)

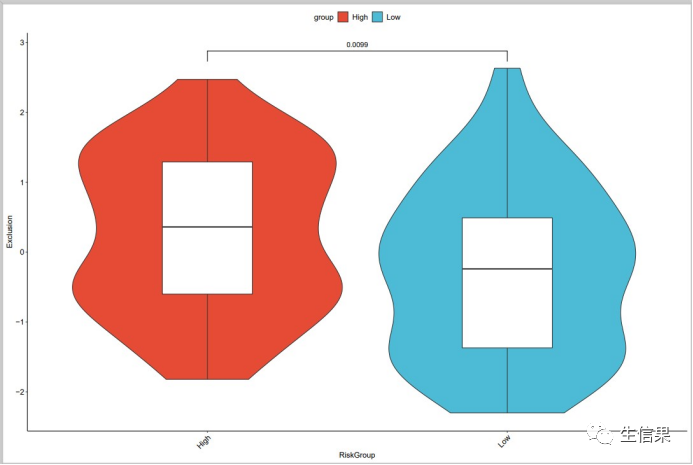
###绘制Exclusion显著差异小提琴图+箱线图boxplot_Exclusion<- ggplot(final, aes(x = group, y =Exclusion ))+labs(y="Exclusion",x= "RiskGroup")+geom_violin(aes(fill=group))+geom_boxplot(width=0.3)+scale_fill_npg()+#修改主题theme_classic() +theme(axis.title = element_text(size = 12,color ="black"),axis.text = element_text(size= 12,color = "black"),panel.grid.minor.y = element_blank(),panel.grid.minor.x = element_blank(),axis.text.x = element_text(angle = 45, hjust = 1 ),panel.grid=element_blank(),legend.position = "top",legend.text = element_text(size= 12),legend.title= element_text(size= 12)) +geom_signif(comparisons=list(c("High","Low")),test=t.test,step_increase=0.2,map_signif_level=F)ggsave(file="Exclusion.pdf",boxplot_Exclusion,height=10,width=15)
###绘制MSI显著差异小提琴图+箱线图boxplot_MSI<- ggplot(final, aes(x = group, y =MSI.Expr.Sig))+labs(y="MSI",x= "RiskGroup")+geom_violin(aes(fill=group))+geom_boxplot(width=0.3)+scale_fill_npg()+#修改主题theme_classic() +theme(axis.title = element_text(size = 12,color ="black"),axis.text = element_text(size= 12,color = "black"),panel.grid.minor.y = element_blank(),panel.grid.minor.x = element_blank(),axis.text.x = element_text(angle = 45, hjust = 1 ),panel.grid=element_blank(),legend.position = "top",legend.text = element_text(size= 12),legend.title= element_text(size= 12)) +geom_signif(comparisons=list(c("High","Low")),test=t.test,step_increase=0.2,map_signif_level=F)ggsave(file="MSI.pdf",boxplot_MSI,height=10,width=15)
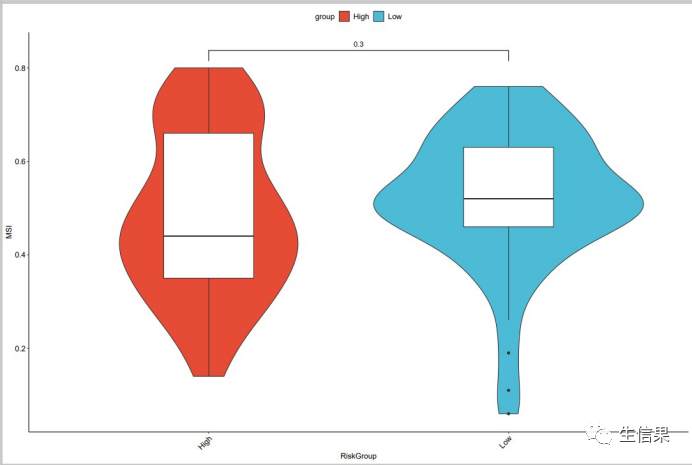
最终小果成功的完成了TIDE评分的计算,看起来图片效果非常不错,欢迎大家和小果一起讨论学习呀
往期推荐
