一分钟教会你在R语言中使用CIBERSORT进行免疫浸润分析
点击蓝字 关注我们
在我们平时进行疾病研究分析过程中必不可免的需要明确免疫细胞在人体微环境内的组成情况,从而能更好的知道在疾病的发生发展过程中究竟是哪种免疫细胞产生了重要作用,这有助于阐明疾病免疫应答的机制,进而指导设计合理的治疗方案。目前,能用于免疫浸润分析的方法有很多,比如 ssGSEA, CIBERSORT, TIMER, EPIC 等。
CCIBERSORT是目前在文献中引用次数最多的免疫细胞浸润分析工具。该工具于2015年在Nature method上首次发表。CIBERSORT是基于线性支持向量回归(linear support vector regression)原理对人类免疫细胞亚型的表达矩阵进行去卷积的工具。芯片表达矩阵和测序表达矩阵均可用该工具进行分析,相较于其他方法,CIBERSORT对未知混合物和含有相近的细胞类型的表达矩阵的去卷积分析能力要更强。该方法是基于默认提供22种免疫细胞亚型的基因表达特征集:LM22来进行分析的。
CIBERSORT支持网页版和本地两种方法。但使用网页版需要先进行账号注册,而且需要用组织邮箱(比如edu的邮箱),这对于一些同学来说很不方便。因此今天小花就来教大家在R语言中使用CIBERSORT进行免疫浸润分析。
首先是数据准备,我们需要准备以下三个文件并放在同一个文件夹中:
(1)LM22.txt获取方法:
在Cibersort论文中(https://www.nature.com/articles/nmeth.3337#MOESM207)下载Supplementry table 1,然后只将其中矩阵部分保留并保存为制表符分割的txt。

(2)Cibersort.R获取方式:
在R中新建一个R Script,复制这个网站(https://rdrr.io/github/singha53/amritr/src/R/supportFunc_cibersort.R)中的内容,保存为“Cibersort.R”即可。
(3)自己的表达矩阵:
直接用前面下载的分析得到的表达矩阵就行了,第一列是基因名,第一行是样品名,不能有重复基因名,第一列列名不能空白,矩阵中不能存在空白或NA值。还有就是第一列的列名需要与LM22.txt第一列列名一致(小花这里都为GeneSymbol)。然后也将其保存为制表符分割的txt。
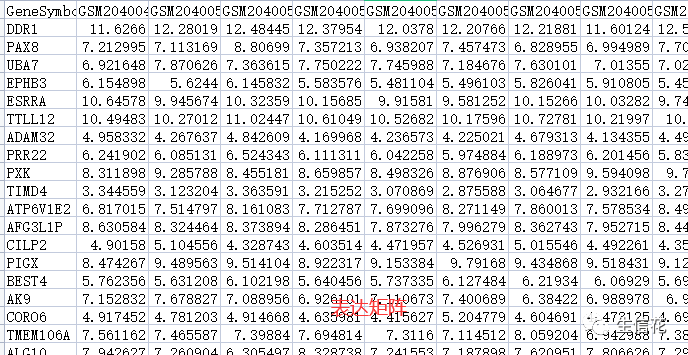
然后就可以开始分析了。大家跟着小花一起来操作一遍吧!
第一步:进入工作目录并加载分析所需的R包
setwd("三个文件所在的文件夹") #进入工作目录install.packages('e1071')install.packages('preprocessCore')library(e1071)library(preprocessCore)library(parallel) #安装并加载所需R包
第二步:进行CIBERSORT分析
source("Cibersort.R") # 加载R脚本result1 <- CIBERSORT("LM22.txt", "expr.txt", perm = 1000, QN = T) #运行R脚本进行CIBERSORT分析;#perm:表示置换次数,数字越大运行时间越长,一般文章都设置为1000;#QN:如果为芯片数据这里设为“T”;如果为测序数据设为“F”最后会在工作目录下自动生成结果文件“CIBERSORT-Results.txt”。
最后会在工作目录下自动生成结果文件“CIBERSORT-Results.txt”。
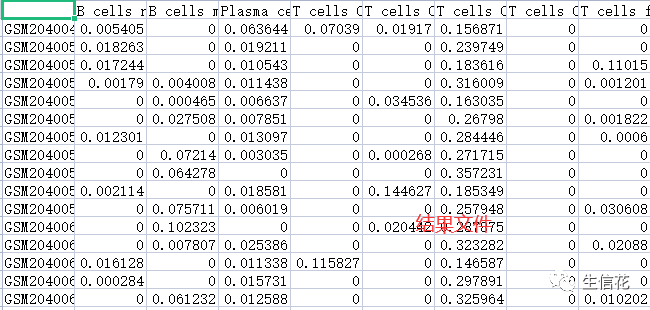
拿到结果文件以后我们就可以根据自己研究的分组情况来绘制箱线图,看在两者之间各免疫细胞的差异情况。首先我们这里除了上面得到的结果文件,还需要准备一个分组文件,如图所示。

好了一切准备就绪,跟着小花一起来绘制箱线图吧!
第一步:加载分析所需R包以及导入数据
library(tidyverse)b <- read.table("group.txt",sep = "t",row.names = 1,check.names = F,header = T)a <- read.table("CIBERSORT-Results.txt",sep = "t",row.names = 1,check.names = F,header = T)
第二步:对数据进行一个简单处理,使其能用于后续分析
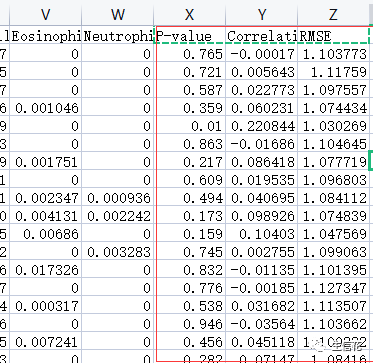
在我们免疫浸润分析得到的结果文件最后有三列内容“P-value,Correlation和RMSE”后续分析是用不到的,所以我们需要将其删掉。
a <- a[,1:22] #去除浸润分析结果文件后三列
然后我们还需要判断一下,两个文件中样本的顺序是否一致,因为后面需要将两个文件的信息进行合并,如果不是一致的会导致分析错误。
identical(rownames(a),rownames(b)) #判断两个文件中样本顺序是否一致,返回值为“TRUE”,则是一致的。
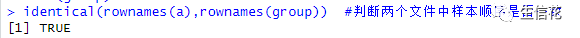
class(b$group)a$group <- b$group #将分组信息添加到a后面a <- a %>% rownames_to_column("sample") #把行名变成一列,并加上“sample”library(ggsci)library(tidyr)library(ggpubr)b <- gather(a,key=CIBERSORT,value = Proportion,-c(group,sample))#绘制箱线图ggboxplot(b, x = "CIBERSORT", y = "Proportion",fill = "group", palette = "lancet")+stat_compare_means(aes(group = group),method = "wilcox.test",label = "p.signif",symnum.args=list(cutpoints = c(0, 0.001, 0.01, 0.05, 1),symbols = c("***","*","ns")))+theme(text = element_text(size=10),axis.text.x = element_text(angle=45, hjust=1))dev.off()
最后我们就能得到想要的结果了。
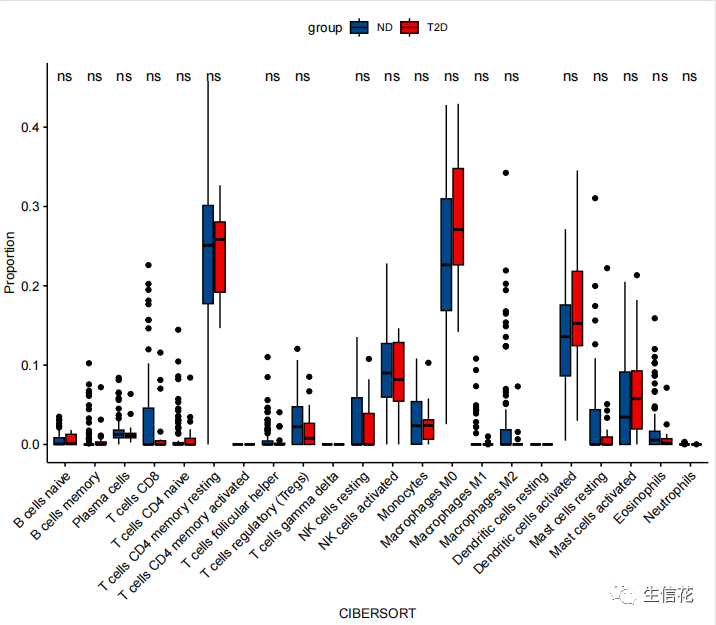
好了!今天的内容分享就到这里了。大家有任何问题都可以留言和小花讨论哦!我们下期再见!
欢迎使用:云生信 – 学生物信息学 (biocloudservice.com)
如果想用服务器可以联系微信:18502195490(快来联系我们使用吧!)

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

