【手把手教你】Cytoscape—基因互作网络图的保姆级讲解
点击蓝字
关注小图
大家还记不记得小图之前有提过Cytoscape,今天就是小图给大家兑现承诺的时候!
Cytoscape是一款使用很广泛的网络图绘制美化软件,是现在组学数据后期处理不可或缺的好用工具,今天小图就给大家详细介绍下Cytoscape的图片美化操作。
我们马上开始学起来~
一、数据准备
首先,你得先下载Cytoscape
(1)导入STRING互作分析结果数据“string_interactions_short.tsv”,数据中node1与node2为一对节点,combined_score为互作分数。
导入途径:File->Import->Network from File->OK。操作界面如下图:
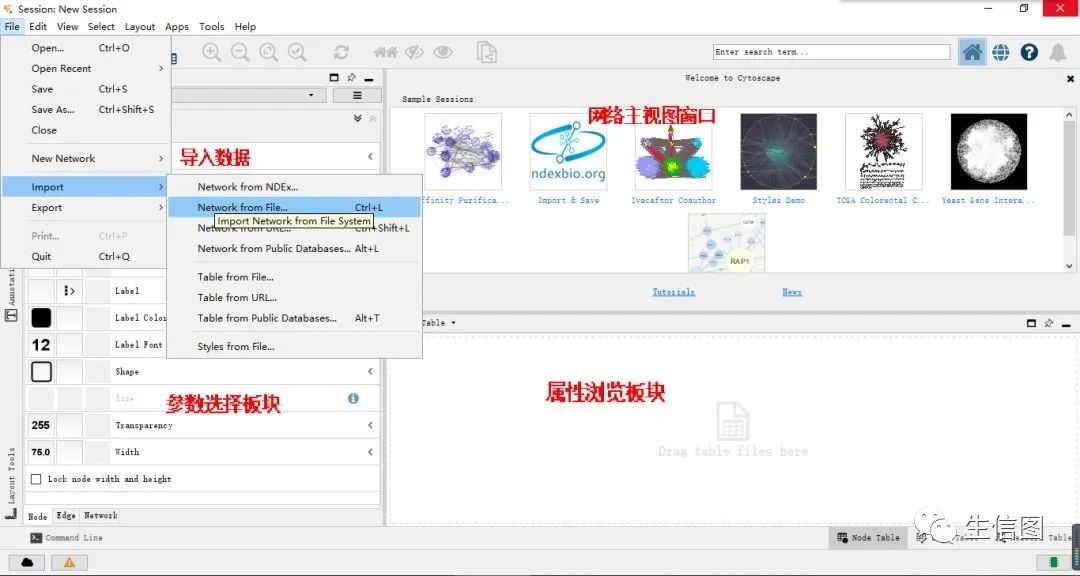
(2)进行节点选择。操作界面如下图:
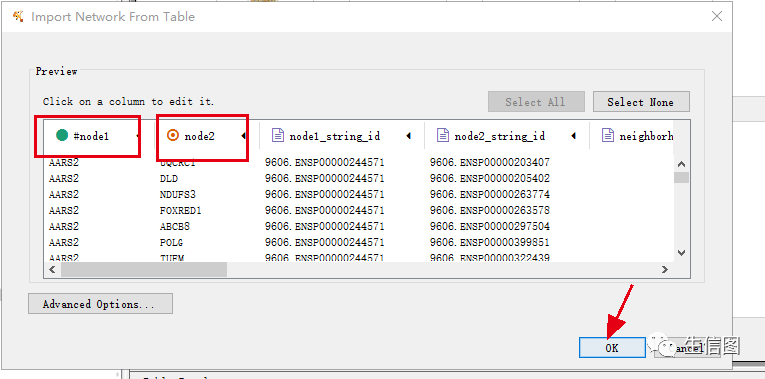
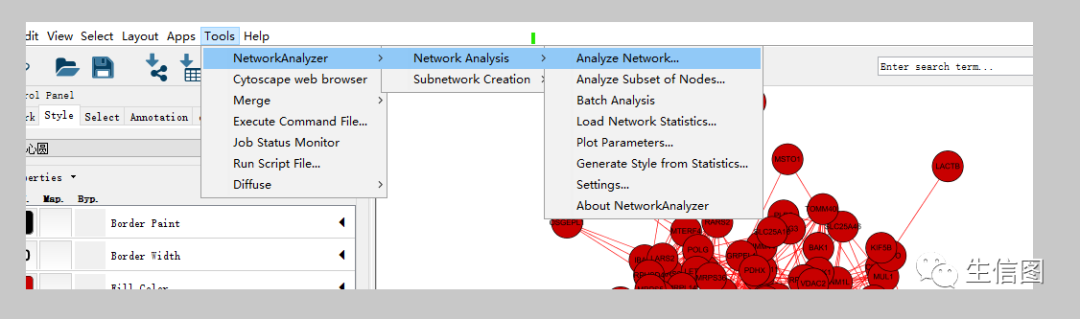
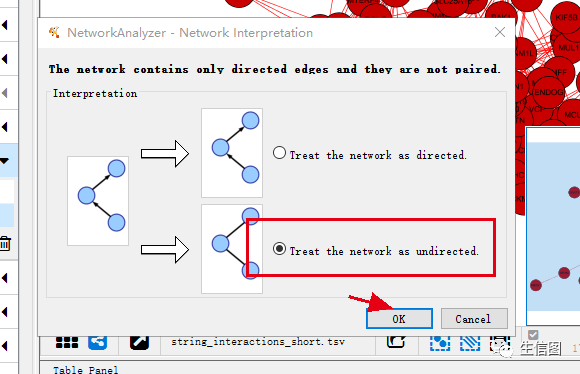
(3)对导入的数据进行网络化分析:在工具栏中选择Tools->Analysis Network->OK。操作界面如下图:
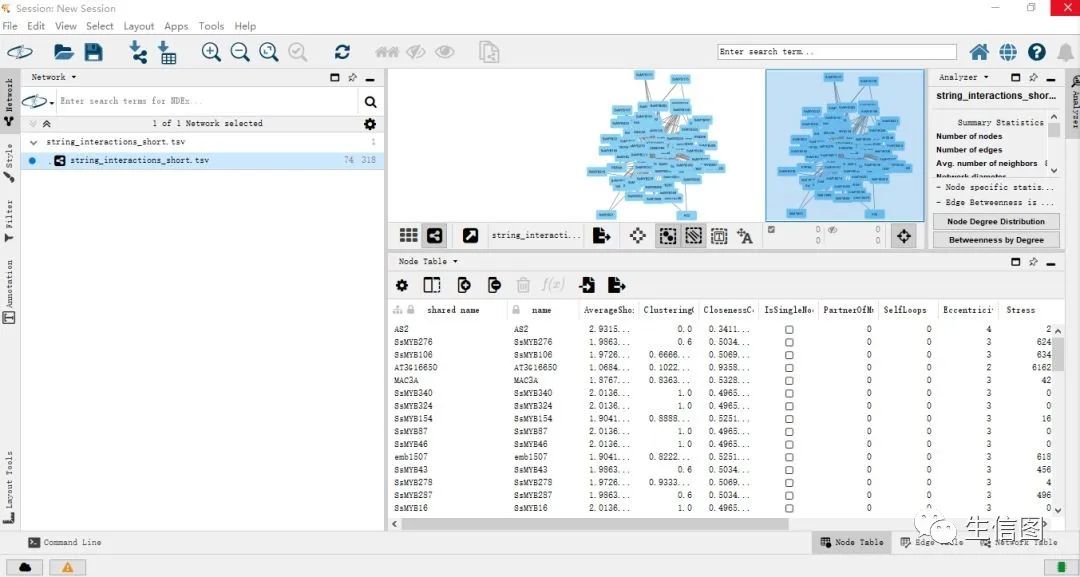
(4)如果没有小伙伴想要的类型可以进行自己构建,比如基因数目较多,可以采用同心圆的展示类型。在工具栏中选择Style->三->Create New Style。操作界面如下图:
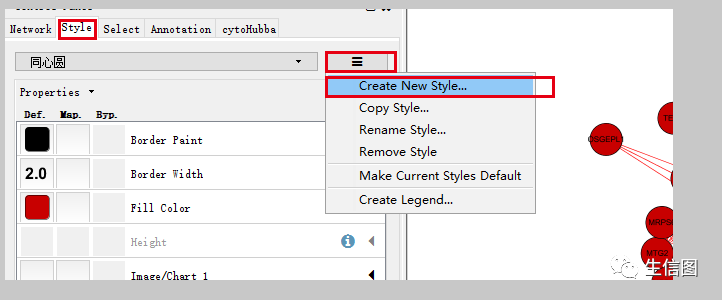
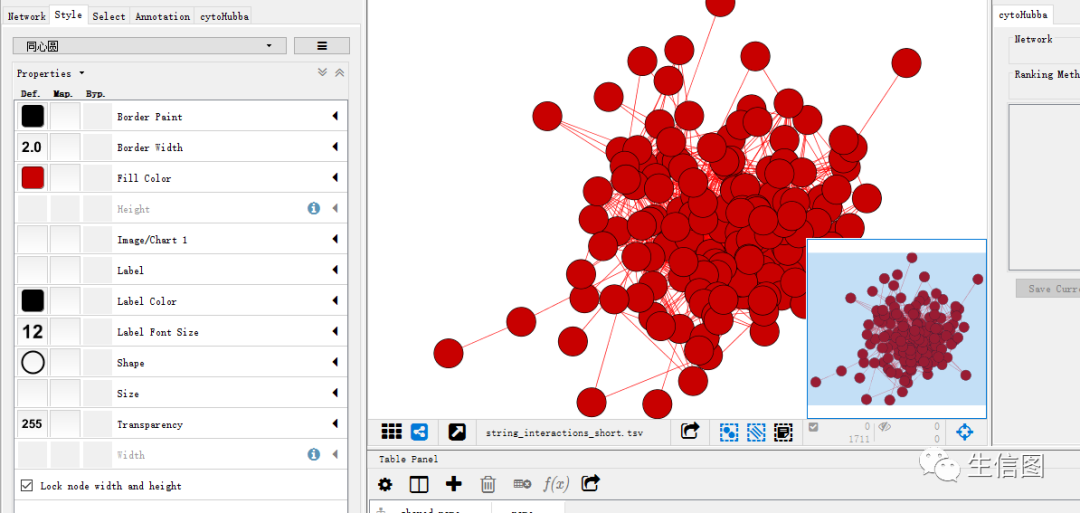
是不是发现没有基因名
(5)在工具栏中选择Style->label-shared name->Passthrough Mapping。操作界面如下图:
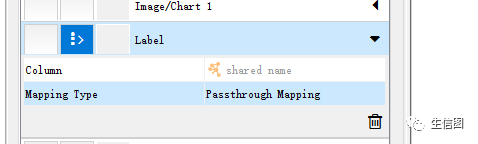
这样名字就显现出来了
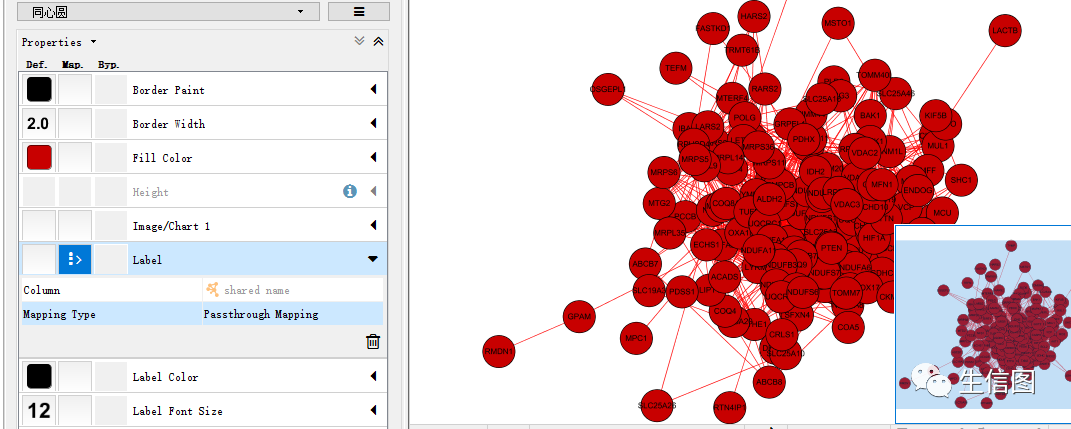
接下来就要进行展示网络的美化
二、美化
对于最初版的网络图,小图相信没有人会觉得好看,也绝对满足不了杂志的要求,那么,我们就动手美化它吧。
其实Cytoscape的可视化界面非常人性化,按照小图下面的批注,大家都点一点,应该很快就能掌握。
在页面左侧选择style(参数选择版块)对网络图进行美化。Cytoscape也自带了美化模板,大家可以依次尝试效果,也可以根据绘图需求在基础模板上进行参数调整。
style板块分为三个美化模块Node、Edge和Network。Node是美化节点,Edge是美化互作关系线、Network是作用于整张图。
(1)更改节点形状和大小。
选择Shape->Ellipse->Apply将节点形状改为圆形。操作界面如下图:
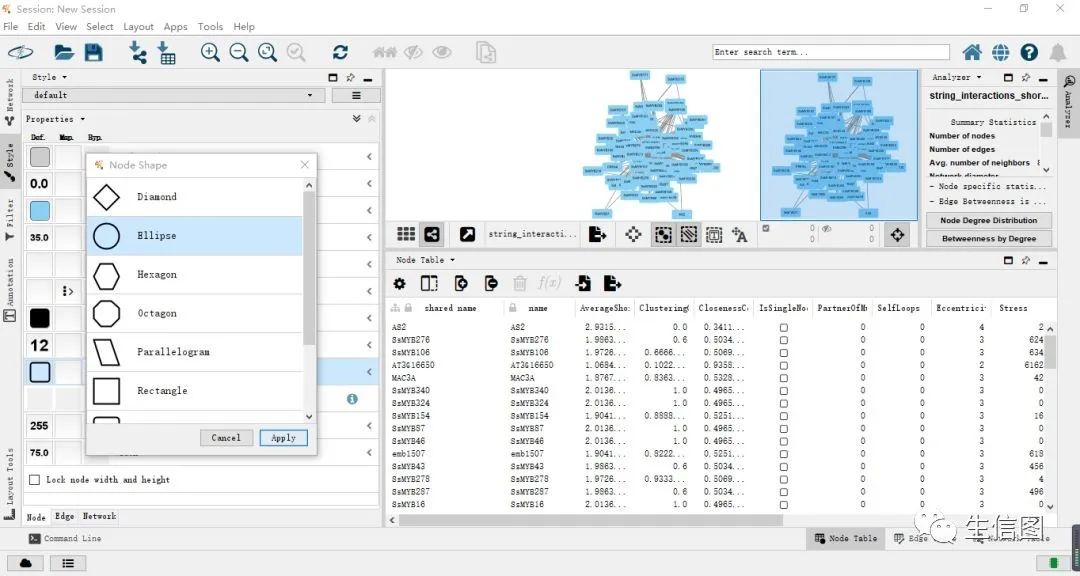
选择Height将节点高度改为75;
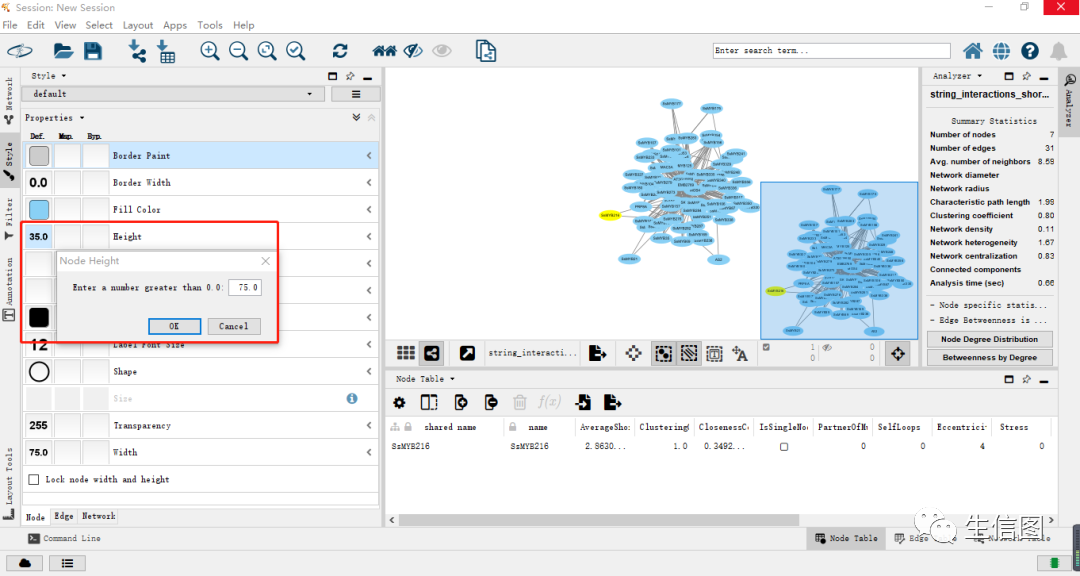
需要注意的是,在每个模块上都有三列,Def.列代表参数为全局参数,Map.列代表参数随互作关系而变化,Byn.列代表参数只作用于所选某一节点或互作关系。
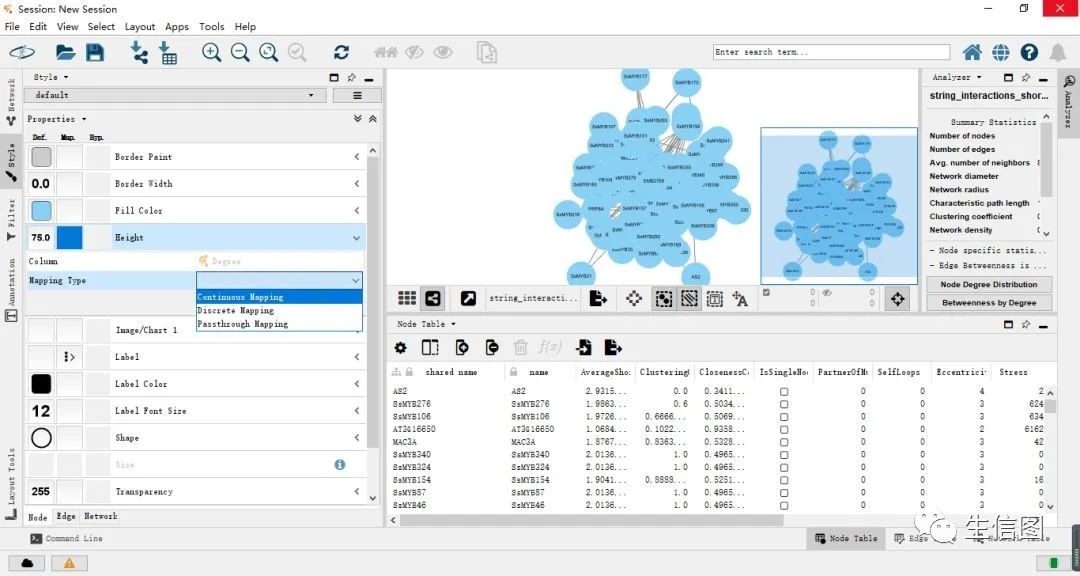
将节点的大小改为连续变量,当一个节点与其它节点的互作关系线越多时,节点越大。选择Map.列的Height->Column->Degree,Mapping Type参数选择Continuous Mapping连续映射。操作界面如下图:
在连续映射中,节点的高度是一个连续变量,为了附和上图节点高度,将变量最大值改为75,最小值改为30。
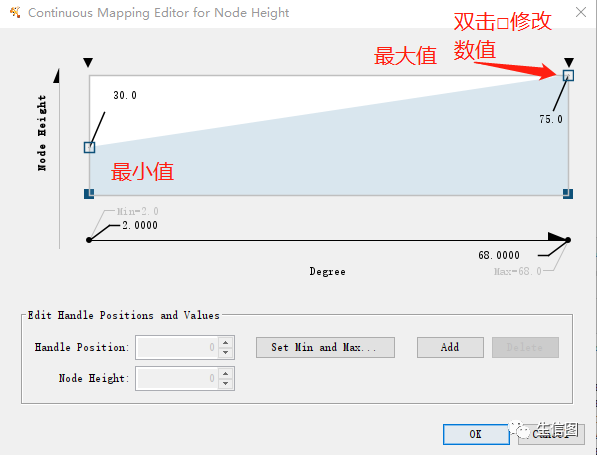
节点的宽度Width如上操作。
(2)更改互作关系线的粗细
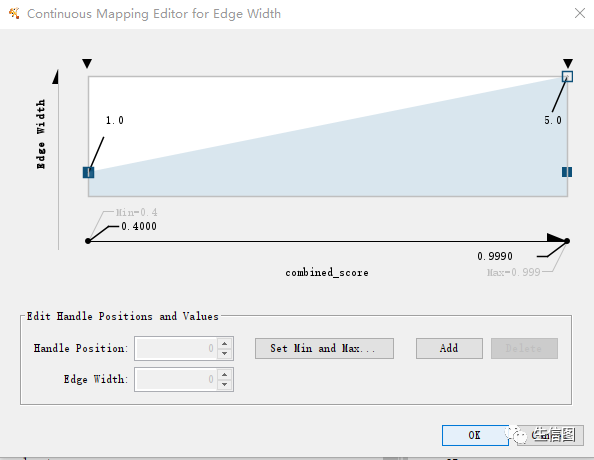
在Edge模块中,选择Map.列的Width->Column->combined_score,combined_score代表节点之间的互作强度。Mapping Type参数选择Continuous Mapping连续映射。在连续映射中,线的高度是一个连续变量,将变量最大值改为5,最小值改为1。操作界面如下图:
(3)数据挖掘
Cytoscape的数据挖掘主要是用到“筛选”工具。网络图常见的筛选方式是利用连通性筛选关键基因,Cytoscape可以直接计算点的连通性(K),点击菜单栏Tools->NetworkAnalyzer->Network analysis->Analyze Network,点击确认,连通性数据就会出现在“点信息页”,列名为“degree”。操作界面如下图:
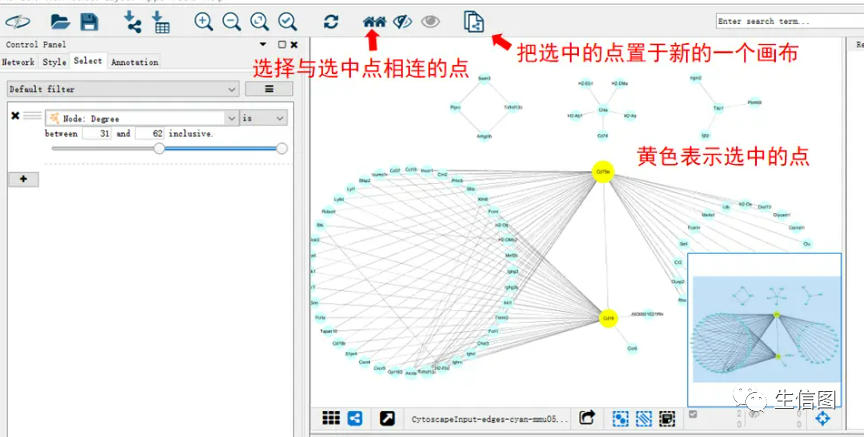
点击筛选模块,新建筛选条件,在下拉菜单中选择“degree”,即可用连通性作为筛选条件,选择满足连通性要求的点。如果图片中点和线特别多,可以用该方法选择连通性较高的点新建画布,只对这些连通性高的基因做图。当然,筛选条件不仅仅是K值,也可以用WGCNA分析里的TOM值,或者基因的表达量等,只要是数值型变量就行,大家尝试一下就明白了。
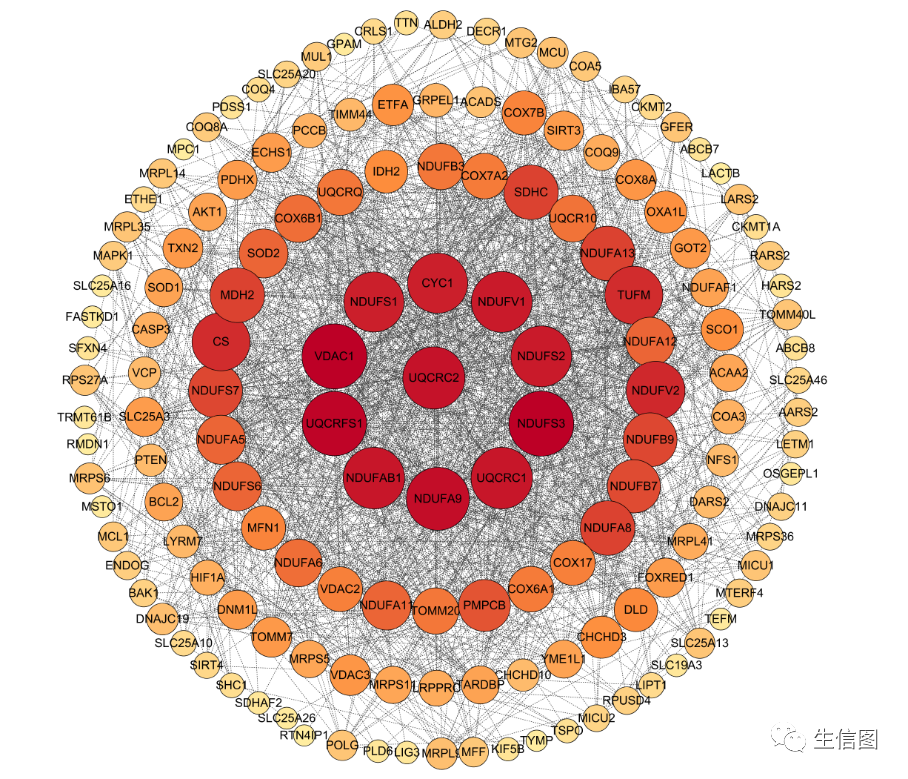
这样漂亮的图就画好了,是不是小伙伴们都给已经跃跃欲试了吧,赶紧动手画起来!
本次分享到这里就结束了,如有疑问或者不清楚的也可以向公众号留言,小图会一一解答,如果大家觉得小图的分享有助于您的科研工作,可以继续关注小图的微信公众号(生信图)和云生信生物信息学平台(http://www.biocloudservice.com/home.html)。
下期会有更多的分享与小伙伴相约!
欢迎使用:云生信平台 ( http://www.biocloudservice.com/home.html)

|
往期推荐 |
|
|
|
|
|
|
👇点击阅读原文进入网址