mlr3中的机器学习(6)——使用mlr3tuning进行超参数调优
点
击蓝字 关注我们
小师妹曾在之前的章节中向大家简单介绍了机器学习中的超参数概念,并向大家教学如何手动调节机器学习中的超参数,但在实际问题中,尤其对于机器学习小白,超参数的确定是非常困难的,所以通过手动的方法往往并不能给出合理的答案,这个时候,需要开发一种基于数据的超参数学习方法,mlr3包中的mlr3tuning就是争对这一问题开发的。
首先,让我们载入需要的R包:(注意,在这里需要下载mlr3learners包,以获取更多的learner方法)
library(mlr3)library(mlr3tuning)library(mlr3learners)
●
一.模型调优
当涉及到使用mlr3包进行超参数调优时,让我们以一个生动的例子来具体展示这个过程。假设我们想要针对一个数据集上的支持向量机(SVM)模型进行超参数调优。这个数据集是用于声纳信号分类的任务,我们将使用mlr3包来优化SVM的性能。
首先,让我们逐步看一下如何设置调优实例,以便更好地了解如何控制调优过程以及如何设计和使用调优器。在我们的示例中,我们将使用SVM来学习声纳任务,同时采用三折交叉验证来优化分类误差度量。为了方便演示,我们将把终止器设置为“无限制”,以便没有时间限制。
首先,我们需要创建一个数据任务对象来加载sonar数据集:
tsk_sonar = tsk("sonar") 接下来,我们定义一个学习器对象,即支持向量机模型,并根据经验设置它的超参数范围:
learner = lrn("classif.svm",cost = to_tune(1e-1, 1e5),gamma = to_tune(1e-1, 1),kernel = "radial",type = "C-classification")
通过ti()创建一个调优实例,这里我们使用三折交叉验证和分类误差度量,并通过terminator = trm(“none”) 设置终止器为“无限制”:
instance = ti(task = tsk_sonar,learner = learner,resampling = rsmp("cv", folds = 3),measures = msr("classif.ce"),terminator = trm("none"))
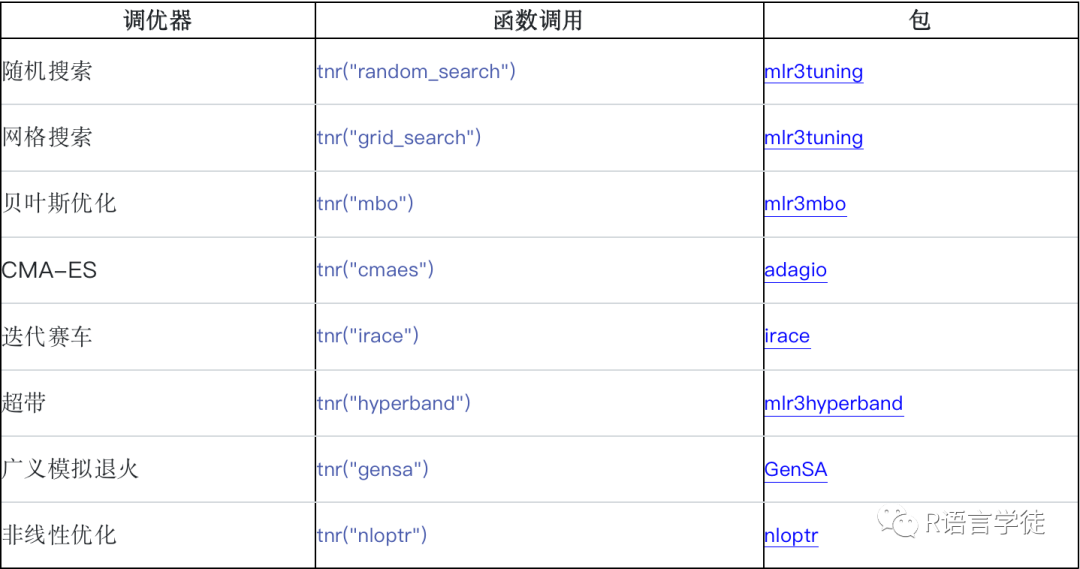
超参数优化的调优器有很多种,我们列出常见的:
选择并设置我们的调优器,我们使用网格搜索调优器,分辨率为5,在实践中,首选更大的分辨率。分辨率是每个超参数要尝试的不同值的数量,这意味着在我们的示例中,调谐器将在指定的上限和下限之间构造一个包含 5 个等间距点配置的 25×4 网格。调谐器将尝试所有配置(以随机顺序),直到评估所有配置或终止器发出预算用尽的信号:
#设置调优器tuner = tnr("grid_search", resolution = 5, batch_size = 10)
最后,我们运行调优过程,让调优器优化我们之前创建的调优实例,优化器返回最佳超参数配置和相应的性能:
#调优tuner$optimize(instance)
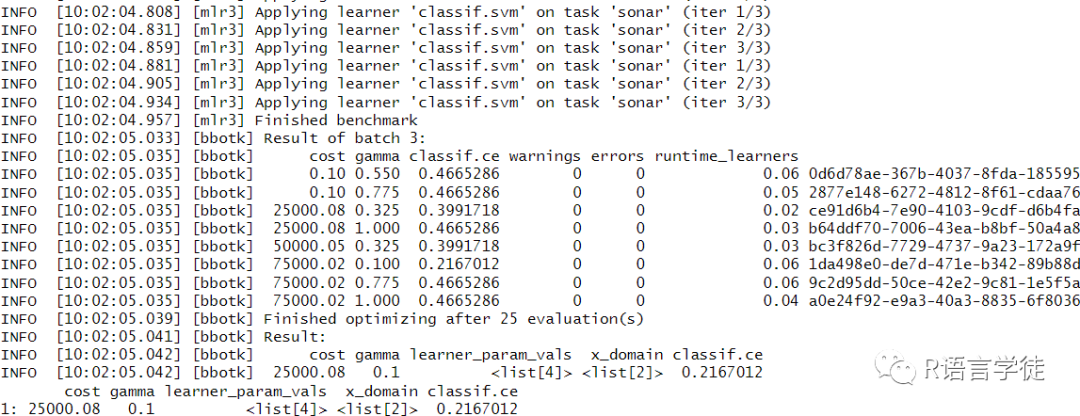
可以在这里看出,最终调优的结果显示应选择超参数cost=25000.09,gamma=0.1。
还可以在超参数调优中使用对数变换,在超参数调优中使用对数变换是一个常见的技巧,主要是为了在搜索超参数空间时更有效地探索可能的值范围。这种技巧在某些情况下可以提供一些优势,尤其是当超参数的取值范围跨越多个数量级时。使用对数变换很简单,只需要在to_tune()中加入输入logscale = TRUE.
#--------、#对数变换learner = lrn("classif.svm",cost = to_tune(1e-5, 1e5, logscale = TRUE),gamma = to_tune(1e-5, 1e5, logscale = TRUE),kernel = "radial",type = "C-classification")instance = ti(task = tsk_sonar,learner = learner,resampling = rsmp("cv", folds = 3),measures = msr("classif.ce"),terminator = trm("none"))tuner$optimize(instance)

●
二.结果分析
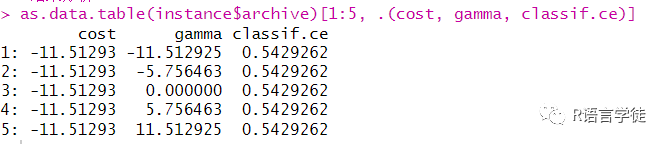
我们使用对数变换后的超参数优化示例进行结果分析,可以通过archive函数调取每次超参数优化的结果,先将该结果转化成data.table格式再进行索引:
#结果分析as.data.table(instance$archive)[1:5, .(cost, gamma, classif.ce)]
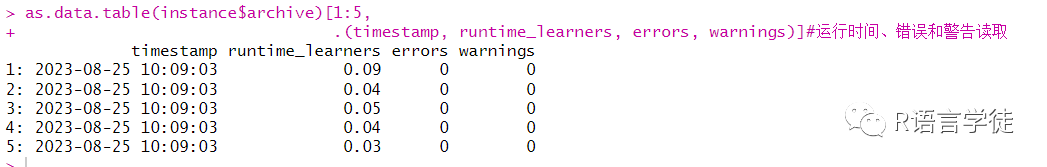
还可以读取超参数优化中的调优时间、警告和错误:
as.data.table(instance$archive)[1:5,.(timestamp, runtime_learners, errors, warnings)]#运行时间、错误和警告读取
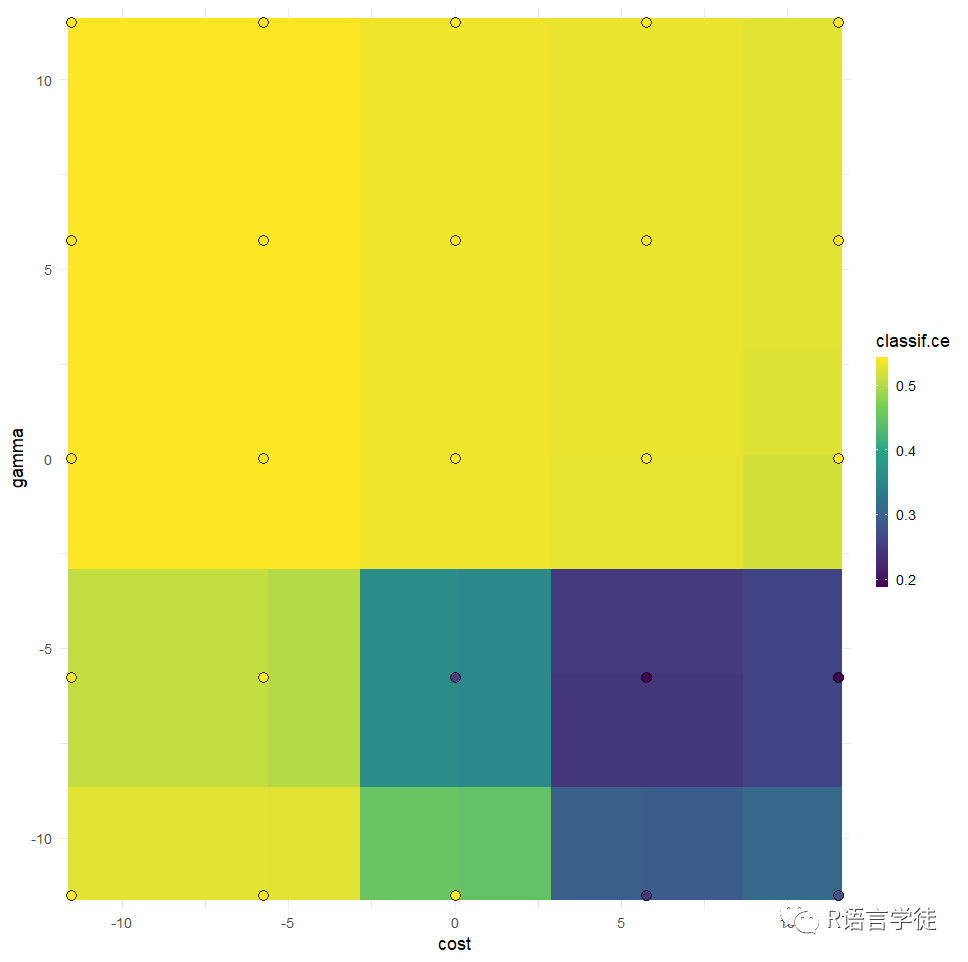
同样可以使用mlr3viz对结果进行可视化,可以通过网格的颜色和横纵坐标来观察不同参数的模型优劣:
#可视化library(mlr3viz)autoplot(instance, type = "surface")
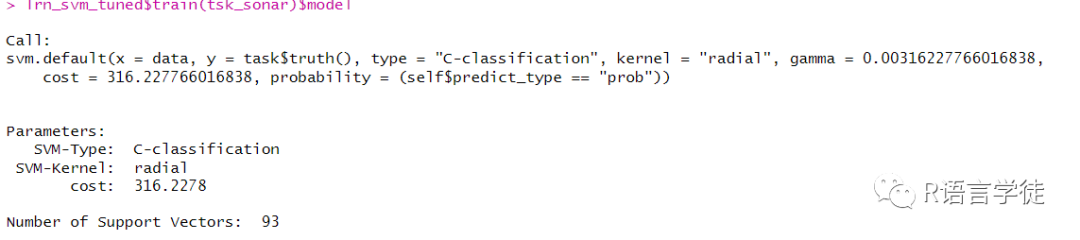
最后,当我们选择了合适的超参数,就可以用其对我们需要的数据进行训练,来训练整个数据的最终模型。为此,我们只需使用相同的底层算法构造一个新的学习器,并将学习器超参数设置为最佳配置:
#训练优化后的模型lrn_svm_tuned = lrn("classif.svm")lrn_svm_tuned$param_set$values = instance$result_learner_param_vals#将优化后的超参数读入我们所使用的模型中lrn_svm_tuned$train(tsk_sonar)$model
●
三.自动调优方法
在上面的调优示例中,小师妹带大家使用ti()函数并选择需要的优化方法进行超参数的调优,而再mlr3tuning中,提供了对新手更为友好的调优方法,小师妹更推荐同学们尝试!
其一是tune()函数,同时,它可以在内部设置相应的重采样方法,更为准确地评估模型:
tnr_grid_search = tnr("grid_search", resolution = 5, batch_size = 5)lrn_svm = lrn("classif.svm",cost = to_tune(1e-5, 1e5, logscale = TRUE),gamma = to_tune(1e-5, 1e5, logscale = TRUE),kernel = "radial",type = "C-classification")rsmp_cv3 = rsmp("cv", folds = 3)msr_ce = msr("classif.ce")instance = tune(tuner = tnr_grid_search,task = tsk_sonar,learner = lrn_svm,resampling = rsmp_cv3,measures = msr_ce)instance$result
还有一个更简单的方法,使用auto_tuner()函数,它可以提前设置,然后再调用数据时,自动为数据提供一个相对最优的超参数,直接使用auto_tuner返回的对象,就可以针对特定数据集分别给出合适的超参数。
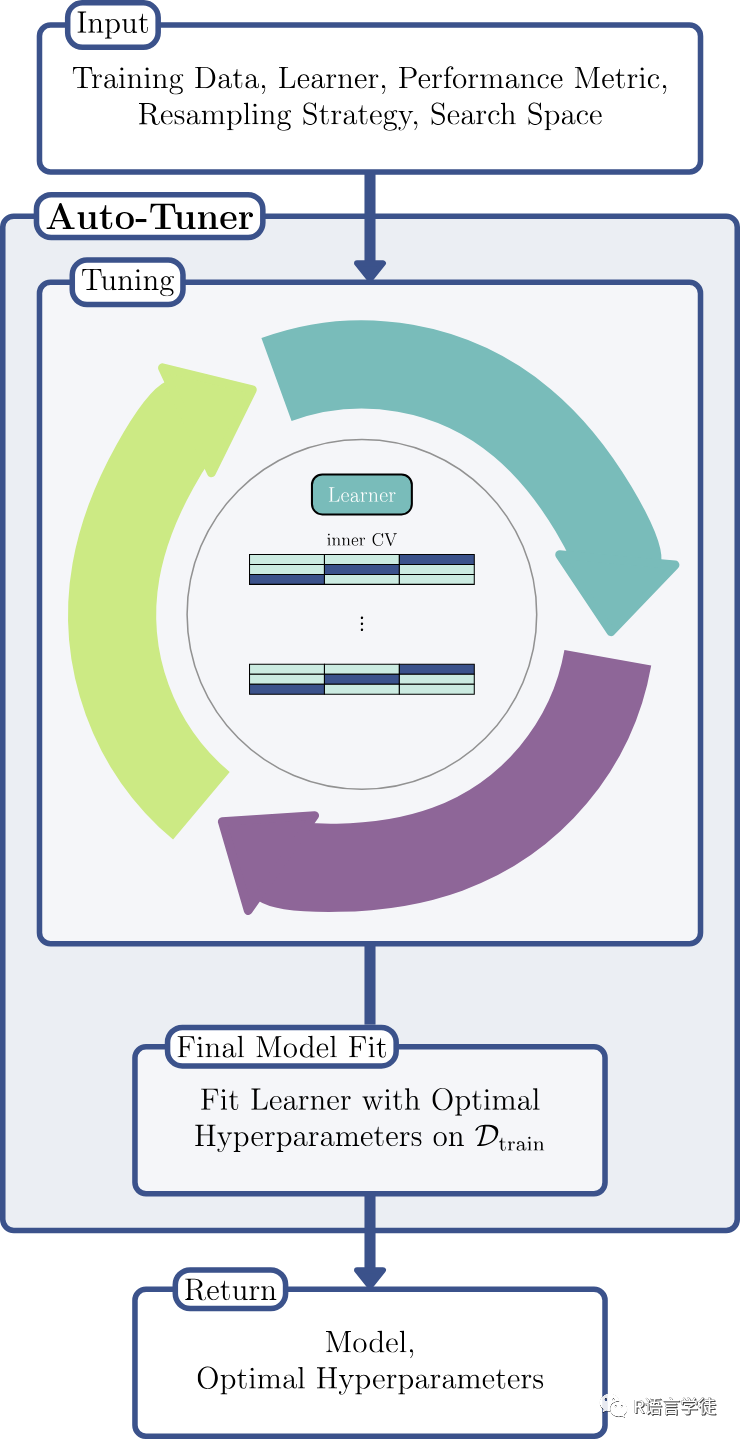
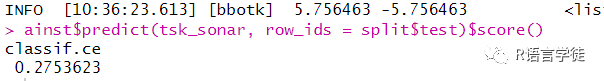
#auto_tunerainst = auto_tuner(tuner = tnr_grid_search,learner = lrn_svm,resampling = rsmp_cv3,measure = msr_ce)ainstsplit = partition(tsk_sonar)ainst$train(tsk_sonar, row_ids = split$train)ainst$predict(tsk_sonar, row_ids = split$test)$score()
●
使用mlr3tuning进行超参数调优的学习就到这里结束啦,小伙伴们是否学会了呢,希望课下大家可以打开R语言一起练习哦。小师妹要和大家说再见咯,一定要自己练习一下哦,同时如果大家想要继续了解更多有关R语言内容可以持续关注小师妹哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html
使用mlr3tuning进行超参数调优的学习就到这里结束啦,小伙伴们是否学会了呢,希望课下大家可以打开R语言一起练习哦。小师妹要和大家说再见咯,一定要自己练习一下哦,同时如果大家想要继续了解更多有关R语言内容可以持续关注小师妹哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html

