20分文章的单细胞热图!!还不跟小果去学习一下如何绘制!
小果最近在看文献的时候,发现一个IF>20!文章中有一张图是有关单细胞的热图,图片中是marker基因的热图外加+Go富集条形图,我们先看一下图片
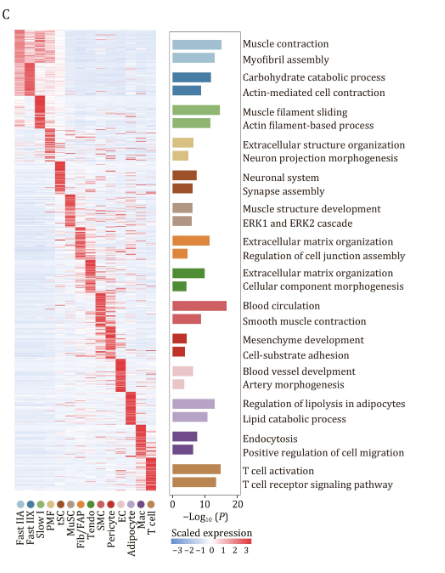
这个图片中左图显示了每种细胞类型按LogFC排名的前50个基因的表达谱。右,每种细胞类型的标记基因的丰富GO术语,这个图可以展示很多的信息,小伙伴学会这样图就可以把自己单细胞数据还有富集分析放进去。那么今天小果就带小伙伴复现一下这张图。

我们使用R包中自带的数据,选择前5名的marker基因去进行热图绘制。
数据来自marker基因数据呢来自scRNAtoolVis。我们先下载一下这个包
公众号后台回复“111″,领取代码,代码编号:231012
#if not install ggunchull#devtools::install_github("sajuukLyu/ggunchull", type = "source")#install.packages('devtools')#devtools::install_github('junjunlab/scRNAtoolVis')
我们先看一下数据
library(scRNAtoolVis)httest <- system.file("extdata", "htdata.RDS", package = "scRNAtoolVis")pbmc <- readRDS(httest)# load markergenemarkergene <- system.file("extdata", "top5pbmc.markers.csv", package = "scRNAtoolVis")markers <- read.table(markergene, sep = ',', header = TRUE)dim(markers)
我们先绘制热图,数据呢其实就是每个细胞亚群中排名前提marker基因并集的表达热图。
library(Seurat)# AverageExpression 计算均值df <- as.data.frame(AverageExpression(object = pbmc)$RNA)df %>%filter(row.names(.) %in% markers$gene) %>%rownames_to_column(var = "gene") %>%right_join(markers) %>%arrange(cluster, avg_log2FC) %>%pivot_longer(cols = `Naive CD4 T`:`Platelet`, names_to = "cluster_name", values_to = "exp") %>%group_by(gene) %>%mutate(exp = as.numeric(scale(exp))) %>%ungroup() -> df
绘制热图:
##热图# 一共9个亚群,这里只展示8个,不含NKcluster_name <- c("Naive CD4 T", "CD14+ Mono", "Memory CD4 T", "B", "CD8 T", "FCGR3A+ Mono", "DC", "Platelet")df %>%filter(cluster_name != "NK", # 过滤掉NK细胞群/没有任何意义cluster != 6) %>%ggplot(aes(x = cluster_name,y = reorder(gene, -cluster),fill = exp)) +geom_tile() +scale_fill_gradient2(low = "blue", mid = "white", high = "red") +scale_x_discrete(limits = cluster_name)

一个简单的热图我们就绘制出来了,我们在来调整一下细节,去掉横纵坐标还有刻度,在修改一下图例的位置。
p1 <- df %>%filter(cluster_name != "NK",cluster != 6) %>%ggplot(aes(x = cluster_name,y = reorder(gene, -cluster),fill = exp)) +geom_tile(color = "grey60") +scale_fill_gradient2(low = "blue", mid = "white", high = "red", breaks = c(-1,0,1,2)) +scale_x_discrete(limits = cluster_name) +theme_void() +theme(legend.position = "left")p1
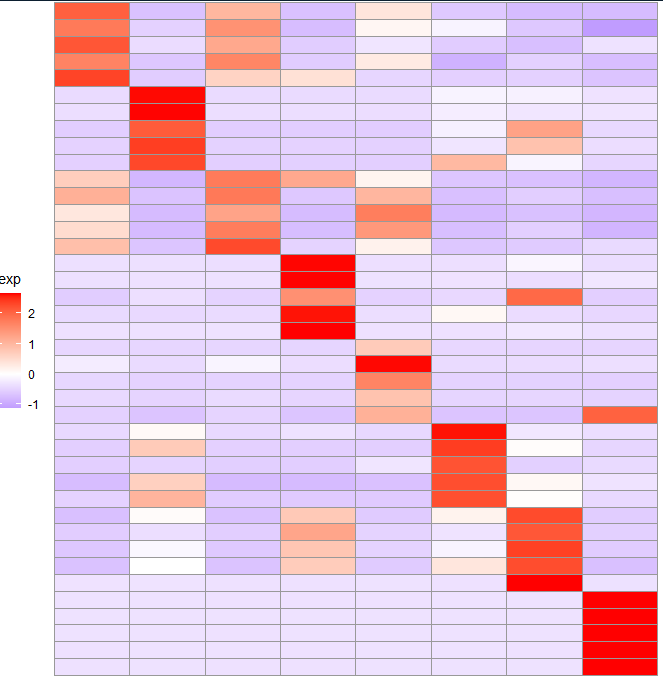
这样一来文献中左边的图大致绘制出来了,我们一点一点的绘制,下面我们绘制点图,
点图作为热图的每一列细胞亚群(名称)注释
#点图colors <- c("#4A9D47", "#F19294", "#E45D61", "#96C3D8", "#5F9BBE", "#F5B375", "#67A59B", "#A5D38F")df2 <- data.frame(x = 0, y = levels(pbmc)[-7], stringsAsFactors = F)df2$y <- factor(df2$y, levels = df2$y)
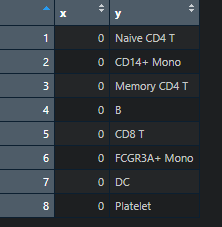
下面绘制点图:
#点图p2 <- df2 %>%ggplot(aes(x = y, y = 1, color = factor(y))) +geom_point(size = 9, show.legend = F) +scale_color_manual(values = colors) +scale_y_continuous(expand = c(0,0)) +theme(legend.position = "none",panel.spacing = unit(0, "lines"),panel.background = element_blank(),panel.border = element_blank(),plot.background = element_blank(),plot.margin = margin(0, 0, 0, 0, "pt"),axis.text.x = element_text(angle = 90,size = 12,hjust = 1,vjust = 0.5,color = "black"),axis.title = element_blank(),axis.ticks = element_blank(),panel.grid = element_blank(),axis.text.y = element_blank())p2
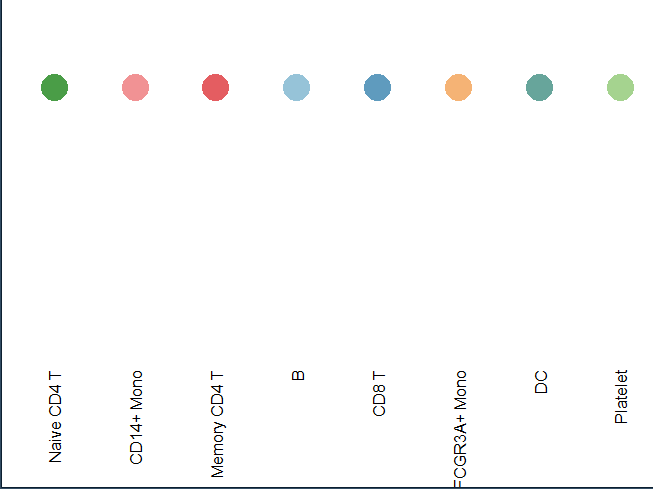
好了点图的绘制我们就绘制完成了,下面我们去绘制条形图,就是Go富集的结果,我们没有找富集结果,小果这里模拟富集结果,小伙伴自己的富集结果放进去就行,就是对你每个亚群的topmarker基因进行富集分析
整理成下面的格式,来看一下,每个亚群展示2个代表性的Goterm
##条形图enrich_res <- "Cluster GO_term Log10(padj)Naive CD4 T GO:00001 -20.00CD14+ Mono GO:00002 -18.00Memory CD4 T GO:00003 -14.00B GO:00004 -12.00CD8 T GO:00005 -19.00FCGR3A+ Mono GO:00006 -17.00DC GO:00008 -18.00Platelet GO:00009 -13.00Naive CD4 T GO:00010 -12.00CD14+ Mono GO:00011 -10.00Memory CD4 T GO:00012 -6.00B GO:00013 -11.00CD8 T GO:00014 -12.00FCGR3A+ Mono GO:00015 -9.00DC GO:00017 -8.00Platelet GO:00018 -8.00"df3 <- read.table(textConnection(enrich_res),sep = "t", header = T,check.names = F)df3$Cluster <- factor(df3$Cluster, levels = levels(pbmc)[-7])
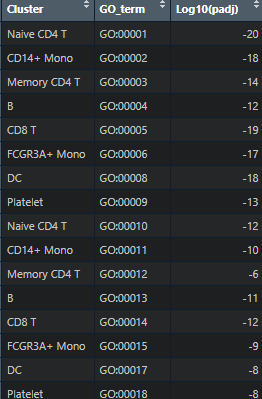
下面我们绘制GO富集条形图
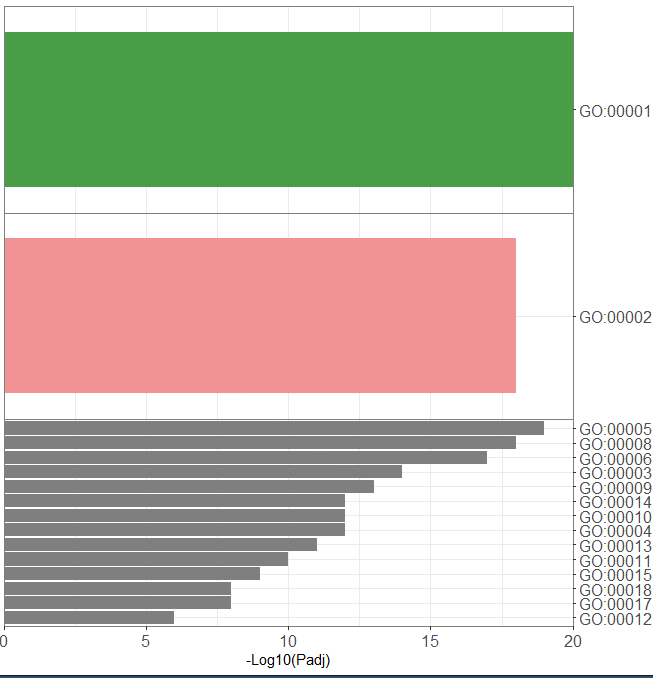
这样一来,我们的三张图基本上就绘制完成了,我们下面就去把图片组合一下
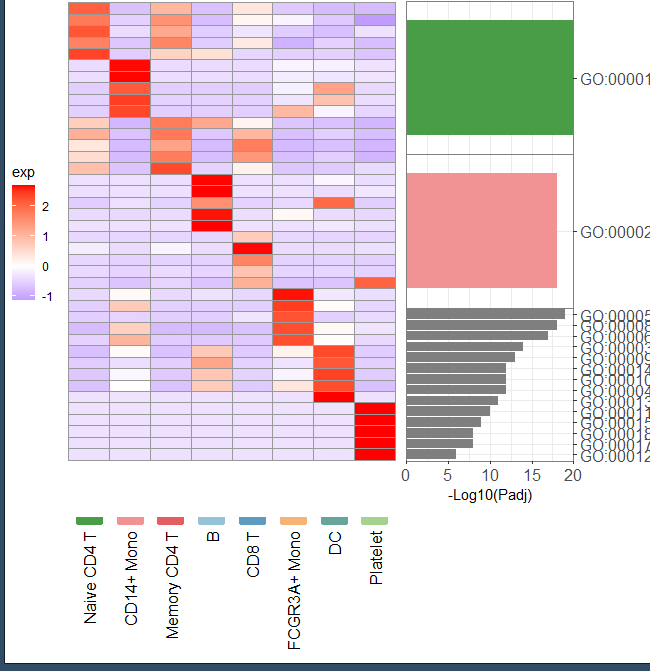
这样一来就大致复现了,不过就是颜色不太对,还有富集的结果是我们自己创建的可能数据形式可能不太像,不过小伙伴要多多理解代码的意义,把自己的数据放进去绘制出SCI文章!快去绘制出自己图片吧!

往期推荐
