DNA生信分析(六):小白必看!五分钟教你序列比对可视化
点击蓝字 关注我们
五分钟教你序列比对可视化
在前面的内容中,小花给大家详细地分享了序列比对的操作过程和原理,相信小伙伴们都是收获满满。不过可能有小伙伴们会有疑问,序列比对虽好,我们怎么可以更直观地感受到它呢?在第一期小花就跟大家介绍过生信个体分析中可能会遇到的各种文件类型,其中在介绍.BAI文件时小花就说过,有一款功能很强大、使用很便捷的可视化软件IGV小花很喜欢用,相信小伙伴们都有印象,已经迫不及待了吧~不要心急,这一次小花就给大家带来详细的IGV使用教程。
什么是可视化?
可视化是指利用图形、图表、地图、动画等视觉元素的方式来表达数据、信息、概念或思想的过程。简单来说,可视化就是将抽象的数据或概念通过视觉手段转化为可以直观理解和感知的形式。
可视化的目的是为了更好地传达和解释信息。通过将数据以可视化形式展示出来,人们可以更容易地理解数据之间的关系、趋势、模式和异常。可视化有助于发现数据中的隐藏信息、进行数据探索和分析、支持决策制定,并且提供更有说服力和沟通效果的方式。
基因组可视化是将基因组和相关生物学信息以图形化的形式呈现出来,以帮助科学家和研究人员更好地理解和分析基因组数据。基因组可视化的目的是通过视觉化的方式展示基因组的结构、特征、变异和功能等信息。
基因组可视化可以用于多个层面的研究,包括基因组结构、基因组注释、基因定位、基因表达、基因调控、遗传变异等。它可以显示基因、染色体、DNA序列、SNP位点、启动子、转录因子结合位点等在基因组上的位置和相互关系。
常见的基因组可视化方法包括:
染色体视图:将染色体的长度按比例绘制为线条,并在上面标识基因、重要区域等。
基因结构视图:显示基因的外显子、内含子、启动子、终止子等结构,并标识功能区域。
序列比对视图:将不同基因组序列进行比对,突出显示相似和差异的区域。
网络图谱:展示基因网络、蛋白质相互作用网络、代谢通路等复杂关系。
热图:用颜色表示基因或基因组的表达水平、甲基化水平等,以反映不同样本或条件下的差异。
三维结构可视化:通过呈现蛋白质和DNA的三维结构,揭示分子之间的相互作用。
什么是IGV?
IGV(Integrative Genomics Viewer)是一款用于可视化和分析基因组数据的软件工具,是一个功能强大的基因组浏览器,可以帮助研究人员可视化和解释复杂的生物学数据。
IGV支持多种基因组数据类型,包括基因组序列、注释信息、基因表达数据、染色质亲和性测序(ChIP-seq)、DNA甲基化测序、RNA测序等。通过IGV,我们可以将这些数据在基因组上进行显式,以便更好地理解基因组结构和功能。
IGV的功能也很丰富,包括基因组导航、区域缩放、搜索功能、变异和SNP分析、基因表达量分析等。我们可以通过IGV直观地查看基因组上的特定区域,并对基因组进行详细的分析和解读。此外,IGV还支持数据的交互操作和导出,方便用户进行数据的进一步分析和共享。
除了这些,IGV还有一个非常大的优点,那就是不管你用何种系统基·本上都可找到对应的安装包,通用性很强,并且操作非常简单,很适合初入生信大门的小伙伴们。在小花学习生信之初,面对密密麻麻的代码和序列难免会觉得有些枯燥,直到使用IGV等可视化软件后才发现了生信的有趣之处,这也是为什么小花在第一期就专门提到要出一期IGV教程的原因。
顺便打个小广告,如果小伙伴们平时在生信分析的操作过程中遇到困难,面对密密麻麻的代码和报错不知所措,欢迎大家使用小花开发的生信工具平台http://www.biocloudservice.com/home.html哦~
小花在这里提醒一下大家,由于IGV是基于Java开发的软件,使用IGV时本地也需要装有Java环境哦~
好啦,相信小伙伴们已经迫不及待地想一睹IGV的芳容啦,那么下面小花就给大家详细地介绍一下IGV的页面和操作流程吧~
IGV页面
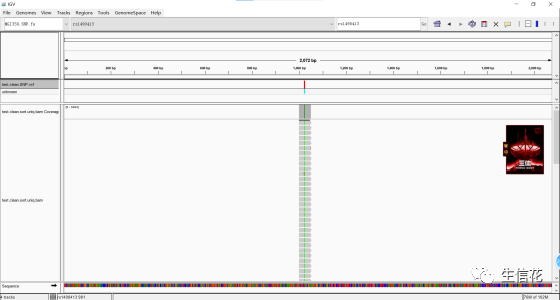
IGV最上面一栏的几个按钮包括 File、Genome、View、Tracks、Regions、Tools、Genomespace 和 Help,以下是每个按钮的作用介绍:
-
File:该菜单提供了加载基因组数据和会话文件、导出图像、保存会话等功能,我们可以使用该按钮来打开和保存项目文件、导入或导出数据以及进行其他与文件相关的操作。
2.Genome:在此菜单中可以选择和加载参考基因组数据。我们可以从本地文件系统或从公共数据库(如UCSC、ENSEMBL)加载基因组,以便在IGV中浏览和分析基因组数据。
3.View:该菜单提供了控制IGV显示设置的选项。我们可以调整视图参数,如缩放级别、颜色设置、轨道高度等,以满足特定的可视化需求。
4.Tracks:该菜单提供了添加、删除、配置和管理数据轨道的选项。我们可以在此菜单中选择要显示的轨道类型,如基因注释、测序 reads、甲基化数据等,并针对每个轨道进行个性化配置。
5.Regions:该菜单提供了定义、管理和导航基因组区域的选项。我们可以输入染色体坐标或基因名来定位到特定的基因组区域,也可以管理已保存的感兴趣区域。
6.Tools:该菜单提供了一些实用的分析和操作工具。例如,我们可以进行序列搜索、比对查找、突变注释等工作。
7.Genomespace:Genomespace 是一个用于数据存储和共享的在线平台。在IGV中使用 Genomespace 功能可以将数据直接传输到 Genomespace,并从 Genomespace 中加载数据到IGV中进行分析。
8.Help:该菜单提供了关于IGV的帮助文档、快捷键参考以及连接到在线支持和社区的选项。小伙伴们可以在这里找到有关IGV功能和操作的详细说明。
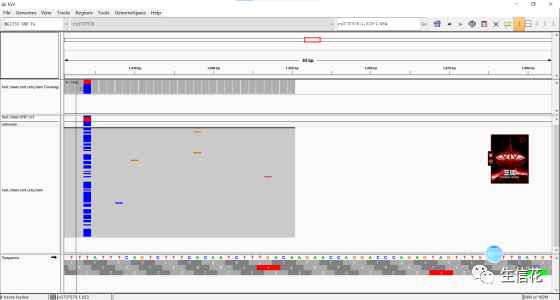
第二行包括三个下拉框和一些按钮,三个下拉框分别是:当前参考基因组、染色体和染色体区位。几个按钮的功能分别是home键、刷新键、标注区域、填充屏幕和调整文本显示习惯。
第三、四、五大行分别是基因组标尺(可以通过缩放改变显示范围),测序数据(track,每一行代表一个样本数据)和基因注释,最下方是当前基因组的注释。
操作流程
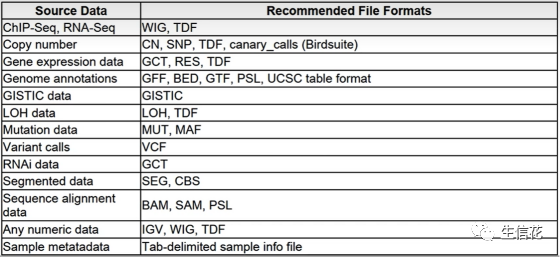
小花这里先把IGV支持的数据格式给大家展示出来:
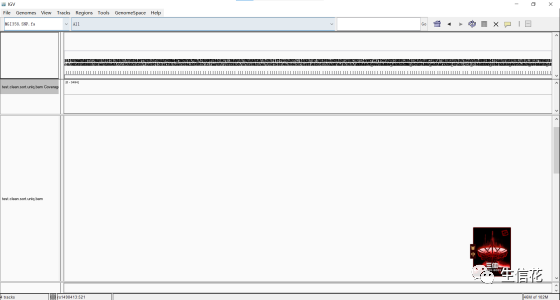
首先通过左上角的File按钮导入需要可视化的.bam文件,通过file下面的第一个下拉框导入IGV中内置的参考基因组文件,若需要导入本地文件作为参考基因组,则通过左上角第二个按钮Genome导入(一般为.fa文件),导入成功时会生成同名的.fai文件。
因为一般参考基因组只是DNA的碱基序列,若想了解编码情况,还需要导入注释文件,依然从File文件中导入。
导入成功后,在第二行第二个下拉框中可以选择要查看的具体染色体,第三个下拉框输入要查看的染色体具体位置或基因名称,即可展示展示出所选区域reads的覆盖情况。在不同基因间输入空格,可以同时查看多个基因。如果你选择的区间过大,在reads覆盖区会出现Zoom in to see coverage,这时点击放大按钮即可展示覆盖情况。
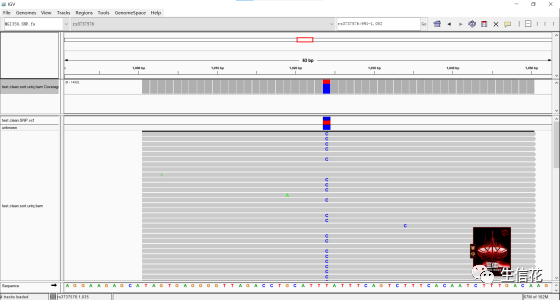
此时第三行显示的是碱基数,红框表示的是目前选中的染色体区域。拖动红框或中心页面都可以拉动预览区域。最下面的sequence显示的是碱基顺序,箭头代表现时方向为5‘端到3’端,点击箭头可查看另一条链的顺序。单击任意核苷酸会出现三行氨基酸序列,展示三种不同起始位点的核苷酸翻译结果,绿色的M代表起始密码子,红色的*代表终止密码子。
以上就是IGV使用的简单教程啦,是不是很简单呢?其实IGV还有更多强大的功能,感兴趣的小伙伴们可以自行上网去搜索学习哦~如果小伙伴们平时在生信分析的操作过程中遇到困难,欢迎大家使用小花开发的生信工具平台http://www.biocloudservice.com/home.html哦。本次的分享就到这里啦,我们下次再见~



长按识别二维码关注我们哟

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

