预后分析不会做?选Lasso-Cox准没错
点击蓝字 关注我们
大家好啊,我是大海哥,我们都知道临床预后评估一直是医学研究中的重要议题,对于患者的治疗选择和疾病管理起着至关重要的作用。结合临床展开分析也一直是生信分析的一大热点。那么选择什么方法来对临床信息做预后分析呢?今天大海哥就推荐一个曾引起医学界广泛关注的Lasso-Cox回归分析。了解Lasso-Cox之前,我们需要先分别了解一下Lasso回归和Cox比例风险模型。
Lasso(Least Absolute Shrinkage and Selection Operator)回归是一种线性回归的改进算法,它在普通线性回归的基础上添加了一个惩罚项。Lasso回归的目标是最小化损失函数和L1范数的组合。L1范数是指参数向量中各个参数绝对值之和。这个额外的L1惩罚使得Lasso回归能够将一些不相关的特征的系数压缩到零,从而实现特征选择的功能。这对于高维数据集或者特征较多的问题非常有用。而Cox是什么呢?Cox比例风险模型(Cox Proportional Hazards Model)是用于生存分析的一种半参数模型。它考虑了时间到事件发生的比例风险,即某个时间点上事件发生的概率与其他时间点相比的相对风险。Cox模型不需要假设风险比在时间上是恒定的,因此可以适用于各种生存数据的分析。
因此结合Cox回归模型后,其可以用于同时进行特征选择和生存预测,这使得Lasso-Cox回归在生存分析中具有独特优势,能够探索影响患者生存率的关键因素,为个体化治疗和预后评估提供新的视角和策略。
今天大海哥将通过一个实例给小伙伴们演示其在预测患者生存率、发现潜在生存相关因素等方面的强大潜力。首先分析之前我们需要准备一个至少包含以下列:SurvivalTime(生存时间)、Status(生存状态,1表示死亡,0表示存活)、其他临床特征列作为预测变量的临床数据。
#大海哥图个方便,随机生成一个临床数据^-^clinical_data <- data.frame(SurvivalTime = sample(100:5000, 500, replace = T),Status=sample(0:1, 500, replace = T),edu=sample(1:4, 500, replace = T),BMI=rnorm(500, mean = 22, sd = 3),Age=sample(10:80, 500,replace = T))#看一下数据,一共有500行

#然后就是加载所需要的库library(survival)library(glmnet)#然后需要做一些数据预处理,确保预测变量为数值型,这一步看具体情况哦!大海哥就不需要用了,小伙伴们需要的可以直接使用。#clinical_data$Age <- as.numeric(clinical_data$Age)# clinical_data$BMI <- as.numeric(clinical_data$BMI)#生存时间和状态需要转换为double型clinical_data$SurvivalTime<-as.double(clinical_data$SurvivalTime)clinical_data$Status <- as.double(clinical_data$Status)#设置因变量,生存时间和生存状态信息y<-data.matrix(Surv(time=clinical_data$SurvivalTime,event= clinical_data$Status))#关键的地方到了,假设我们现在要从50个免疫正相关的基因中筛选具有预后效能的基因,那么这些基因在样本中是高度共表达的,也就是说这些基因存在共线性关系,这种情况正是最适合使用Lasso回归#表达谱数据这里我们使用exp来替代,大家自行导入哦!注意不可以有缺失值!!!大海哥就随机生成一个具有线性关系的数据#看看exp数据吧
#这里需要注意,行为基因,列为样本哦!# 设置alpha参数为1,即LASSO回归,如果为0就是岭回归,为0-1之间就是弹性网络alpha_value <- 1# 在数据集上进行Lasso-Cox回归lasso_cox_fit <- glmnet(x=exp, y,family=”cox”, alpha = alpha_value)

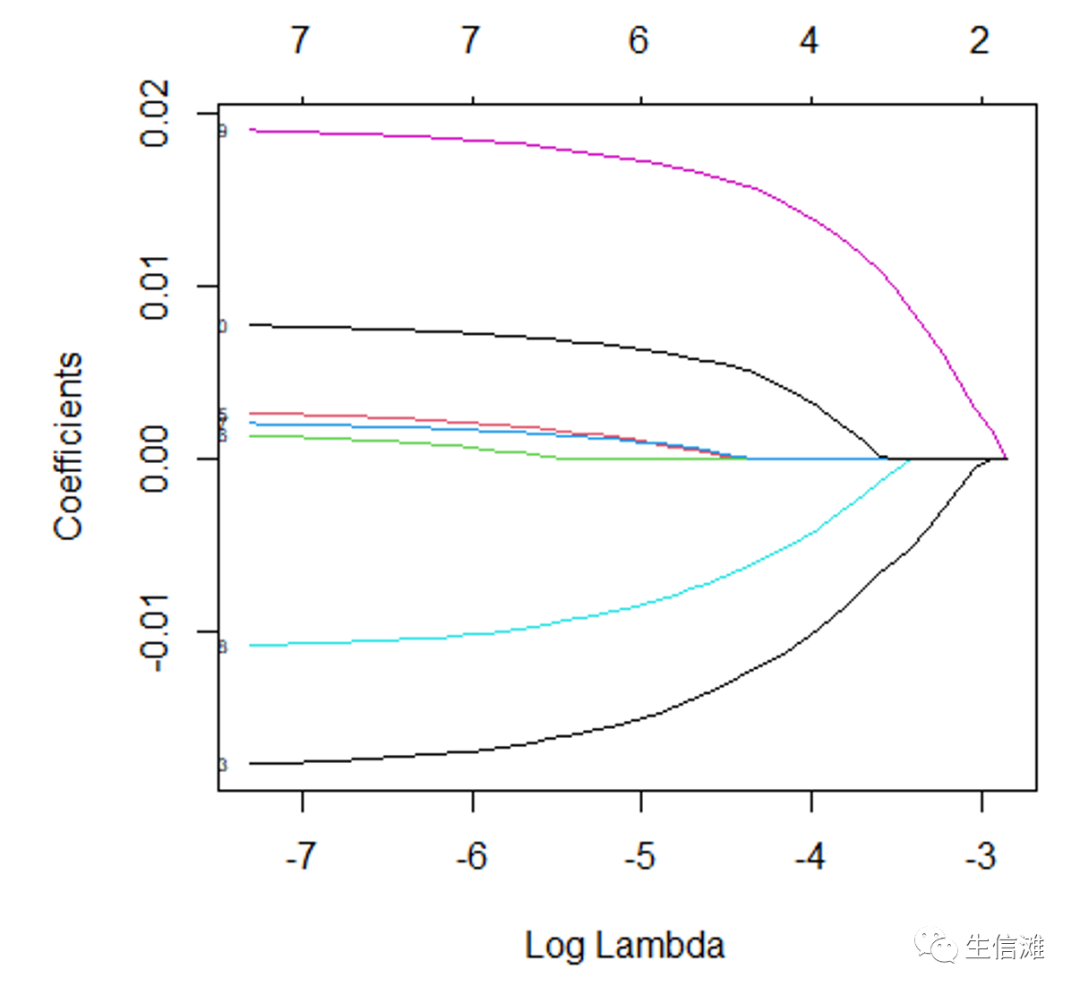
#看看不同基因随着lambda值得变化而变化的趋势plot(lasso_cox_fit,xvar="lambda",label=TRUE)
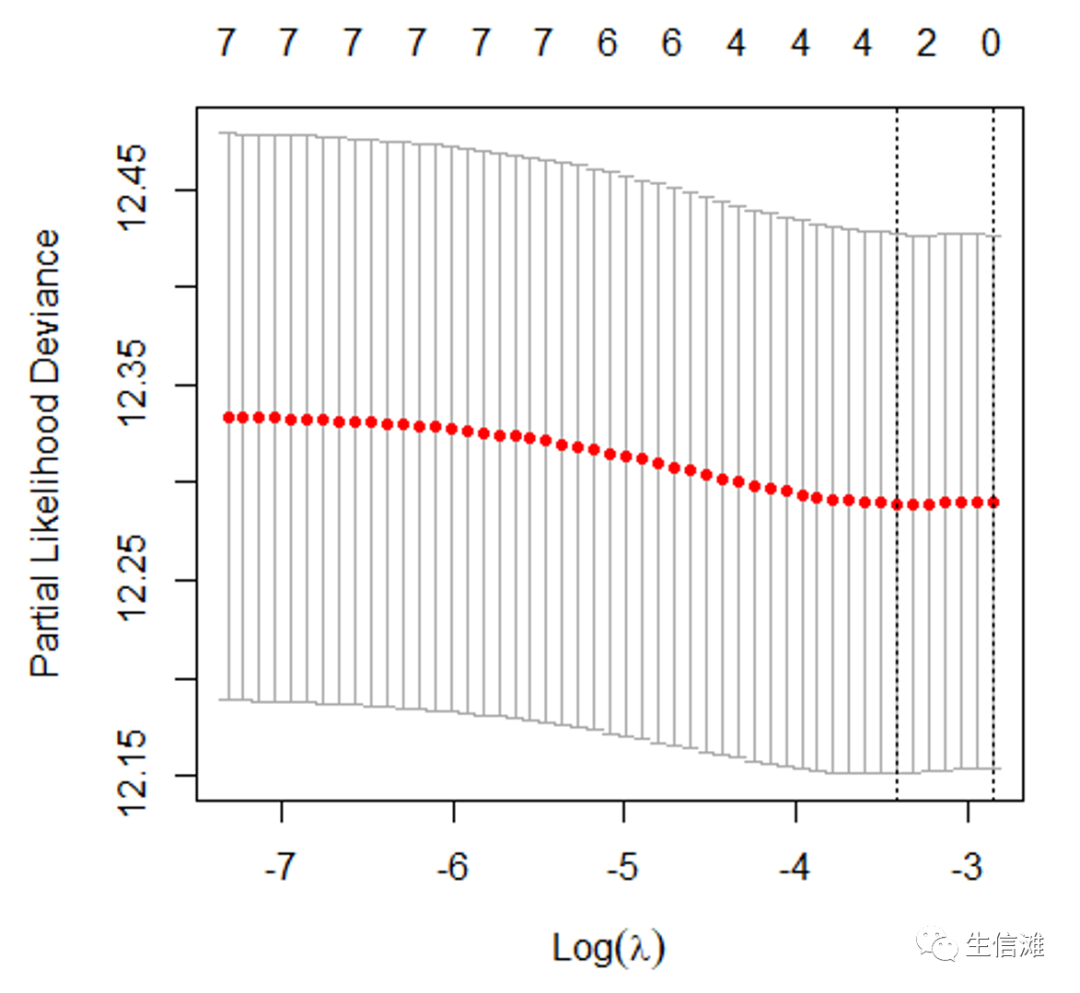
# 执行交叉验证(这一步一般需要稍微等一会会哦!)final_lasso_cox_fit <- cv.glmnet(x=exp, y, family=’cox’,type.measure=’deviance’)# 输出结果plot(final_lasso_cox_fit,label=T)# 可以根据系数的大小来判断哪些基因对预后有显著影响
#现在就可以筛选基因啦!coefficient <- coef(lasso_fit, s = lasso_fit$lambda.min)Active.Index<- which(as.numeric(coefficient)!= 0)active.coefficients <- as.numeric(coefficient)[Active.Index]sig_gene<-rownames(coefficient)[Active.Index]#输出关键基因名(大海哥就不输出了,因为是随机生成的数据)sig_gene
#整体的分析就结束啦!很多小伙伴肯定就会问,为什么要使用这种方法来筛选呢?其实是因为Lasso-Cox回归拥有着一些其他方法没有的优点, 首先asso可以自动选择最重要的特征,将不相关的特征的系数缩减为0,从而降低了模型复杂性,同时当输入特征之间存在多重共线性时,Lasso可以有效地处理,并避免估计过高的系数。其次Lasso-Cox通过使用Cox比例风险模型,Lasso-Cox可以处理时间相关的生存数据,避免了假设风险比是常数的限制。正是因为这些优势,使得Lasso-Cox在处理生存数据时更加全面和有效。
那么在分析过程中需要注意什么呢?
这里大海哥需要提醒大家,Lasso-Cox方法在使用时需要进行调参,例如选择合适的L1惩罚参数。此外,它也有一些限制,比如对于较小的样本量和严重的共线性可能表现不佳。因此,在应用Lasso-Cox方法时,仍然需要仔细考虑数据的特点和模型的调整。所以有些地方需要自己好好琢磨一二哦!
点击“阅读原文”进入网址