「付费」【代码分享-15】8+肿瘤生信文章的宠儿-CMAP肿瘤药物预测分析

小果知识大讲堂又来啦!今天小果为大家带来的分享内容为利用CMAP数据库进行肿瘤药物预测,如何进行相关肿瘤药物预测?不慌!小果来为小伙伴们进行解答,CMAP分析主要包含以下三个部分:
一是疾病的分子特征(disease signature):以肿瘤为例,这个signature可以通过常规的癌与癌旁的差异分析得到。
二是药物的分子特征(druf signature):这部分数据可以来自大家熟悉CMap数据库(包括了1309种药物在5个细胞系中的药物处理后的表达特征)。
三是特征匹配算法(signature matching methods):大家常用的网页版的CMap通常使用基于Kolmogorov-Smirnov (KS)的方法来计算CMap score。
通过这三个部分就可以完成相关肿瘤的药物预测,这就是小果今天带来的分享内容,有需要的小伙伴可以跟着小果开始今天的实操学习。
1.准备需要的R包
#安装需要的R包install.packages("stringr")install.packages("ggrepel")install.packages("ggplot2")install.packages("ggthemes")install.packages("tidyverse")install.packages("dplyr")#加载需要的R包library(parallel)library(dplyr)library(stringr)library(tidyverse)library(tibble)library(clusterProfiler)library(ggplot2)library(ggrepel)library(ggthemes)library(gridExtra)
2.数据读取与处理
#LIHC_TPM.txt,基因表达矩阵文件,第一列为基因名,其他列为样本名
data <- read.table(“LIHC_TPM.txt”, sep=”t”, check.names=F, header=T, row.names=1)

# 得到癌与癌旁比较的logfctumor <- data[, str_detect(colnames(data), "-01")]normal <- data[, str_detect(colnames(data), "-11")]dis_sig <- data.frame(id=rownames(data),fc=log2(rowMeans(tumor)/rowMeans(normal)))dis_sig <- dis_sig[dis_sig$fc != "Inf" & dis_sig$fc != "-Inf",]dis_sig <- na.omit(dis_sig)dis_sig <- rbind(top_n(dis_sig, 150, fc), top_n(dis_sig, -150, fc))# 将logfc转成二分类变量(logfc>0的基因用1表示,logfc小于0的基因用-1表示)dis_sig$fc[dis_sig$fc>0] <- 1; dis_sig$fc[dis_sig$fc<0] <- -1rownames(dis_sig) <- NULL# 保存结果write.table(dis_sig, "dis_sig.csv", sep=",", quote=F, row.names=F, col.names=T)# dis_sig只要整理成以下格式都可以在后面的分析中使用head(dis_sig)
5.结果图片
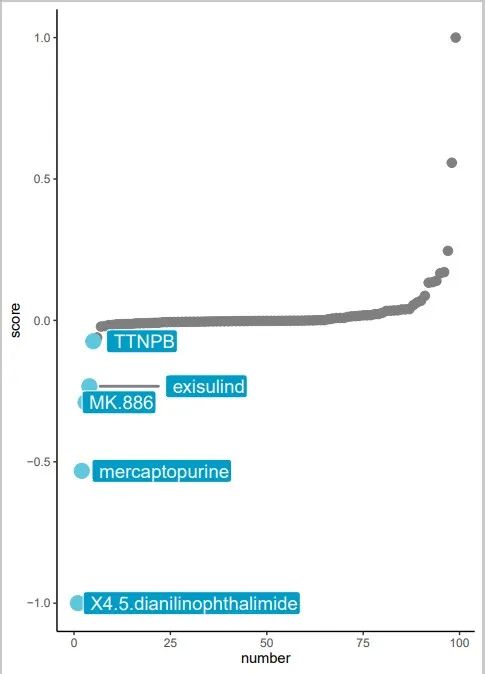
5.1 CMAP_XSum.pdf
该结果图片为绘制的CMAP score散点图,突出显示药物top5的药物,标出药物名;这里我们得到的分数越低,这个药物越有可能逆转疾病的分子特征,理论上更有可能具有治疗该疾病的能力。该演示数据结果提示 X4.5.dianilinophthalimide 是最有可能治疗肝癌的药物。
5.2 dis_sig.csv
该结果文件为通过差异分析得到疾病分子特征,第一列为基因名,第二列为分类特征,是camp分析最简单的一种应用方式,大家也可以开发更多的使用方式(如通过比较转移与非转移的肿瘤样本得到与转移相关的分子特征,基于这个分子特征进行cmap分析就有可能找到潜在对抗转移的药物。

往期推荐
