直击热点!生信文章必备之LASSO,randomForest和xgboost三种机器学习算法进行特征基因筛选
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

小果今天给大家带来的分享绝对是生信热点分析内容,基于三种机器学习算法进行生存资料特征基因筛选,最终将三种算法获得的交叉基因作为我们的特征基因。
接下来跟着小果开启今天的学习之旅吧!关注小果回复111领取相关文件哟!
1. 三种机器学习算法进行特征筛选
小伙伴们是不是在想到底是那三种机器学习算法?不急!小果来慢慢告诉大家,今天小果将利用LASSO回归,随机森林和xgboost三种算法进行生存资料特征基因筛选,这三种机器学习的算法原理小果在这里不做过多的介绍,感兴趣的小伙伴们可以自行查询学习,今天小果将通过实操,让大家轻松掌握如何利用这三种算法进行特征基因筛选,其实分析非常简单,只需要输入带有生存信息的表达矩阵文件,就可以很轻松的完成该分析,对小白也很适用,话不多说,马上跟着小果开始今天的实操吧!
2. 准备需要的R包
#安装需要的R包install.packages("ggplot2")install.packages("tidyverse")install.packages("ggvenn")install.packages("glmnet")install.packages("survival")install.packages("xgboost")install.packages("randomForestSRC")#加载需要的R包library(randomForestSRC)library(tidyverse)library(ggplot2)library(ggvenn)library(glmnet)library(survival)library(xgboost)
3. 数据准备
exp_surv.txt
#生存信息加基因表达矩阵文件,行名为样本信息,第一列为OS.time(生存时间),第二列为OS(生存状态),其他列为基因。

4 . LASSO回归分析
#读取生存信息的基因表达矩阵train<-read.table("exp_surv.txt",header=T,sep="t")#提取基因表达矩阵x1 <- as.matrix(train[, 3:ncol(train)])x2 <- as.matrix(Surv(train$OS.time, train$OS))cvfit = cv.glmnet(x1, x2,nfold=10,family = "cox")#绘制lamba图pdf("lambda.pdf",width = 10,height = 8)plot(cvfit)dev.off()#此处使用`lambda min`cvfit$lambda.min #最佳lambda值

fit <- glmnet(x1, x2,family = "cox")#用包自带的函数绘制lasso回归图pdf("lasso.pdf",width = 10,height = 8)plot(fit)dev.off()#最小值一个标准误时各变量的系数值coef.min = coef(cvfit, s = "lambda.min")#非零系数active.min = which(coef.min != 0)#非零变量,基因名geneids <- rownames(coef.min)[active.min]#提取选中的基因对应的coefficient,回归系数index.min = coef.min[active.min]#按列合并两个变量combine <- cbind(geneids, index.min)#将数据转换为数据框lasso.result<-as.data.frame(combine)#将index.min数据类型改为数值型。lasso.result$index.min<-as.numeric(lasso.result$index.min)

#通过LASSO回归筛选的特征基因文件,第一列表示基因名,第二列表示coefficient。
5.随机森林分析
fit <- rfsrc(Surv(OS.time,OS)~., data = train,ntree = 1000, nodesize = 14,splitrule = 'logrank',importance = T,proximity = T,forest = T)#绘制OBB图和VIP图pdf("forest.pdf")plot(fit)dev.off()#筛选特征基因rftop<-var.select(fit)#将数据类型转化为数据框rftop2<-data.frame(Feature=rftop$topvars,vimp=rftop$varselect[rftop$topvars,2])

注:通过随机森林算法筛选的特征基因,第一列表示基因名,第二列为VIP值。
6.xgboost算法
x_label<-ifelse(train$OS==1,train$OS.time,-train$OS.time)x_train<-as.matrix(train[,c(3:ncol(train))])x_val<-xgb.DMatrix(x_train,label=x_label)xgb_param<-list("objective"="survival:cox","eval_metric"="cox-nloglik")xgb.model<-xgb.train(params=xgb_param,data=x_val,nrounds=100,eta=0.1,watchlist=list(val2=x_val))e<-data.frame(xgb.model$evaluation_log)ggplot(aes(x=iter,y=val2_cox_nloglik),data=e)+geom_point(color="pink",size=3)+geom_line()+theme_bw()ggsave("train_error.pdf")#筛选VIP值排名前十的基因。imp<-xgb.importance(feature_names=colnames(x_train),model=xgb.model)top_10<-imp[1:10,1:2]

注:通过xgboost算法筛选的VIP值排前十的基因,第一列表示基因名,第二列表示VIP值。
绘制venn图筛选三种算法获得的交叉基因
gene<-list(LASSO=geneids,RF=rftop2$Feature,Xgboost=top_10$Feature)ggvenn(gene,fill_color=c("#0073C2FF","#EFC000FF",”#CD534CFF”),stroke_size=0.5,set_name_size=4)ggsave("venn.pdf")
7.结果文件
1. lambda.pdf
该结果图片为LASSO回归lambda图。
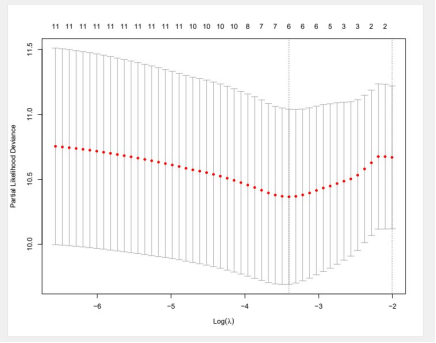
2. lasso.pdf
该结果图片LASSO回归为10折交叉验证图。

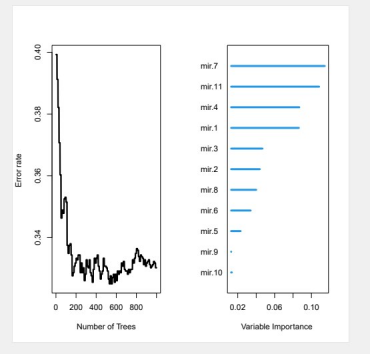
3. forest.pdf
该结果图片为随机森林OBB图和VIP图。
4. train_error.pdf
该结果图片为xgboost算法cox风险比例回归的负偏对数随着迭代次数的变化。

5. venn.pdf
该结果图片为三种算法特征基因的venn图,将交叉基因筛选为候选基因。
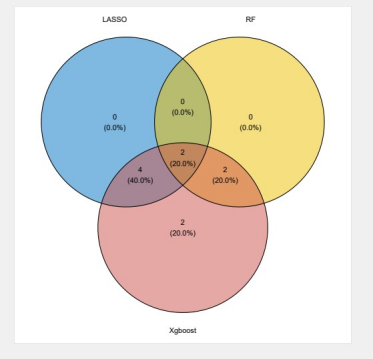
最终小果顺利完成了利用 lassso回归,随机森林和xgboost三种机器学习算法进行了特征基因筛选。机器学习相关其他分析内容欢迎尝试本公司新开发的云平台生物信息分析小工具,零代码完成分析,云平台网址:http://www.biocloudservice.com/home.html。

点击“阅读原文”立刻拥有
↓↓↓