三分钟带你快速学习在R语言中数据转换和分析的保姆级教程
点击蓝字,关注我们
在R中,数据转换(transformation)是一种广泛应用于生物学数据处理与分析的重要技术。生物学领域涉及各种实验和观测数据,这些数据可能具有复杂的结构和分布。数据转换能够有效地改变数据的表示形式,使其更易于理解、解释和建模。在进行生物学数据处理时,数据转换是一个不可或缺的步骤,有助于消除噪音、纠正偏差、增强模式的检测和提高数据的可解释性。
数据转换可以包括但不限于以下几种常见的方法:
在基因表达数据和计数数据分析中,数据通常呈现出高度偏斜的分布,而对数转换可以将其转换为对数空间,使得数据更加符合正态分布的假设,从而适用于更多统计分析和建模方法。
示例:
当进行log transformation时,我们可以使用R中内置的iris数据集作为示例。iris数据集是一个经典的数据集,包含了三个鸢尾花的品种(setosa、versicolor、virginica)的萼片(sepal)和花瓣(petal)的长度和宽度的测量值。
首先,我们需要加载iris数据集并绘制一个散点图,来查看两个特征变量(如花瓣长度和宽度)之间的关系。然后,我们将对其中一个特征变量进行log transformation,并再次绘制散点图来观察数据转换后的效果。
# 加载iris数据集> data(iris)# 绘制原始数据的散点图> library(ggplot2)> ggplot(iris, aes(x = Petal.Length, y = Petal.Width, color = Species)) ++ geom_point() ++ labs(title = "Scatter plot of Petal Length vs. Petal Width (Original Data)",+ x = "Petal Length", y = "Petal Width") ++ theme_minimal()
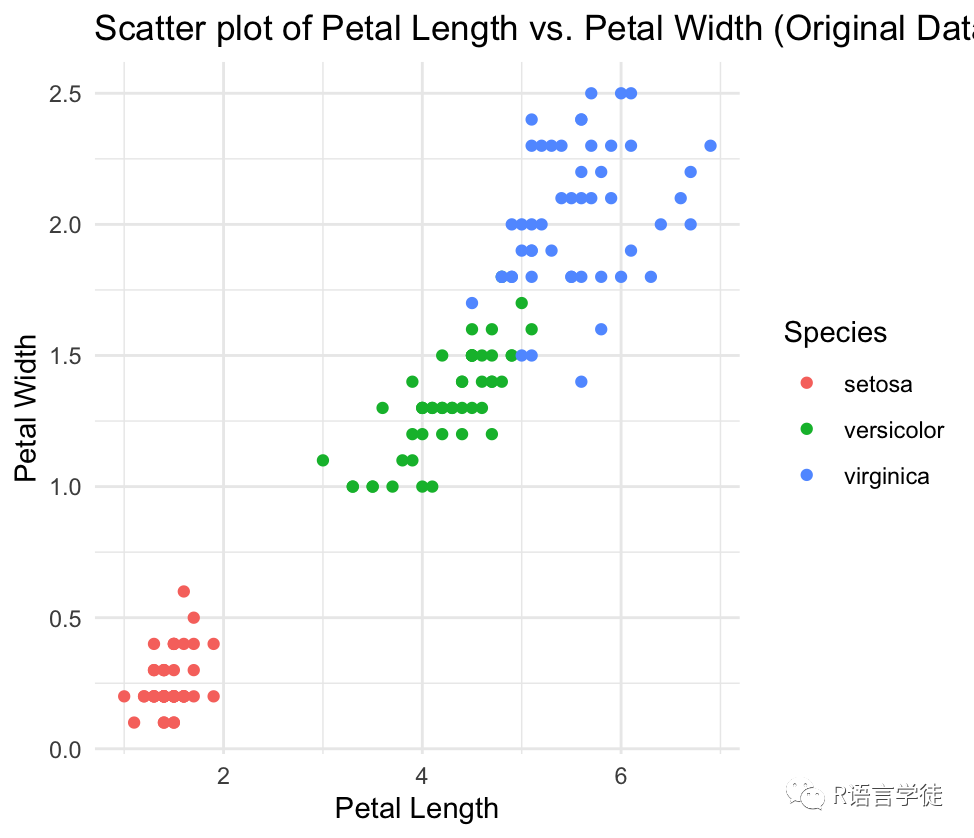
# 对Petal.Length进行log transformation> iris$log_Petal_Length <- log(iris$Petal.Length)# 绘制log transformed数据的散点图> ggplot(iris, aes(x = log_Petal_Length, y = Petal.Width, color = Species)) ++ geom_point() ++ labs(title = "Scatter plot of log(Petal Length) vs. Petal Width",+ x = "log(Petal Length)", y = "Petal Width") ++ theme_minimal()

可以处理广泛范围的正数数据,并且可以根据数据的特点选择合适的转换参数λ。当数据存在偏斜时,通过选择合适的λ,Box-Cox转换可以将数据转换为更接近正态分布的形式。在实验数据分析和建模中,假设数据符合正态分布是很常见的,因为许多统计方法都依赖于该假设。通常使用最大似然估计法来估计转换参数λ。在实际应用中,可以通过尝试不同的λ值来找到最佳的转换。
示例:
# 加载plant growth数据集> data(PlantGrowth)# 使用体重(weight)作为响应变量,拟合线性模型> fit_lm <- lm(weight ~ 1, data = PlantGrowth)# 进行Box-Cox转换,并找到最佳的lambda值> library(MASS)> boxcox_result <- boxcox(fit_lm)

# 输出最佳的lambda值> lambda_optimal <- boxcox_result$x[which.max(boxcox_result$y)]> cat("Optimal lambda:", lambda_optimal)Optimal lambda: 1.393939# 对数据进行Box-cox转换> data_boxcox_transformed <- (PlantGrowth$weight^lambda_optimal - 1) / lambda_optimal# 绘制转换后的数据直方图> hist(data_boxcox_transformed, main = "Histogram of Box-Cox Transformed Data", xlab = "Box-Cox Transformed Weight")

通过减去数据的均值,使数据集围绕零点对称分布,从而消除整体偏移。这对于一些统计模型的拟合和解释非常有用,因为一些模型(例如回归模型)在数据没有整体偏移时表现更好。只是对数据进行平移,不改变数据之间的相对关系和排列顺序。数据的相对差异和方差不会改变,也不会影响数据的比例关系。在多变量分析中,如果变量之间存在高度相关性,会引起共线性问题。通过对数据进行中心化,可以减少共线性,从而改善模型的稳定性和解释性。
示例:
我们将绘制原始数据的散点图,其中横轴是花瓣长度(Petal.Length),纵轴是花瓣宽度(Petal.Width)。然后,我们将对数据进行Centering Transformation,再绘制转换后的散点图,观察数据转换的效果。
# 加载iris数据集> data(iris)# 绘制原始数据的散点图(如示例1)> library(ggplot2)> ggplot(iris, aes(x = Petal.Length, y = Petal.Width, color = Species)) ++ geom_point() ++ labs(title = "Scatter plot of Petal Length vs. Petal Width (Original Data)",+ x = "Petal Length", y = "Petal Width") ++ theme_minimal()# 对Petal.Length进行Centering Transformation> centered_Petal_Length <- iris$Petal.Length - mean(iris$Petal.Length)# 绘制Centering Transformation后的数据的散点图> ggplot(data = iris, aes(x = centered_Petal_Length, y = Petal.Width, color = Species)) ++ geom_point() ++ labs(title = "Scatter plot of Centered Petal Length vs. Petal Width",+ x = "Centered Petal Length", y = "Petal Width") ++ theme_minimal()
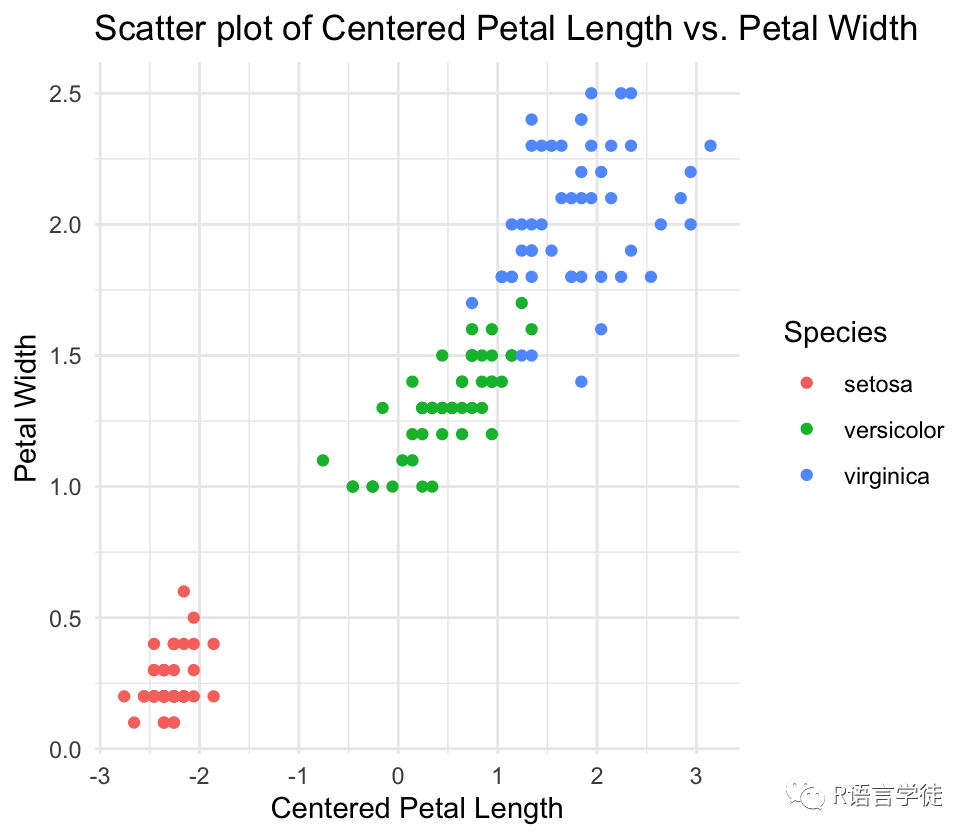
通过运用这些数据转换技术,生物学家能够从原始数据中获取更有意义的信息,帮助他们做出科学假设、探索基因功能、发现生物学模式和解释实验结果。值得强调的是,在进行数据转换时,研究人员需要深入了解数据的背景和研究目的,并根据数据的特性选择合适的转换方法。正确地应用数据转换能够提高数据质量和分析的可靠性,为生物学研究和应用奠定坚实基础。
以上就是对在R中如何运用数据转换的简单介绍啦,”transformations” 通常指对数据进行变换或转换,以便更好地理解和分析数据。在生物学数据方面,数据转换是常见的操作,可以用于预处理、标准化、归一化或转换生物学实验数据,以及执行数据可视化和统计分析。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小师妹哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html


END

