跟着小果展现数据的生动变化,轻松复现IF64.8文献中炫酷热力图,实现指定角度旋转!



公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240115
需要租赁服务器的小伙伴可以扫码添加小果,此外小果还提供生信分析,思路设计,文献复现等,有需要的小伙伴欢迎来撩~

library(ggplot2)#建一个散点图pp<-ggplot()+# 创建一个ggplot对象geom_point(aes(x=1,y=1))+#添加散点图的几何对象coord_cartesian(clip = "off")#设置坐标系为Cartesian坐标系
ppcowplot::ggdraw()+#创建一个ggdraw对象,用于绘制图形#将之前创建的ggplot对象pp转换为ggplotify对象,#指定旋转角度为-45度。#将前面定义的散点图进行了角度旋转的处理cowplot::draw_plot(ggplotify::as.ggplot(pp,angle = -45),width = 0.5,height = 0.5,#设置绘制图形的宽度和高度为0.5#设置水平和垂直方向的对齐方式hjust = -0.2,#vjust = -0.5)

library(readxl)#install.packages("rlang")dat<-read_excel("data1.xlsx")dat

#基础library(ggplot2)ggplot(data=dat,aes(x=x,y=y))+#添加热力图的几何对象geom_tile(aes(fill=log2(value)))+#表示要创建矩形热力图,#fill指定了矩形的颜色映射,使用log2(value)来填充颜色coord_equal(clip = "off") -> p#设置坐标系为等比例坐标系#clip = "off“表示图形元素可以超出坐标系的范围而不被裁剪p
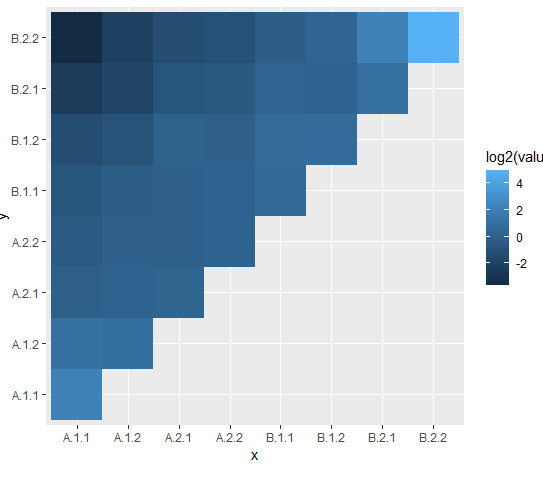
#添加文本标签library(tidyverse)dat %>% pull(x) %>% unique()#从数据集dat中提取x列的唯一值p+#添加文本标签的几何对象geom_text(data=data.frame(x=1:8,y=0:7,#创建了一个新的数据框label=dat %>% pull(x) %>% unique()),aes(x=x,y=y,label=label),angle=90)+theme_void() -> p1#将图形的主题设置为空p1

#之前创建的带有文本标签的热力图上添加了两个方框p1+#geom_path添加路径的几何对象,用于绘制两个方框geom_path(data=data.frame(x=c(0.5,0.5,4.5,4.5,3.5,3.5,2.5,2.5,1.5,1.5,0.5),#定义方框的形状y=c(0.5,4.5,4.5,3.5,3.5,2.5,2.5,1.5,1.5,0.5,0.5)),aes(x=x,y=y),#指定方框路径的x和y坐标color="black",size=1)+#设置路径的颜色为黑色,大小为1geom_path(data=data.frame(x=c(0.5,0.5,4.5,4.5,3.5,3.5,2.5,2.5,1.5,1.5,0.5)+4,y=c(0.5,4.5,4.5,3.5,3.5,2.5,2.5,1.5,1.5,0.5,0.5)+4),aes(x=x,y=y),color="black",size=1) -> p2p2
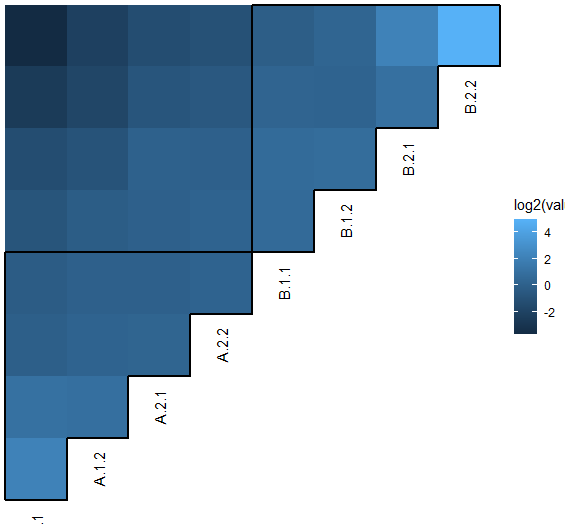
#修改配色###library(ggsci)library(scales) #用来展示颜色## 自然出版集团 (Nature Publishing Group) 风格的颜色方案mypal <- pal_npg("nrc", alpha = 0.7)(10)mypalshow_col(mypal,ncol = 5)#####之前创建的图形(带有两个方框的热力图)进行颜色和主题的调整#scale_fill_gradient2函数调整填充颜色的渐变p2+ scale_fill_gradient2(low = scales::muted("#DC0000B2"),#设置低值颜色为深红色,#使用muted函数调整颜色透明度 mid = "white",#设置中间值颜色为白色 high = scales::muted("#3C5488B2"),#设置高值颜色为深蓝色 midpoint = 0)+#设置颜色变化的中点为0#不显示图例 theme(legend.position = "none") -> p3p3
#额外注释#annotate函数添加线段注释p3+annotate(geom = "segment",x=0.4,xend=0.4,y=0.5,yend=4.5,#从坐标(0.4, 0.5)到(0.4, 4.5)的线段size=2,#设置线段的粗细color="#DC0000B2")+annotate(geom = "text",x=0.2,y=2.5,label="A",#在坐标(0.2, 2.5)处添加文本"A"angle=45,#设置文本的角度为45度size=5)+#文本大小为5annotate(geom = "segment",x=4.5,xend=8.5,y=8.6,yend=8.6,size=2,#置线段的粗细color="#3C5488B2")+annotate(geom = "text",x=6.5,y=8.8,label="B",#在坐标(6.5, 8.8)处添加文本"B"angle=45,#置文本的角度为45度size=5) -> p4p4
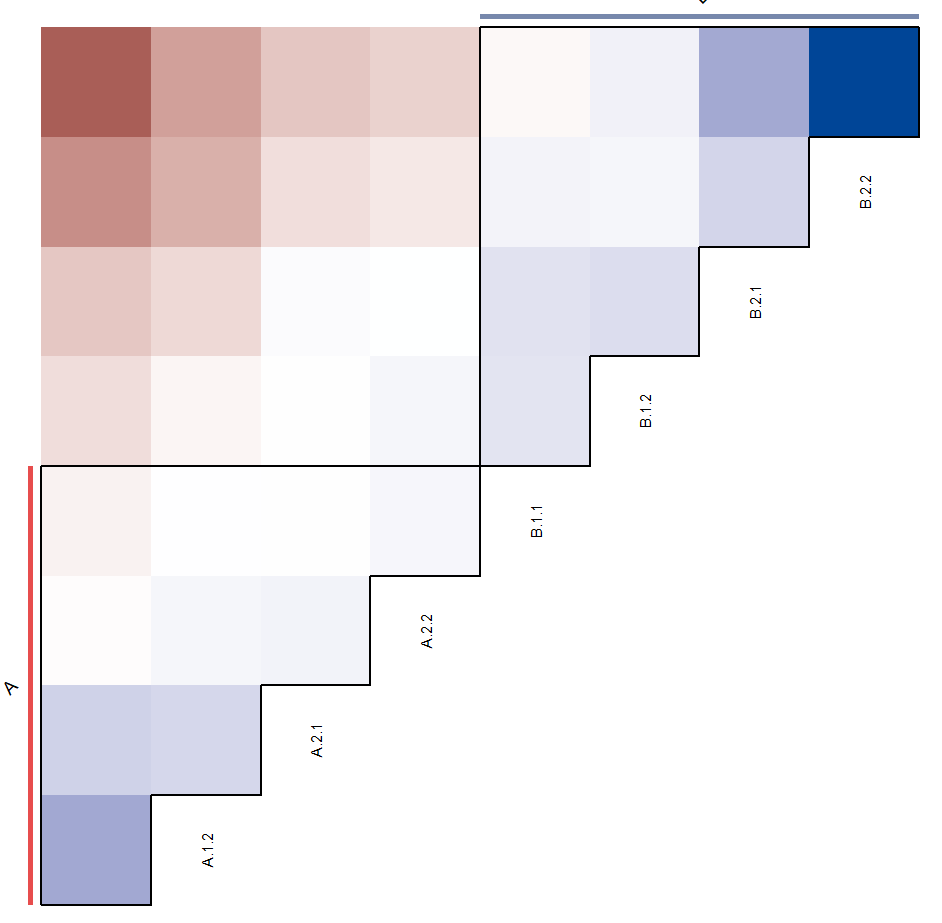
#旋转45library(cowplot)ggdraw()+#创建一个ggdraw对象draw_plot(ggplotify::as.ggplot(p4,angle = -45),#将之前创建的p4图形转换为ggplot对象,指定旋转角度为-45度width = 0.7,height = 0.7,#设置插入图形的宽度和高度为0.7#设置插入图形的水平和垂直方向的对齐方式hjust = -0.2,vjust = -0.1)+#annotate(geom = "text",#添加文本注释x=0.5,y=0.2,label="Chromatin subcompartments") -> p5p5

#对之前创建的图形p2进行一些调整p2+scale_fill_gradient2(low = scales::muted("#DC0000B2"),mid = "white",high = scales::muted("#3C5488B2"),midpoint = 0)+theme(legend.position = "bottom")+#将图例位置设置在底部guides(fill=guide_colorbar(title.position = "top",title.hjust = 0.5,barwidth = 20)) -> p3.1p5+annotation_custom(grob = ggpubr::get_legend(p3.1),#获取p3.1图形的图例xmin = 0.1,xmax=0.9,#置插入图例的水平位置范围ymin=0.1,ymax=0.2#设置插入图例的垂直位置范围)-> p6p6

#多个library(patchwork)p6+p6

小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果


往期回顾
|
01 |
|
02 |
|
03 |
|
04 |