宏基因组质控过程中FastQC使用及结果解读
点击蓝字 关注我们
今天小花就为大家介绍一下宏基因组分析的第一步—质控中质量评估软件FastQC的使用和结果解读。
FastQC用于快速评估测序数据的质量。它能够生成质量报告,显示各个碱基的质量分数和序列质量分布,帮助检测低质量的碱基和潜在的测序问题。接下来小花就来教大家如何安装和使用FastQC。
小花主要对网页版为大家解读。

接下来小花就从上到下依次为大家展示和分析结果图。

这个图横轴为read长度,纵轴为质量得分,柱状表示该碱基位置所有序列的测序质量的统计,我们这个reads每个碱基质量都是很好的。

这个图是每条reads的quality的均值的分布,横轴表示质量分数,纵轴表示每个值对应的reads数目,当测序结果主要集中在高分中,证明测序质量良好。
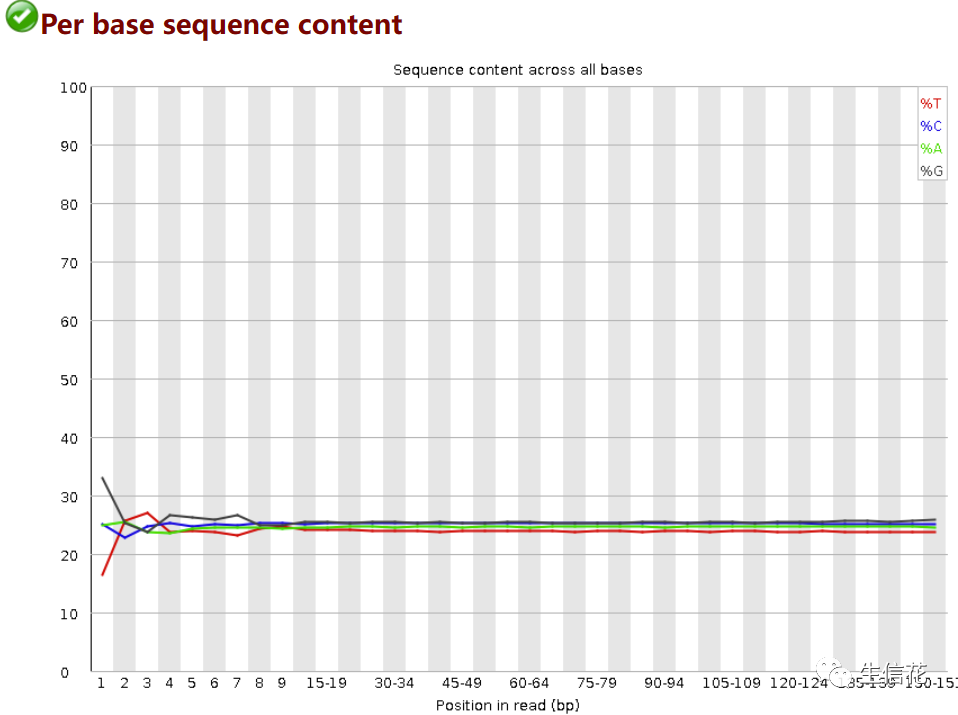
这个图是对所有reads的每一个位置的ATCG四种碱基的分布图,用于检查是否有AT,GC分离现象。横轴为碱基长度分布,纵轴表示该位置所含对应碱基的百分比,图中4条线分别代表A,C,T,G在每个位置上的平均含量。
接着我们就可以使用fastp进行质量控制、适配体修剪、NGS引物修剪等质控过程啦。

这个图统计reads的平均GC含量的分布,红线是我们的数据的情况,蓝线是理论分布(正态分布),注意这儿小花也是一个×,不过宏基因组数据的GC含量不合格是很正常的。

这个图是显示reads每个位置出现N的比率。小花为大家解释一下N是什么:当出现测序仪不能分辨的碱基时会产生N,因此过多的N就代表我们的测序数据质量并不高。

这个图是reads长度的分布,质量好的数据往往reads的长度都集中分布在一个地方。
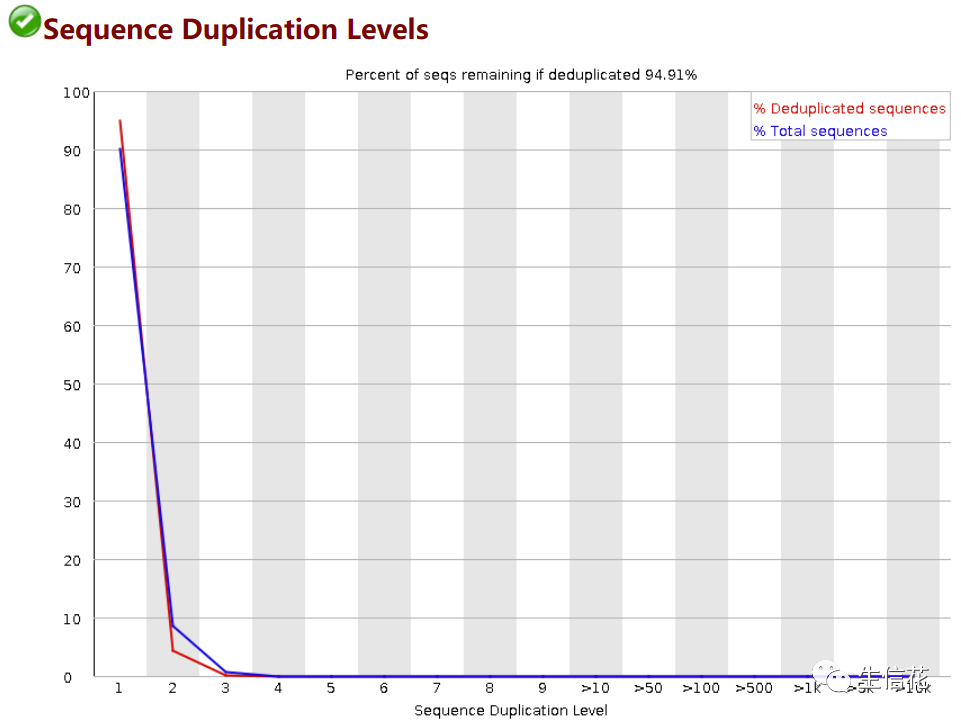
这个图是统计序列完全一致的reads的频率,横坐标是duplication的次数,纵坐标是duplicated reads的数目,以unique reads的总数作为100%。

这个结果说明我们数据中没有over-represented的序列,即没有某个大量出现的序列。

这个图显示我们的序列中是否有接头,如果有就必须要去除,横轴表示碱基位置,纵轴表示接头所占的百分比。
好啦,这就是FastQC的使用教程和结果解读啦,后续小花会为大家带来质量控制、适配体修剪、NGS引物修剪等质控软件,希望大家能够跟着小花学习更多宏基因组的知识。
欢迎使用:云生信 – 学生物信息学 (biocloudservice.com)
如果想用服务器可以联系微信:18502195490(快来联系我们使用吧!)

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

