一看就会的scRNA-seq细胞通讯分析,用CellChat揭示细胞间的加密通话
 亲爱的果粉儿们,小果又来和大家分享新知识啦!今天我要告诉大家的是,细胞之间并不是孤立存在的,他们其实是通过一种神秘的方式——细胞通讯,来进行信息的传递。而小果今天要讲的CellChat,就是那把可以解密这种通讯的神奇钥匙!接下来,就让小果带大家一起破解他们的加密通话吧。
亲爱的果粉儿们,小果又来和大家分享新知识啦!今天我要告诉大家的是,细胞之间并不是孤立存在的,他们其实是通过一种神秘的方式——细胞通讯,来进行信息的传递。而小果今天要讲的CellChat,就是那把可以解密这种通讯的神奇钥匙!接下来,就让小果带大家一起破解他们的加密通话吧。
#加载工具包library(CellChat)library(patchwork)options(stringsAsFactors = FALSE)
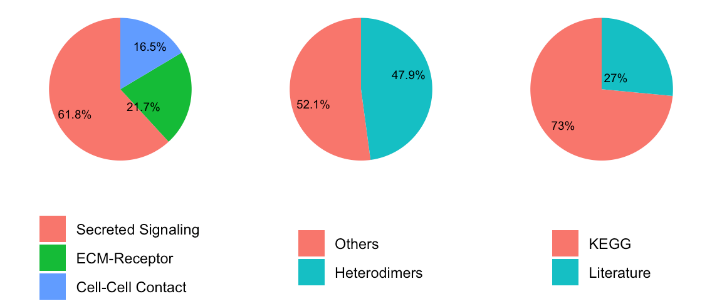
# CellChat需要两个输入文件:一个是标准化的基因表达矩阵,而另一个是细胞分组信息。#这里我们下载并载入官方提供的示例文件#包含正常(NL)和疾病(LS)两种样本(https://ndownloader.figshare.com/files/25950872)load("data_humanSkin_CellChat.rda")# 提取标准化矩阵data.input = data_humanSkin$data# 提取细胞分组信息,meta是一个数据框,包含示例数据中细胞类型、批次、样本等信息meta = data_humanSkin$meta#此处我们只提取疾病数据做后续分析cell.use = rownames(meta)[meta$condition == "LS"]# 准备CelChat分析的输入数据data.input = data.input[, cell.use]meta = meta[cell.use, ]#查看细胞类型标签unique(meta$labels)# 创建CellChat对象cellchat <- createCellChat(object = data.input, meta = meta, group.by = "labels")#给cellchat对象添加元数据cellchat <- addMeta(cellchat, meta = meta)#设置"labels"作为默认的细胞标签cellchat <- setIdent(cellchat, ident.use = "labels")#显示有多少种细胞类型levels(cellchat@idents)#查看每种细胞类型的细胞数groupSize <- as.numeric(table(cellchat@idents))# 设置配体-受体相互作用数据库,CellChatDB提供人类和鼠两个物种的数据库# 如果分析的是鼠类数据则设置CellChatDB.mouseCellChatDB <- CellChatDB.human#展示数据库中具体信息,人类CellChatDB包含1,939个经过验证的分子相互作用,包括61.8%的旁分泌/自分泌信号相互作用,21.7%的细胞外基质(ECM)受体相互作用和16.5%的细胞-细胞接触相互作用。showDatabaseCategory(CellChatDB)
# 展示数据库结构dplyr::glimpse(CellChatDB$interaction)# 使用CellChatDB的子集进行细胞-细胞通讯分析CellChatDB.use <- subsetDB(CellChatDB, search = "Secreted Signaling")# 在对象中设置我们要使用的数据库cellchat@DB <- CellChatDB.use#预处理细胞-细胞通讯分析的表达数据#使用subsetData函数,比对整个数据库,筛选出cellchat对象中有意义的基因和细胞,cellchat <- subsetData(cellchat)#设置并行计算的模式和工作数,加快后续的分析速度,这里设置为多进程模式,指定了4个工作数。future::plan("multiprocess", workers = 4)#使用identifyOverExpressedGenes函数,识别cellchat对象中每个细胞类型中相对于其他细胞类型过表达的基因。cellchat <- identifyOverExpressedGenes(cellchat)#使用identifyOverExpressedInteractions函数,识别出cellchat对象中每个细胞类型中相对于其他细胞类型过表达的配体-受体对。cellchat <- identifyOverExpressedInteractions(cellchat)#将基因表达数据投射到PPI,这样做的目的是为了寻找那些可能参与细胞间通讯的蛋白质复合物。cellchat <- projectData(cellchat, PPI.human)

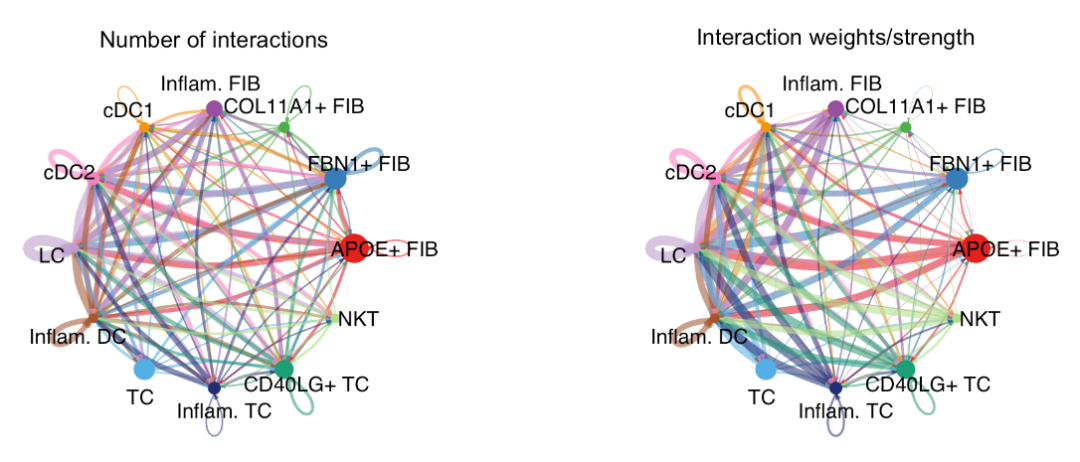
# computeCommunProb函数,计算cellchat对象中每对细胞类型之间的通讯概率。cellchat <- computeCommunProb(cellchat)#如果某些细胞类型中只有少量细胞,则过滤掉细胞间通信cellchat <- filterCommunication(cellchat, min.cells = 10)# computeCommunProbPathway函数,计算cellchat对象中每对细胞类型之间的通讯通路概率,通讯通路概率是指在给定的信号分子对下,一个细胞类型向另一个细胞类型发送信号的概率,同时考虑了信号分子对所属的通讯通路(communication pathway),通讯通路是指一组具有相同或者相似功能的信号分子对。cellchat <- computeCommunProbPathway(cellchat)# 计算聚合细胞-细胞通信网络cellchat <- aggregateNet(cellchat)# 我们还可以看到聚合的细胞-细胞通信网络。groupSize <- as.numeric(table(cellchat@idents))#将图形设备分为一行两列的布局,用来显示两个图形。设置xpd参数为TRUE,表示允许图形元素超出图形区域。par(mfrow = c(1,2), xpd=TRUE)#使用netVisual_circle函数,绘制cellchat对象中的net元素中的count和weight两个矩阵#的圆形网络图,count矩阵表示每对细胞类型之间的通讯信号对的数量,weight矩阵表示#每对细胞类型之间的通讯概率。netVisual_circle(cellchat@net$count, vertex.weight = groupSize, weight.scale = T, label.edge= F, title.name = "Number of interactions")netVisual_circle(cellchat@net$weight, vertex.weight = groupSize, weight.scale = T, label.edge= F, title.name = "Interaction weights/strength")
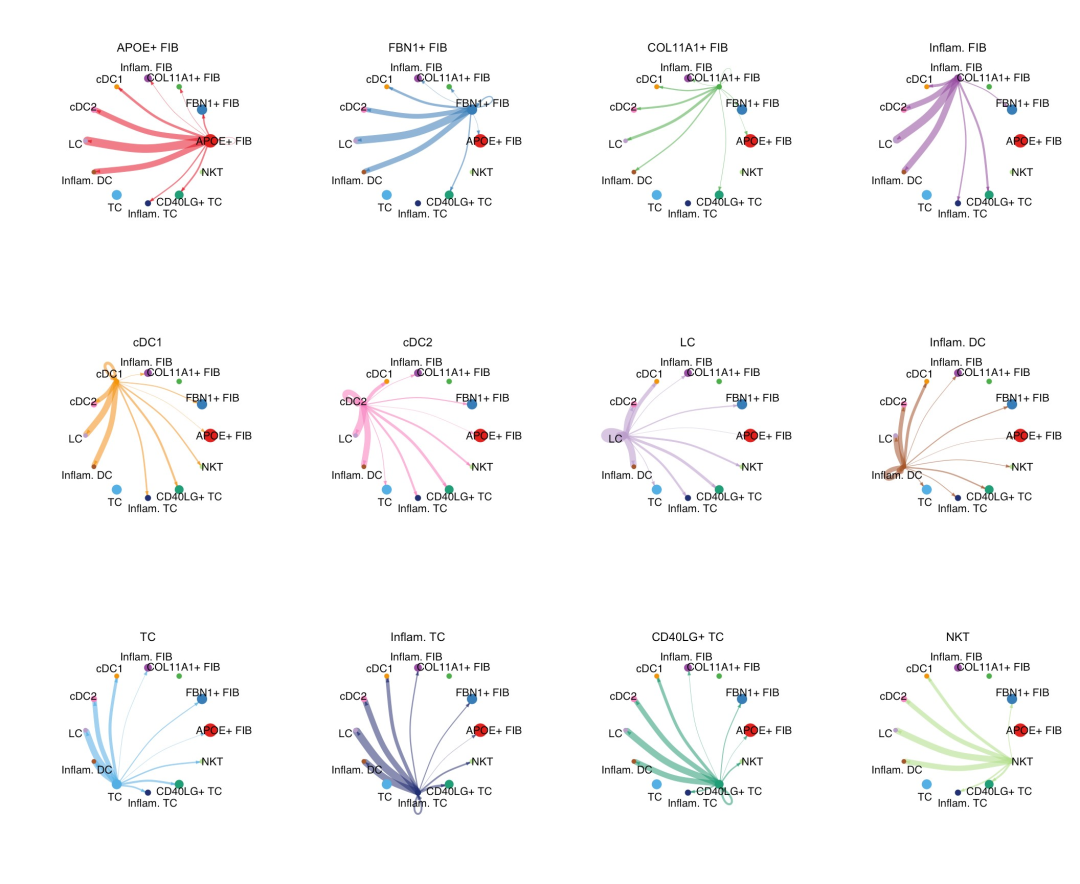
# 由于复杂的细胞-细胞通信网络,我们可以检查从每个细胞群发送的信号。这里我们还控制了参数,以便我们可以比较不同网络之间的边权重mat <- cellchat@net$weightpar(mfrow = c(3,4), xpd=TRUE)for (i in 1:nrow(mat)) {mat2 <- matrix(0, nrow = nrow(mat), ncol = ncol(mat), dimnames = dimnames(mat))mat2[i, ] <- mat[i, ]netVisual_circle(mat2, vertex.weight = groupSize, weight.scale = T, edge.weight.max = max(mat), title.name = rownames(mat)[i])}
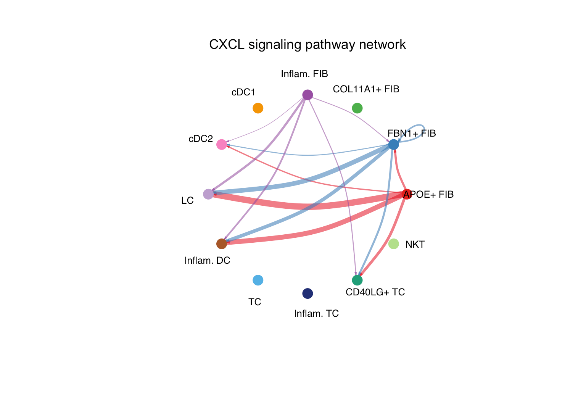
# CellChat提供了几种可视化细胞-细胞通信网络的方法,包括圆图、弦图、热图和气泡图等。# 这里我们以一个信号通路CXCL为例。pathways.show <- c("CXCL")# 圆图par(mfrow=c(1,1))netVisual_aggregate(cellchat, signaling = pathways.show, layout = "circle")
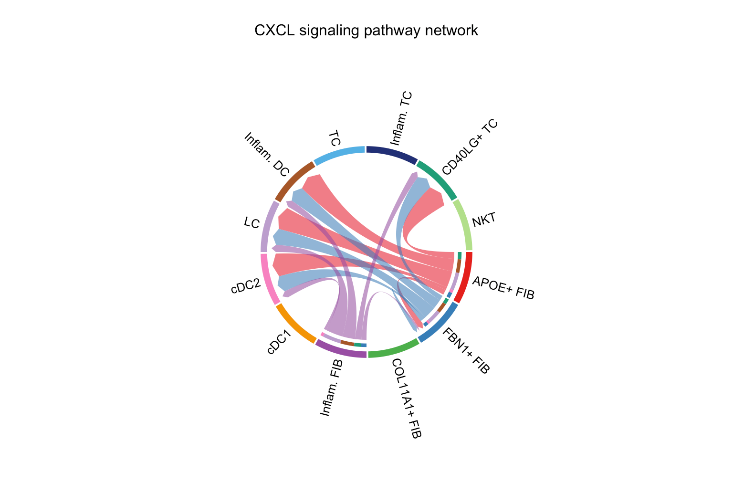
par(mfrow=c(1,1))netVisual_aggregate(cellchat, signaling = pathways.show, layout = "chord")
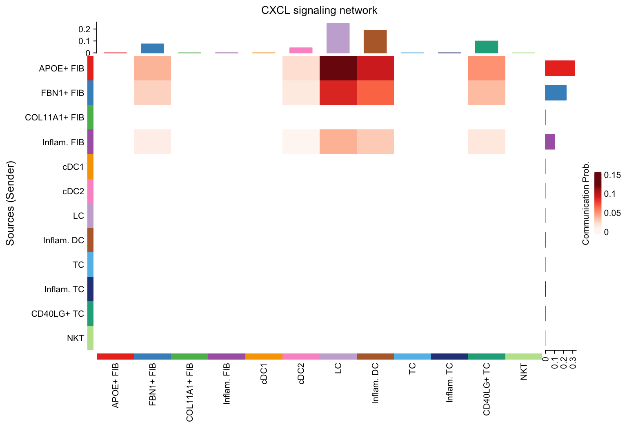
par(mfrow=c(1,1))netVisual_heatmap(cellchat, signaling = pathways.show, color.heatmap = "Reds")
往期推荐

