RNA-seq差异分析实战指南:利用DESeq2 R包精准解析差异表达并可视化基因调控!

当涉及到RNA-seq差异分析时,你是否曾经感到困惑或无从下手?本期,小果将带小伙伴们揭开RNA-seq分析的神秘面纱!RNA-seq技术为我们打开了一个全新的窗口,让我们能够深入了解基因表达的奥秘。而在这个探索过程中,DESeq2 R包成为了我们不可或缺的得力助手。凭借其强大的统计功能和精准的分析能力,我们可以精准解析RNA-seq数据中的差异表达。不仅如此,通过可视化手段,我们能够直观地洞察基因调控,并发现隐藏在数据背后的生物学意义。无论是基础研究还是临床应用,DESeq2都能为我们提供有力的支持。那么,你还在等什么?快来和小果一起探索RNA-seq差异分析的无限可能吧!同时呢,十年的生信之路,小果已经练就了一身扎实的本领,现在准备用这身本事为小伙伴们服务啦!如果你在生信分析上遇到了难题,那就来找小果吧!小果会用自己的专业知识和技能,为你解决困扰,助你一臂之力。期待你的联系哦~
一、DESeq2包简要说明
DESeq2包是一个为RNA-seq数据中的高维计数数据设计的R语言包,专注于数据的归一化、可视化和差异表达分析。其主要功能是利用经验贝叶斯方法(empirical Bayes techniques)来估计对数倍数变化(log2foldchange)和离差的先验值,并计算这些统计量的后验值。这也就是为什么DESeq2能够在RNA-seq数据中精确地探测基因差异表达的原因啦~
lDESeq2包差异表达分析步骤:
ØDESeq2首先对原始reads进行建模,使用标准化因子(scale factor)来解释库深度的差异。
ØDESeq2估计基因的离散度,并缩小这些估计值以生成更准确的离散度估计,从而对reads count进行建模。
ØDESeq2拟合负二项分布的模型,并使用Wald检验或似然比检验进行假设检验。
²小果提示:在进行差异表达分析之前,需要准备好输入数据。DESeq2包使用基于计数的RNA-seq数据作为输入。通常,我们需要将原始测序数据映射到基因或转录本水平,并计算每个基因或转录本的读数。
二、代码实操
小果这里将用一段代码和小伙伴们一起学习DESeq2包进行RNA-seq差异分析的一个典型流程。本次介绍的R包操作会占用内存较大,小果建议使用服务器,欢迎小伙伴们联系小果租赁性价比居高的服务器!
公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240520
需要租赁服务器的小伙伴可以扫码添加小果,此外小果还提供生信分析,思路设计,文献复现等,有需要的小伙伴欢迎来撩~

l包的安装与加载:首先,检查是否安装了BiocManager,如果没有,则进行安装。
if (!requireNamespace("BiocManager", quietly = TRUE)) {install.packages("BiocManager")}BiocManager::install("DESeq2")library(DESeq2)
l进行模拟数据集的生成:小果这里使用了set.seed确保模拟结果的可重复性。定义条件和样本。使用rpois生成矩阵,模拟了基因在样本中的表达水平。
set.seed(123)conditions <- c("Condition1", "Condition2")samples <- paste0("Sample_", 1:6)counts <- matrix(rpois(60, 1000), ncol = 6)colData <- data.frame(sample = samples,condition = factor(rep(conditions, each = 3))
l创建DESeq2数据集对象:用DESeqDataSetFromMatrix创建DESeq2数据集对象dds,其中包含了计数数据和样本信息。
dds <- DESeqDataSetFromMatrix(countData = counts, colData = colData, design = ~ condition)
l数据预处理:先用estimateSizeFactors对数据进行标准化,以消除样本之间的大小差异,然后用estimateDispersionsGeneEst进行基因级别的离散度估计,并更新dds对象。
dds <- estimateSizeFactors(dds)dds <- estimateDispersionsGeneEst(dds)dispersions(dds) <- mcols(dds)$dispGeneEst
l差异表达分析:用nbinomWaldTest拟合负二项模型并进行差异表达分析。
dds <- nbinomWaldTest(dds)
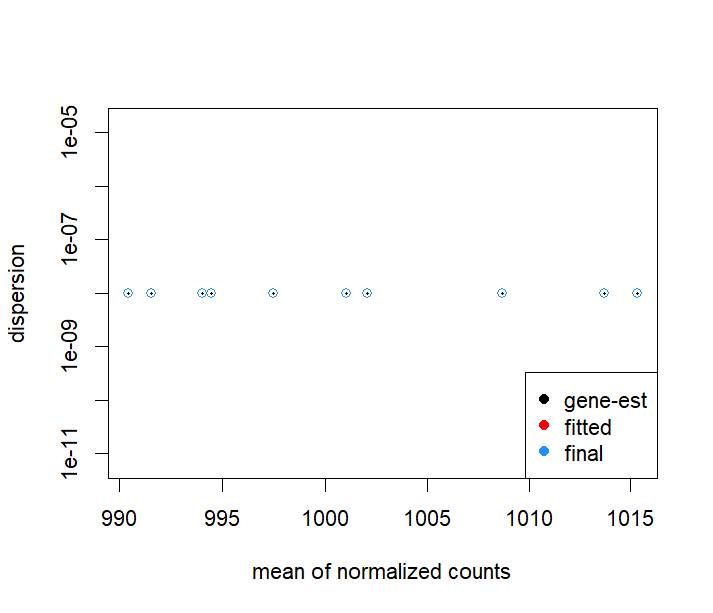
l离散度分布的可视化:用plotDispEsts查看数据的离散度分布,这有助于判断模型的适用性。
plotDispEsts(dds, fitType="local")
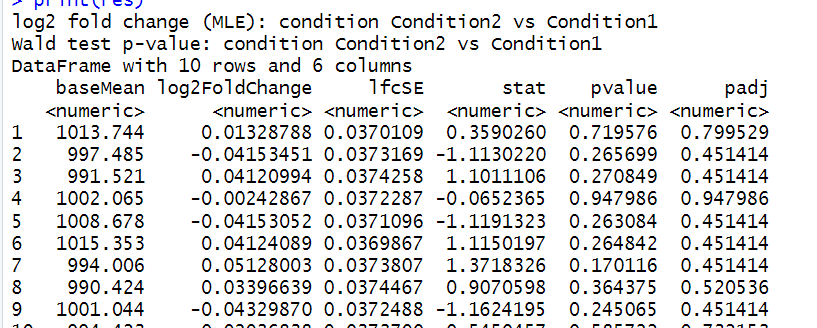
l差异表达基因结果的获取与过滤:使用results获取差异表达分析的结果后通过subset过滤出FDR校正后P值小于0.05的显著性基因。
res <- results(dds)print(res)resSignificant <- subset(res, padj < 0.05)
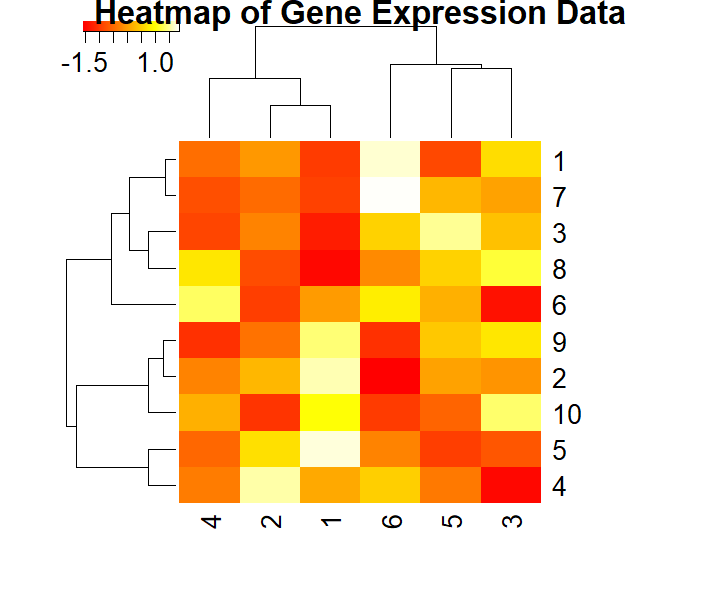
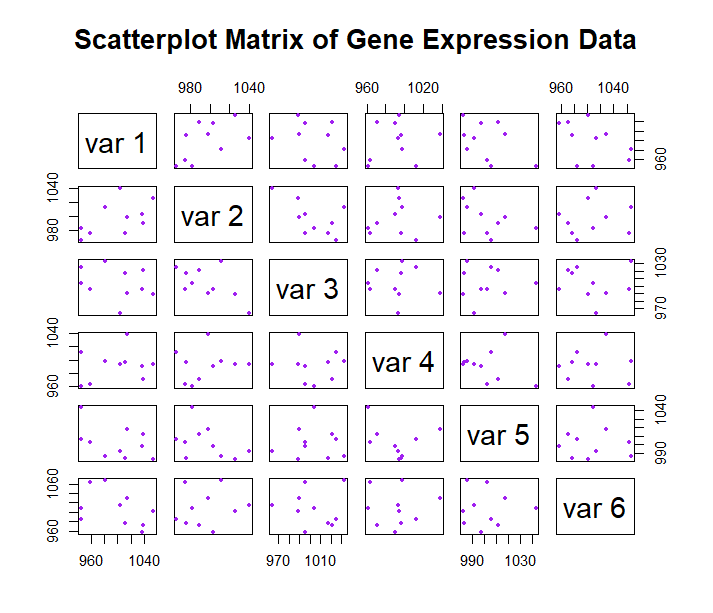
l数据可视化(检查数据质量):小果这里进行了多种可视化~plotMA绘制火山图,展示基因的log2 fold change和显著性水平;boxplot绘制箱线图,检查基因表达数据中的异常值;vioplot绘制小提琴图,展示基因表达数据的分布;stripchart绘制点图,展示每个样本中每个基因的表达量;heatmap3绘制热图,展示基因表达量的分布;pairs绘制散点图矩阵,展示基因表达量之间的成对关系。
plotMA(res, main="Volcano Plot")boxplot(assay(dds), las = 2, col = "skyblue", pch = 20,main = "Boxplot of Gene Expression Data")if (!requireNamespace("vioplot", quietly = TRUE)) {install.packages("vioplot")}library(vioplot)vioplot(assay(dds), col = "lightgreen", pch = 20,main = "vioplot of Gene Expression Data")stripchart(assay(dds), method = "jitter", pch = 20, col = "yellow",main = "Stripchart of Gene Expression Data")install.packages("heatmap3")library(heatmap3)heatmap3(assay(dds), scale = "row", col = heat.colors(100),main = "Heatmap of Gene Expression Data")pairs(assay(dds), col = "purple", pch = 20,main = "Scatterplot Matrix of Gene Expression Data")
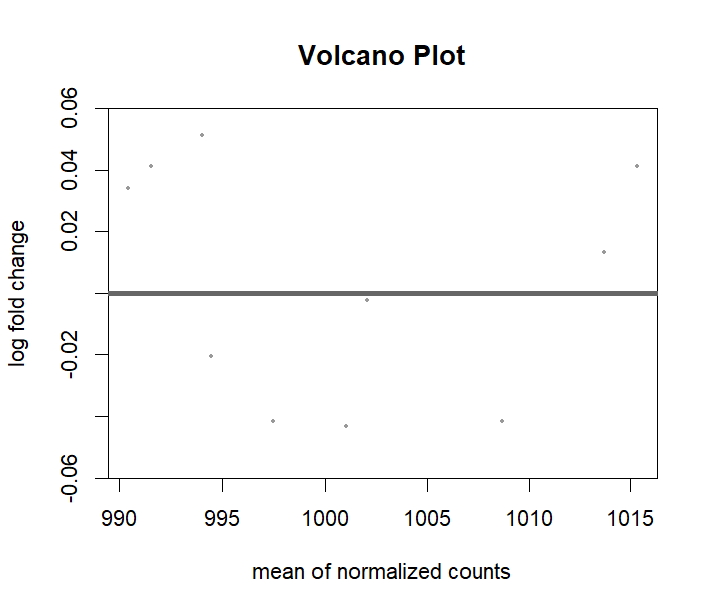
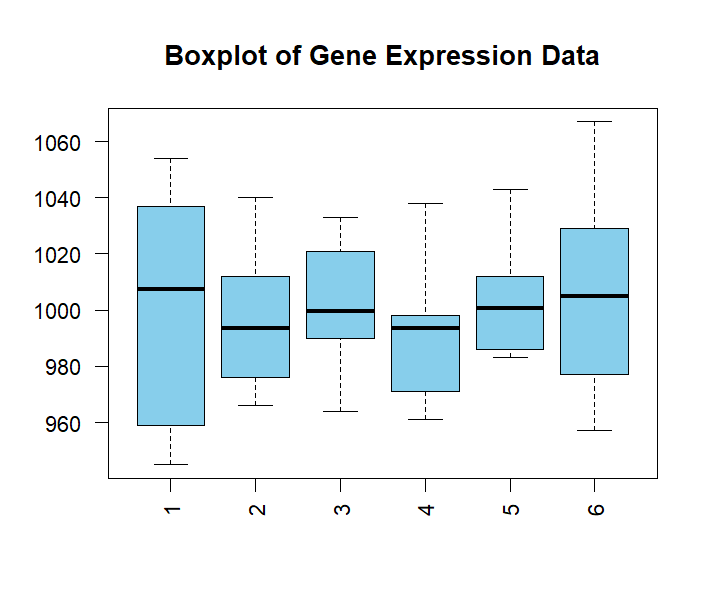
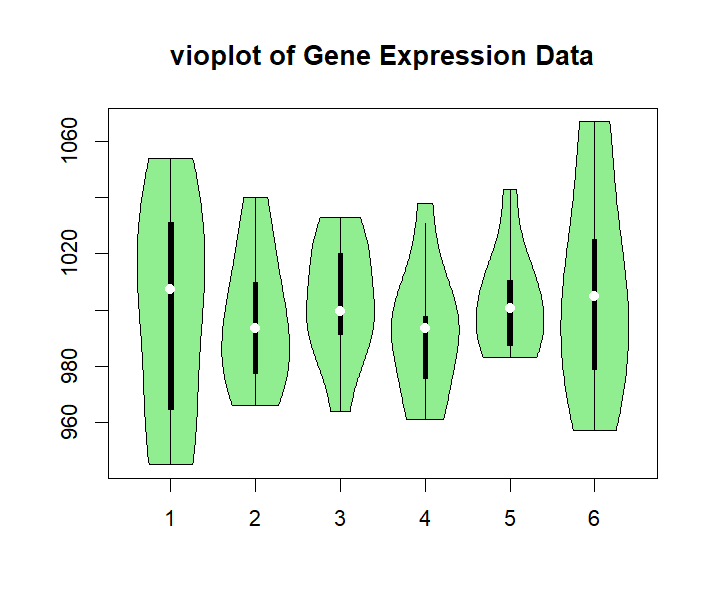
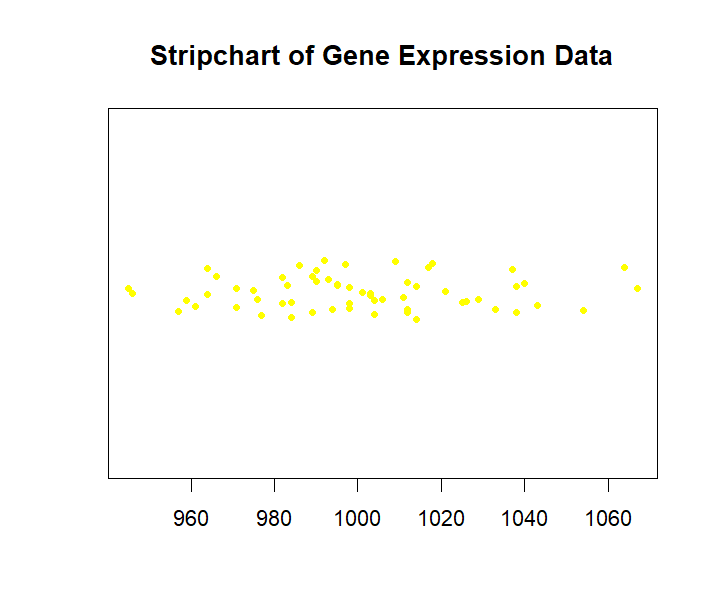
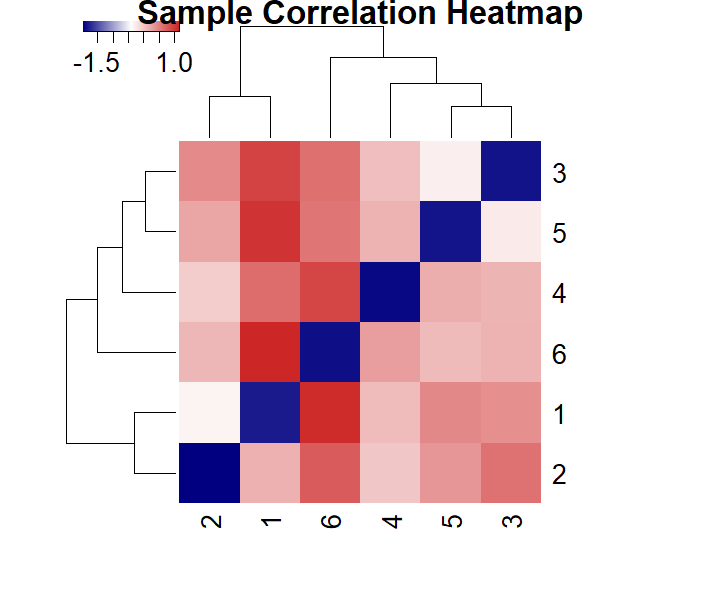
l样本相关性分析:用dist函数计算样本间的欧氏距离,并使用heatmap3绘制样本相关性热图。
sampleDists <- dist(t(assay(dds)))sampleDistMatrix <- as.matrix(sampleDists)heatmap3(sampleDistMatrix, main="Sample Correlation Heatmap")
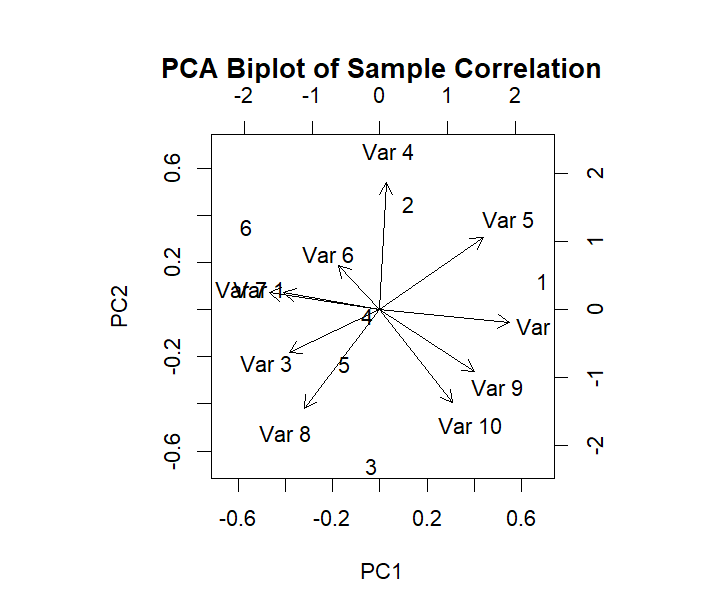
l主成分分析(PCA):使用prcomp进行PCA分析,并通过biplot绘制前两个主成分的二维投影,以展示样本间的相关性。
pca_result <- prcomp(t(assay(dds)), scale. = TRUE)biplot(pca_result, scale = TRUE, col = as.numeric(colData(dds)$condition),pch = 19, main = "PCA Biplot of Sample Correlation")
三、文章小结
本期,小果为小伙伴们讲解了DESeq2 R包在基因表达数据分析中的应用。我们模拟了基因表达数据,并利用DESeq2进行数据预处理、差异表达分析,并通过火山图展示分析结果。此外,通过箱线图、点图、热图和散点图等多种可视化手段,深入探索了数据集的内在结构和样本间的相互关系。最后,通过主成分分析(PCA)进一步分析了样本间的相关性。整个过程不仅揭示了差异表达基因,还深入分析了对数据集。快来和小果一起尝试DESeq2,解锁你的基因表达数据之秘吧!希望本期的内容对你有帮助,期待我们下期再见!最后如果各位小伙伴们觉得自己运行代码太麻烦,欢迎用我们的云生信小工具,只要输入合适的数据就可以直接出想要的图呢,附云生信链接
(http://www.biocloudservice.com/home.html)。
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果

往期回顾
|
01 |
|
02 |
|
03 |
|
04 |