「付费」【代码-6】全网最简单最适合小白!MCPcounter免疫浸润和相关性分析教程
收录于话题
今天小果想带着大家学习 如何利用R包MCPcounter(https://github.com/ebecht/MCPcounter)进行免疫浸润分析,然后与自己感兴趣的目标基因集进行相关性分析,最终绘制相关性热图,通过该分析可以筛选出影响免疫浸润的候选基因,小伙伴们是不是觉得该分析很有用奥,那就赶紧行动起来吧!跟着小果一起开始今天的学习之旅。(Ps:为小伙伴附上如何安装该包的截图以及如何使用该包)
1. 如何进行MCPcounter分析?
如何进行MCPcounter分析,在进行分析之前,小果想简单的介绍一下该包,该R包可以通过归一化后的转录组数据量化异质组织中8个免疫细胞和两个基质细胞的丰度,分数的高低可以显示在免疫微环境中的浸润程度。小伙伴们只需要安装好该包,准备好基因表达矩阵文件作为输入文件,分析就非常非常简单,一句代码就可以完成分析奥!So easy! 非常适合小白!还不快跟着小果开始今天的实操。
2.安装需要的R包
install.packages("ggplot2")install.packages("tidyverse")install.packages("reshape2")install.packages("corrplot")install.packages("devtools")library(devtools)install_github("ebecht/MCPcounter",ref="master", subdir="Source")
结果文件
1. MCPcounter.txt
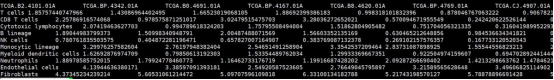
该结果文件为利用MCPcounter包计算的所有样本的8个免疫细胞和两个基质细胞的丰度,行名为免疫细胞类型,列名为样本名。
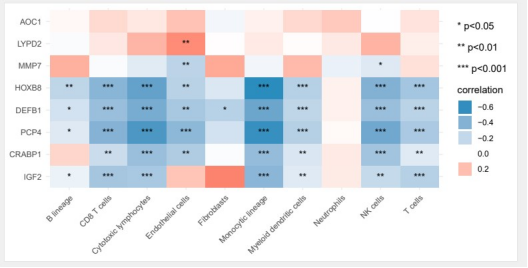
2. correlation.pdf
该结果图片为基于目标基因与免疫细胞的相关性热图,横坐标为免疫细胞类型,纵坐标为基因名称,用颜色来表示相关系数大小,星号来表示pvalue值,不同星号表示不同的pvalue值范围。1个星号表示pvalue值小于0.05大于0.01,两个星号表示pvalue值小于0.01,通过该热图可以很迅速的筛选出感兴趣的目标基因集中,那些基因对免疫浸润的影响大,最终筛选出候选基因。
如果需要完整的代码可以点击付费获取哦!今天小果的分享就到这里,如果小伙伴有其他数据分析需求,可以尝试本公司新开发的生信分析小工具云平台,季代码完成分析,非常方便奥!
往期推荐
(扫码领取整理好的输入文件,代码文件及示例结果)
