小果带你学生信之SRA文件的下载
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

SRA数据库:Sequence Read Archive,是NCBI网站下用于保存测序数据的数据库,大部分发表paper的测序数据都会上传至SRA数据库,SRA则会将原始数据压缩为.sra格式的文件方便大家下载。
ENA数据库:是欧洲生物信息研究所EBI旗下保存测序数据的数据库,与SRA功能类似,相较于SRA数据库只提供.sra格式的文件,ENA还支持直接下载fastq和bam,是生信小白入门的不二之选。
当我们在paper中获得测序数据的SRR号后,可以现在ENA数据库上搜索(ENA下载速度比SRA快不少),如果没有再去SRA上查询。
复现paper当然少不了将高通量测序数据down至本地服务器,目前从数据库down文件的方法主要有两种:其一为使用wget或者curl等命令从FTP上下载文件(一定要加上-c开启断点续传,别问为什么,谁明白down了一晚的文件不能用的苦),其二就是NCBI官网的sratoolkit工具中的prefetch,prefetch还支持SRR号下载,测序文件自动按照SRR号归档。当然,土豪也可以用浏览器、迅雷下载了再上传至服务器,下载速度也都不慢。
首先当然是NCBI SRA Toolkit,作为懒癌星人,不要跟我说什么源码变异、环境路径,没有什么是conda解决不了的,如果有,那就上bioconda。

当然该面对的问题还得面对,conda默认的sratools版本号太低,SRA数据库都该https了,sratools还是http,不过没关系,bioconda里的sratools2.10版本还能用,缝缝补补接着用吧,还能上源码咋滴。

接下来就是实操环节了,给朕上conda
conda install -c bioconda sra-tools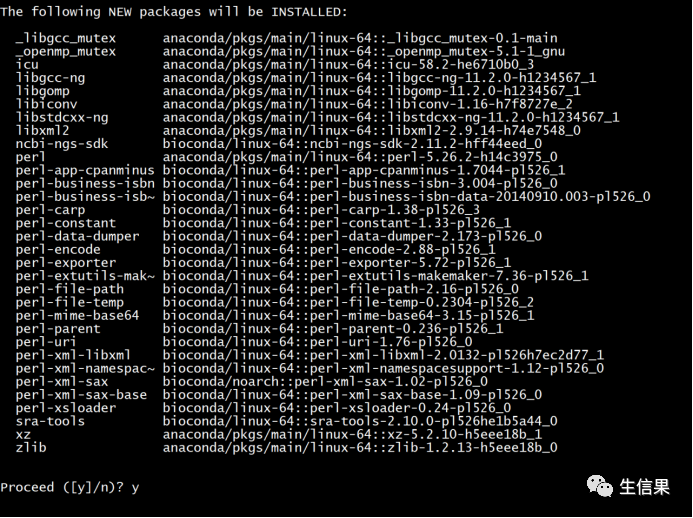
无脑y,依赖什么的对于conda都是撒撒水啦。
接下来上实战,以SRP193866为例
prefetch SRR8956151

Sratools默默开始工作了,当然,服务器也不能一直放着下载,爱妃还是去后台呆着吧
$ prefetch SRR8956151 &在最后加个&就能让命令后台候着了。
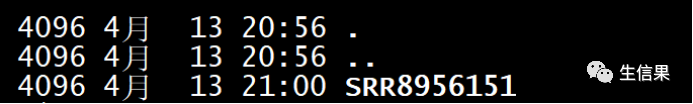
可以看到sratools自动生成SRR号文件夹,并将测序数据.sra下载至文件夹中。
好啦,今天就到这吧,欲知后事(sra2fastq)如何,欢迎关注小果,以免错过更新哟。
如果小伙伴有其他数据分析需求,可以尝试使用本公司新开发的生信分析小工具云平台,零代码完成分析,非常方便奥,云平台网址为:(http://www.biocloudservice.com/home.html),其中也包括了通路表达分析(http://www.biocloudservice.com/313/313.php),单细胞的基因共表达分析(http://www.biocloudservice.com/906/906.php)等各种小工具哦~,有兴趣的小伙伴可以登录网站进行了解。
点击“阅读原文”立刻拥有
↓↓↓