快来!3分钟轻松绘制基因ROC曲线
点击蓝字
关注小图
今天小图绘制了ROC曲线,迫不及待的想分享给小伙伴们~
相信有很多小伙伴在最近的分析中也有用到吧
对于第一次接触ROC曲线的小伙伴也感觉有些云里雾里,这不你的帮手小图来了
应用场景
在选择诊断试验时,许多研究者会在灵敏度和特异度之间进行艰难的取舍。那么,是否可以综合考虑灵敏度和特异度的特点,根据一个指标评价诊断试验的准确性呢?Lusted于1971年提出受试者工作特征曲线(receiver operating characteristic curve,简称ROC曲线),用来描述诊断试验的内在真实程度,应用十分广泛。
ROC(Receiver Operating Characteristic)曲线即受试者工作特征曲线。ROC曲线与坐标轴围成的面积被称为AUC(Area Under Curve),这两个指标和敏感性、特异性和准确性一起,是评估算法模型性能常用的指标。
在进一步介绍ROC曲线如何绘制之前,先和小伙伴一起理解这几个概念。
TP(True Positives),TN(True Negatives),FP(False Positives),FN(False Negatives)分别为阳性样本正确分类数量、阴性样本正确分类数量、阳性样本错误分类数量以及阴性样本错误分类数量。
阳性样本正确分类数量占阳性样本总数的比例TPR(即敏感性,Sensitivity),阴性样本正确分类的数量占阴性样本总数的比例FPR(即1-特异性,1-Specificity)
ROC曲线是以真阳性率(灵敏度)为纵坐标,假阳性率(1-特异度)为横坐标绘制的。每一个点都对应诊断试验的一个截点,我们将这些可能的点连接起来即可制作出经验ROC曲线(empirical ROC curve)。该方法可以帮助研究者简单、直观地分析诊断试验的临床准确性,选择更加合适的截点。
ROC曲线是常见的统计分析方法之一,目前广泛应用于医学诊断、生物信息学、数据挖掘和机器学习等研究中,用来评判分类、检测结果的好坏。ROC曲线可用于评价生物标记物(biomarker)的表现以及比较不同打分方法(scoring methods)。
那么,ROC曲线应该如何绘制呢?
加载pROC包
library(pROC) #需要提前安装数据准备
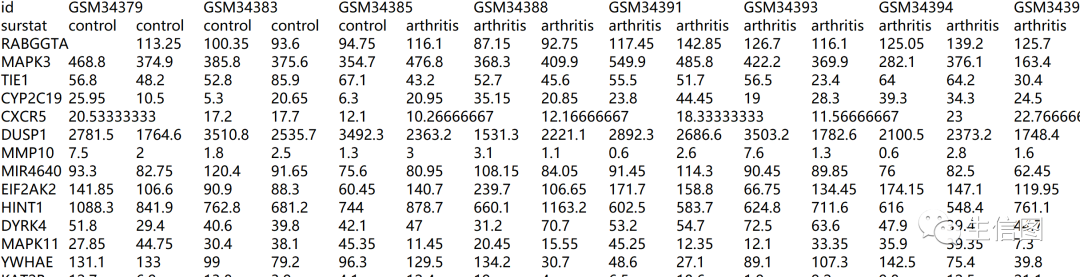
小图这里画两个基因演示
predicted <- read.table("data/数据/GSE1919生存分析.txt",sep= "t" ,header = T)
提取基因
rownames(predicted) = predicted$idpredicted = predicted[c("surstat","CD52","TNFRSF17"),]#根据自己的数据情况进行基因调整
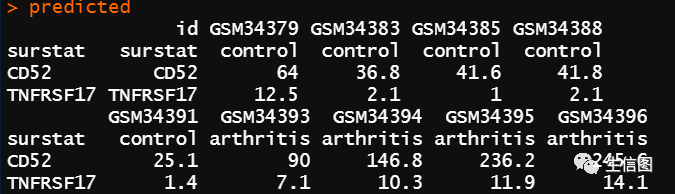
分组信息
actual = predicted[c("surstat"),]actual = t(actual)actual = actual[-1,]actual = gsub("control" , "0", actual)actual = gsub("arthritis" , "1", actual)actual = as.numeric(actual)
predicted = predicted[-1,-1]predicted = t(predicted)
将数据框中所有的列转化为数字型
a = predicteda <- apply(a, 2, as.numeric)rownames(a) = rownames(predicted)predicted = apredicted = data.frame(predicted)
ROC分析
roc = roc(actual, predicted$CD52, percent = TRUE, ci = TRUE) # ROC分析roc2 = roc(actual, predicted$TNFRSF17, percent = TRUE, ci = TRUE) # ROC分析
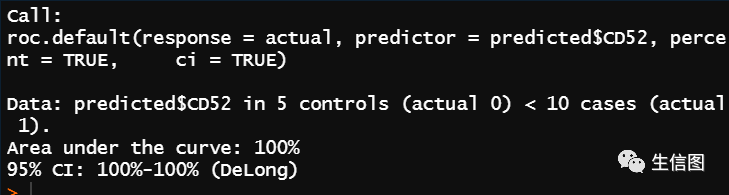
best = coords(roc, "best",ret = "all",transpose = F) #最佳分割点best2 = coords(roc2, "best",ret = "all",transpose = F) #最佳分割点outTab = data.frame(Gene = "CD52",AUC = round(roc$auc,3) ,"95% CI" = paste(round(roc$ci[c(1,3)],3), collapse = "-"),"Cutoff value" = round(best$threshold,3),Spensitivity = round(best$sensitivity,3),Specificity = round(best$specificity,3),check.names = FALSE)outTab2 = data.frame(Gene = "TNFRSF17",AUC = round(roc2$auc,3) ,"95% CI" = paste(round(roc2$ci[c(1,3)],3), collapse = "-"),"Cutoff value" = round(best2$threshold,3),Spensitivity = round(best2$sensitivity,3),Specificity = round(best2$specificity,3),check.names = FALSE)
绘制图片
rocobj1 <- plot.roc(actual, predicted$CD52,percent=TRUE, col="#69C5CE")rocobj2 <- lines.roc(actual, predicted$TNFRSF17, percent=TRUE, col="#B974AD")legend("bottomright", legend=c(paste0("CD52","-AUC", outTab$AUC , "(" , outTab$`95% CI` , ")"),paste0("TNFRSF17","-AUC", outTab2$AUC , "(" , outTab2$`95% CI` , ")")),col=c("#69C5CE","#B974AD"), lwd=2)
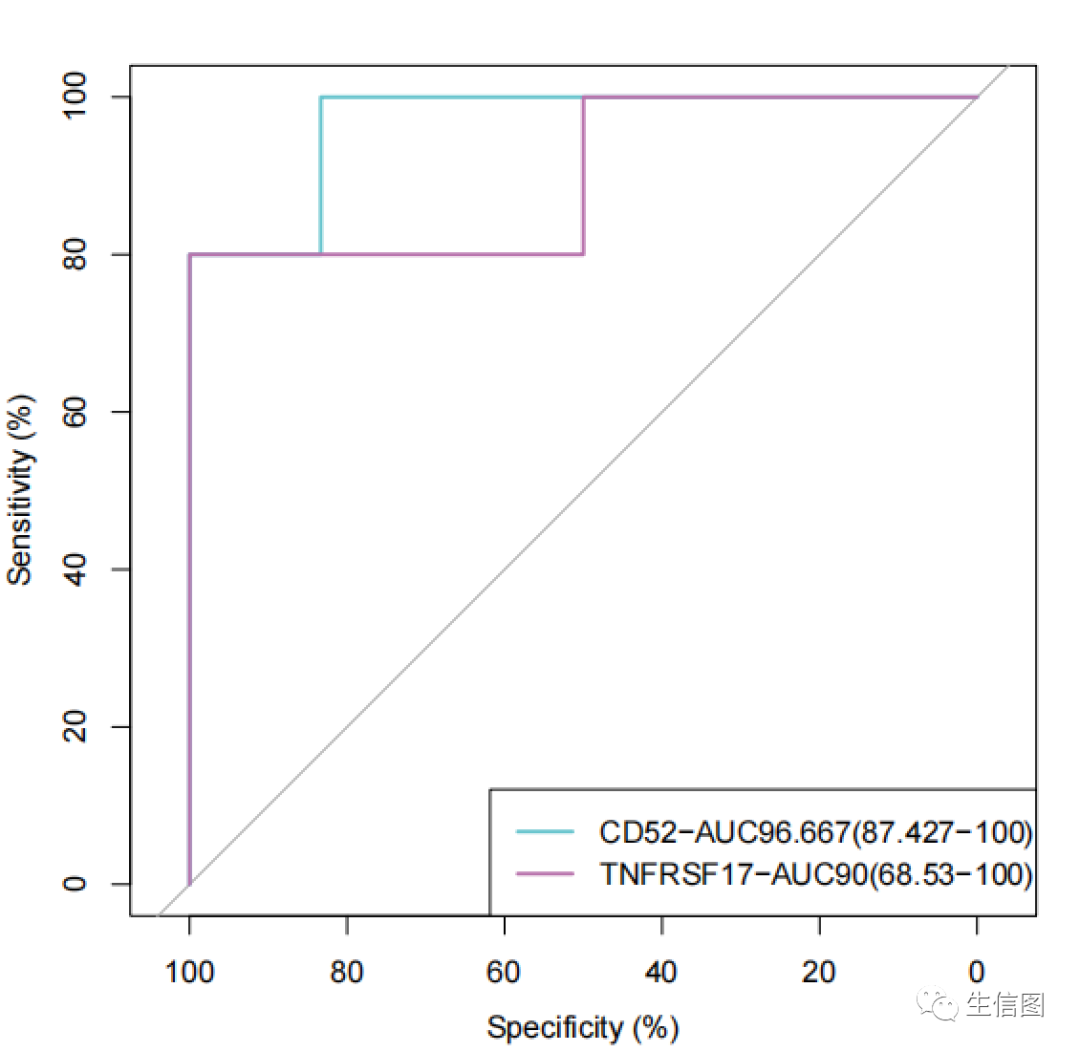
这样图片就画完了,是不是以为结束了~
现在开始图片解读
Q1:
ROC曲线有什么含义?
(1)ROC曲线越靠近左上角,则表示分类越好,正确率越高;
(2)ROC曲线下的面积AUC,介于0.1和1之间,作为数值可以直观的评价分类好坏,数值越大,表示越好,正确率越高(0.6以上为佳)。
Q2:
如何不使用R语言绘制ROC曲线?
小图和团队小伙伴们开发了一个在线的分析网站平台——云生信生物信息学平台(http://www.biocloudservice.com/home.html,免费的哦~)
小伙伴赶紧动手画起来吧~
我们下期见~
欢迎使用:云生信平台 ( http://www.biocloudservice.com/home.html)

|
往期推荐 |
|
|
|
|
|
|
👇点击阅读原文进入网址