「付费」【代码-10】只需三分钟学会,利用xCell进行免疫浸润分析
今天小果继续为大家分享免疫浸润分析的另一种分析方法-xCell
小果首先利用xCell进行免疫浸润分析,然后利用xCell分析结果跟感兴趣基因集表达矩阵进行相关性分析,绘制相关性热图,批量筛选出影响免疫浸润的候选基因,小果觉得这是一个非常值得学习的分析内容,强烈安利给小伙伴,接下来马上跟着小果开始今天的学习吧!小果已经迫不及待想开始今天的分享啦!哈哈。。。。
xcell包简介
在进行分析之前,小果首先为小伙伴简单介绍一下该包的功能和使用方法。xCell是基于ssGSEA的方法,可根据64种免疫细胞和基质细胞类型的基因表达数据进行细胞类型富集分析,Xcell的输入文件是来自人类混合样本的基因表达矩阵,如果基因表达数据来自微阵列,就不需要标准化。该方法适用于基因表达谱和RNA-seq数据,但不使用于单细胞数据。小果为大家附上xCell包的网址:https://github.com/dviraran/xCell,有深入学习了解的小伙伴可以自行前往学习哈!
安装需要的R包
install.packages("ggplot2")install.packages("tidyverse")install.packages("reshape2")install.packages("corrplot")install.packages("devtools")library(devtools)devtools::install_github('dviraran/xCell')
载入需要的R包
library(tidyverse)library(ggplot2)library(reshape2)library(corrplot)library(xCell)
结果文件展示
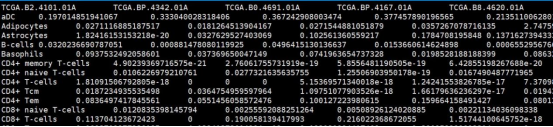
1. xCell.txt
该结果文件为计算的所有样本的64种免疫细胞的含量,行名为不同的免疫细胞类型,列名为样本名。
2. correlation.pdf
该结果图片为64种免疫细胞与目标基因集的相关性热图,横坐标表示基因名,纵坐标表示免疫细胞类型,用颜色来表示相关系数大小,星号来表示pvalue值,不同星号表示不同的pvalue值范围。1个星号表示pvalue值小于0.05大于0.01,两个星号表示pvalue值小于0.01,通过该热图可以很迅速的筛选出感兴趣的目标基因集中,那些基因对免疫浸润的影响大,最终筛选出候选基因。
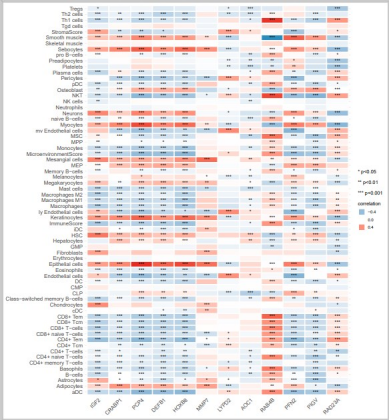
最终小果顺利完成了如何利用XCell包进行免疫浸润程度高低分析,绘制了感兴趣基因集与免疫浸润的相关性热图,通过该热图可以筛选出影响免疫浸润的基因,批量筛选出影响免疫浸润的候选基因。
如果需要完整的代码可以点击付费领取哦!今天小果的分享就到这里~
往期回顾

(扫码领取整理好了的输入文件、代码文件及示例结果)