颜值即正义,高颜值风险因子关联图看过来
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

-
今天小果为大家带来的是利用ggrisk包来绘制好看的风险因子关联图奥,一张图片展示风险得分曲线图,生存时间和风险得分散点图以及基因表达热图,绘制出的图片效果非常不错奥
接下来小果带着大家系统的学习一下该包的使用,从R包安装,示例数据展示和绘图,接下来小果开始今天的分享,如果觉得推文不错,点赞加关注奥。
安装需要的R包
install.packages("ggrisk")install.packages("survival")install.packages("rms")#载入需要的R包library(ggrisk)library(survival)library(rms)

读取数据
#数据文件第一列为生存时间,第二列为生存状态,其他列为基因。
data<-read.delim("data.txt")

代码展示
#构建cox模型fit <- cph(Surv(time,status)~ANLN+CENPA+GPR182+BCO2,data)#默认图形ggrisk(fit)
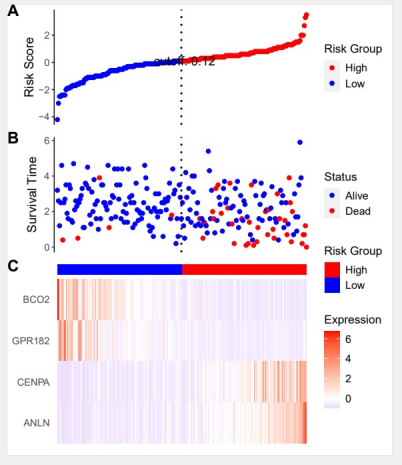
图A为风险得分按照从小到大的顺序排列 (此示例为根据中值分组);
图B为风险得分与生存时间的散点图,并按照结局将散点图分成红色和蓝色;
图C为基因表达量热图;
#调整风险得分的cutoff以及位置ggrisk(fit,cutoff.value='cutoff', #可选‘median’, ’roc’ or ’cutoff’cutoff.x = 150, #“cutoff”文本的水平位置cutoff.y = -1 #“cutoff”文本的垂直位置)
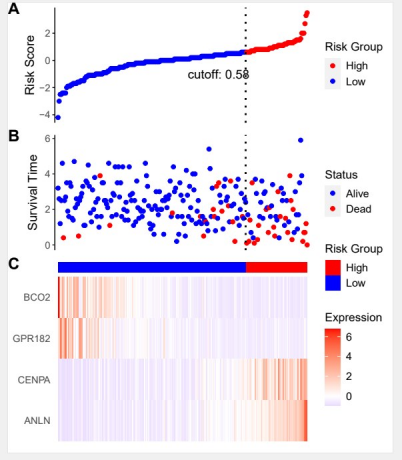
#指定热图展示基因grisk(fit, heatmap.genes=c('GPR182','CENPA','BCO2'))
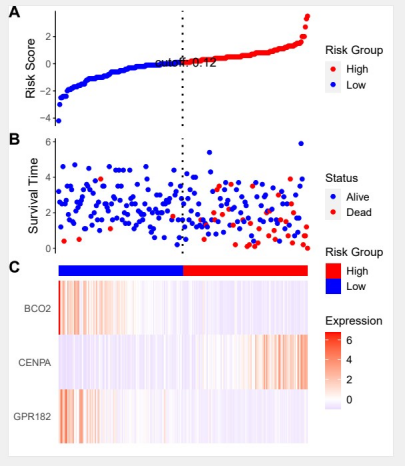
#调整细节和颜色pdf("TIME_heatmap.pdf",height=7,width=6, onefile = F)ggrisk(fit,code.highrisk = 'High Risk',#高风险标签,默认为 ’High’code.lowrisk = 'Low Risk', #低风险标签,默认为 ’Low’title.A.ylab='Risk Score', #A图 y轴名称title.B.ylab='Survival Time(year)', #B图 y轴名称,注意区分year month daytitle.A.legend='Risk Group', #A图图例名称title.B.legend='Status', #B图图例名称title.C.legend='Expression', #C图图例名称relative_heights=c(0.1,0.1,0.01,0.15), #A、B、热图注释和热图C的相对高度color.A=c(low='green',high='red'),#A图中点的颜色color.B=c(code.0='green',code.1='red'), #B图中点的颜色color.C=c(low='green',median='white',high='red'), #C图中热图颜色vjust.A.ylab=1, #A图中y轴标签到y坐标轴的距离,默认是1vjust.B.ylab=2 #B图中y轴标签到y坐标轴的距离,默认是2)dev.off()
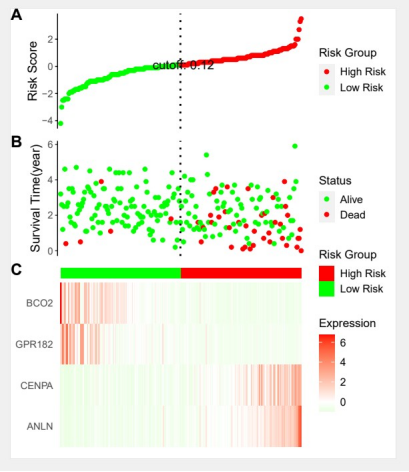
今天小果利用ggrisk进行了风险因子联动图的绘制,绘制效果非常不错奥,小伙伴们如果需要其他生存分析相关的分析或者图片绘制可以尝试本公司新开发的云平台生物信息分析小工具,零代码完成分析,非常方便奥。
云平台网址:http://www.biocloudservice.com/home.html
小果今天的分享就到这里,欢迎大家和小果一起讨论学习奥,下期再见奥。

微信号 | 18502195490
知乎 | 生信果
点击“阅读原文”立刻拥有
↓↓↓