「付费」【代码-3】与小果一起探索,免疫浸润与多个基因相关性热图绘制
收录于话题
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

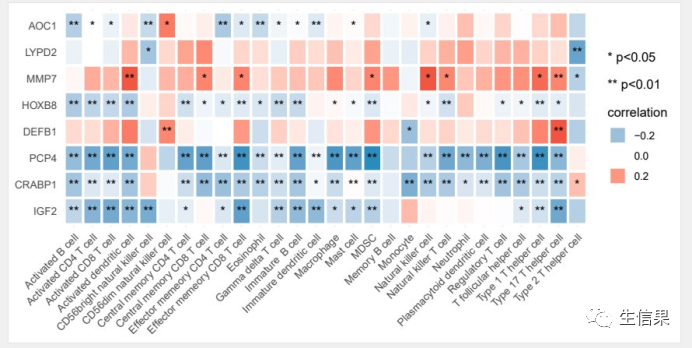
小果大讲堂又来啦!今天小果想利用通过ssGSEA算法获得的免疫细胞矩阵,以及通过基因表达矩阵提取感兴趣目标基因集表达量,来计算目标基因集与免疫浸润的相关性并计算pvalue,然后将结果用热图的形式表现出来,评价基因对免疫浸润的影响,筛选影响免疫浸润的候选基因,该分析在生信分析文章中出现的比例很高,安利小伙伴可以学习一下该分析,使自己的文章内容变得更加丰富多彩一点,话不多说,赶紧跟着小果开启今天的充电学习时间吧!
ssGSEA算法简单介绍
在进行高颜值的相关性热图绘制之前,小果想带着小伙伴了解一下免疫浸润ssGSEA分析方法的原理和分析结果包含的内容,因为只有在获得ssGSEA结果文件我们才能进行与感兴趣基因集相关性分析,接下来小果为大家来做一个简单的介绍吧!
ssGSEA单样本基因富集分析,是针对单个样本无法做GSEA而设计的,它可以使用GSVA R包来实现,目前ssGSEA常被用来评估肿瘤免疫细胞浸润程度,今天小果就不展示如何利用该包进行ssGSEA分析,有这方面分析需要的小伙伴可以联系小果。
准备需要的R包
install.packages("ggplot2")install.packages("tidyverse")install.packages("ggrepel")install.packages("corrplot")install.pachages("reshape2")
如果需要完整的代码可以点击付费获取哦!今天小果的分享就到这里,如果小伙伴有其他数据分析需求,可以尝试本公司新开发的生信分析小工具云平台,季代码完成分析,非常方便奥!
云平台网址为: http://www.biocloudservice.com/home.html,包括根据gsva结果绘制绘制两基因相关性 图(http://www.biocloudservice.com/405/405.php),根 据 gsva 筛 选高相关性基因(http://www.biocloudservice.com/404/404.php)等小工具,欢迎小伙伴们来尝试哟!
(扫码领取整理好的输入文件,代码文件及示例结果)