3分钟学会t-SNE降维!教你秒杀高维生物数据!
{ 点击蓝字,关注我们 }
t-SNE(t-distributed stochastic neighbor embedding)是一种非线性降维算法,被广泛用于高维数据可视化。它通过将高维数据映射到低维空间中,并尽可能地保留原始数据中的局部结构来实现这一目标。
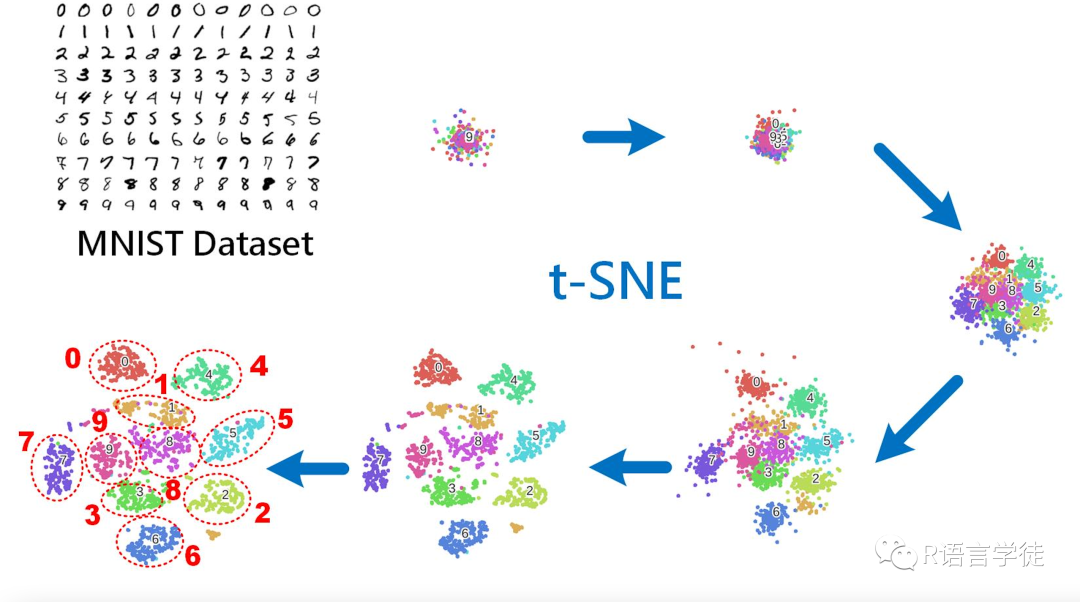
t-SNE算法的基本思想是,将高维数据视为在高维空间中的概率分布,并在低维空间中对这个概率分布进行重新建模,以使得在低维空间中相似的数据点在高维空间中也更有可能是相似的。t-SNE算法使用随机梯度下降法来最小化高维空间中数据点之间的Kullback-Leibler(KL)散度和低维空间中的KL散度之间的差异。
在R中,可以使用Rtsne包中的Rtsne()函数来运行t-SNE算法。该函数的参数包括高维数据矩阵、降维后的维数、困惑度等。Rtsne是一个R语言中的高性能t-SNE降维算法实现,它是基于Barnes-Hut-SNE算法的C++实现封装的。Rtsne包提供了一种快速且可扩展的方法来对高维数据进行降维,以便进行可视化或其他分析。
Rtsne包提供了一个名为Rtsne()的主要函数,它可以使用默认设置或用户定义的参数运行t-SNE算法。该函数的参数包括:
X:高维数据矩阵,必选参数。
dims:降维后的维数,默认为2。
initial_dims:在运行PCA之前保留的初始维数,默认为50。
perplexity:困惑度参数,用于控制每个数据点周围的邻居数量,默认为30。
theta:控制Barnes-Hut近似的精度,默认为0.5。
max_iter:最大迭代次数,默认为1000。
verbose:是否显示详细输出信息,默认为FALSE。
Rtsne包还提供了其他一些函数,例如tsne_pca()函数可以在运行t-SNE之前使用PCA对数据进行降维,以提高算法的效率;tsne_fit()函数可以直接使用t-SNE算法对数据进行拟合,而不使用PCA进行预处理。
接下来就让小师妹以乳腺癌患者活检数据集为例手把手带你学会t-SNE降维吧!
乳腺癌数据库包含了到1992年7月15日为止699名病人的乳腺肿瘤活组织检查;九个属性中的每一个都以1到10的等级进行评分,结果也是已知的。它有699行11列。该数据框包含以下几列:ID样本代码编号(非唯一),V1团块厚度,V2细胞大小均匀性,V3细胞形状均匀性,V4边缘附着,V5单个上皮细胞大小,V6裸核(缺失16个值),V7淡色染色质,V8正常核仁,V9有丝分裂,“良性”或“恶性”类别。

首先需要导入数据和会使用到的包
library(Rtsne)library(MASS)library(ggplot2)data(biopsy)biopsy_matrix <- as.matrix(biopsy)
接下来进行数据预处理,删除有缺失值的行以及重复行。
set.seed(123)v<-biopsy_matrix[, 2:11]v[,1:9]<-apply(v[,1:9], 2, as.numeric)v <- v[complete.cases(v), ]duplicated_rows <- duplicated(v[,1:9])duplicate_indices <- which(duplicated_rows)v <- v[-duplicate_indices, ]v <- v[complete.cases(v), ]v1=v[,1:9]v1<-apply(v1, 2, as.numeric)
处理后得到的矩阵v1包含特征1-9,不包含样本ID、分类结果。
使用Rtsne进行t-SNE降维
tsne <- Rtsne(v1,pca = TRUE, perplexity = 10, theta = 0)接下来就可以实用ggplot对降维后的数据进行可视化啦
res <- as.data.frame(tsne$Y)res$Class =v[,10]length(unique(res$Class))ggplot(res, aes(x = V1, y = V2, color = Class)) + geom_point(size = 1.25) + labs(title = "t-SNE",x = "TSNE1", y = "TSNE2") + theme(plot.title = element_text(hjust = 0.5)) + theme_bw()
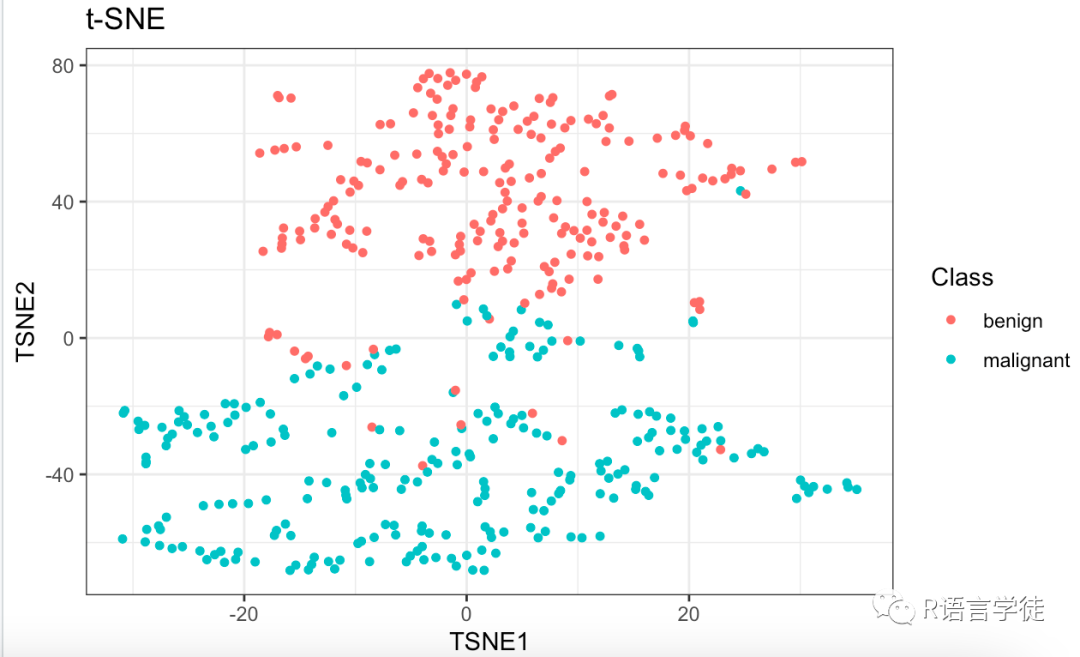
如图所示,降维后的数据分类结果非常不错,基本上能把良性和恶性区分开。
t-SNE算法是一种非线性降维算法,可用于高维数据的可视化和其他分析。在R中,可以使用Rtsne包来运行t-SNE算法,该包提供了一个快速、可扩展和易于使用的方法来对高维数据进行降维,以便进行可视化或其他分析。本文通过乳腺癌患者活检数据集的实例演示了如何使用Rtsne包进行t-SNE降维,并通过ggplot对降维后的数据进行可视化,展示了t-SNE算法在数据分类方面的良好表现。
这就是小师妹今天带来的降维新方法~你学会了吗?
更多实用方便的小工具在云生信平台等着你哦
http://www.biocloudservice.com/home.htm
E
N
D
