敲黑板!!小花手把手教你利用TCGA数据库做单基因甲基化位点分析!!
点击蓝字 关注我们
小伙伴们对TCGA数据库想必已经熟悉的不能再熟悉,我们可以利用TCGA的数据做很多预后的分析,而有的小伙伴筛选了基因就开始做常规的预后分析,做完又感觉少点东西,小花在这里教小伙伴一招,平时做的不多的东西-甲基化!
但是,我们都知道一个基因上有多个甲基化探针,所以甲基化数据及其大,而为了分析单个基因下载全部甲基化数据太亏了。因此,小花在这里选择从xena browser上下载单个基因甲基化数据,再利用R语言绘图分析!
那么接下来跟着小花去学习吧!
首先,我们需要把数据下载下来:
这是下载的网站,小花给小伙伴准备好了:https://xenabrowser.net/,然后点击VISUALIZATION,进入下面的页面:

小花在这里呢使用LUAD的数据给小伙伴做演示。
点击下方的to first variable
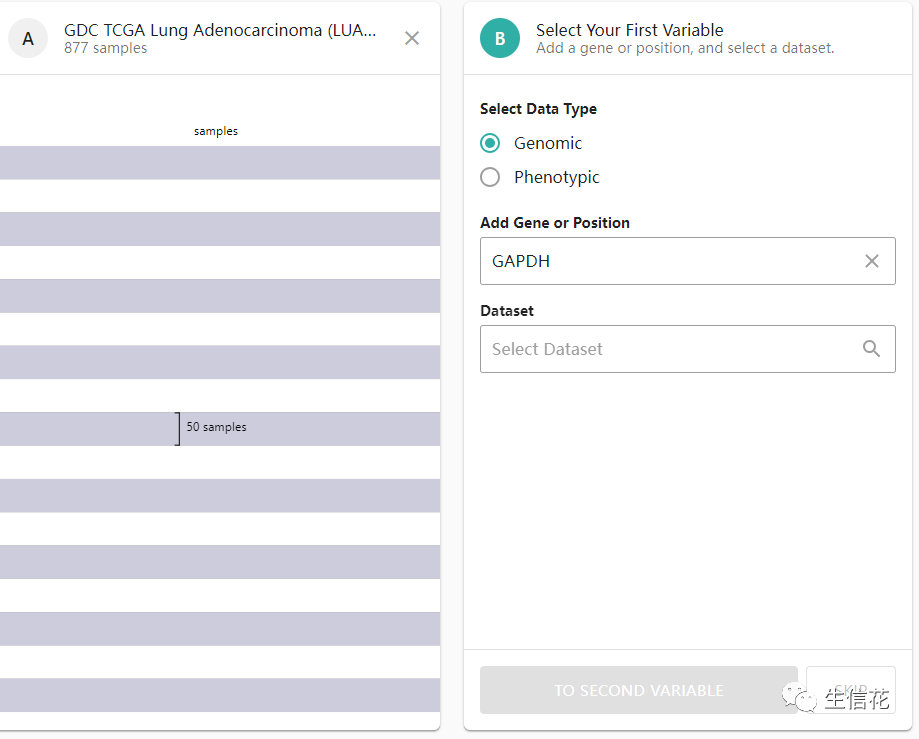
这里选择小伙伴自己的基因,小花在这里使用的是示例。注意Dataset数据集这里选择两个:一般是
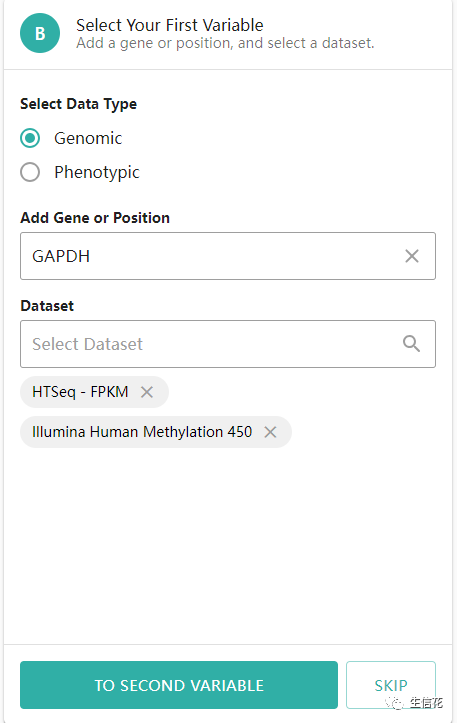
这两个,FPKM数据。接下来就是去下载啦:点击下一步就会进到这个页面
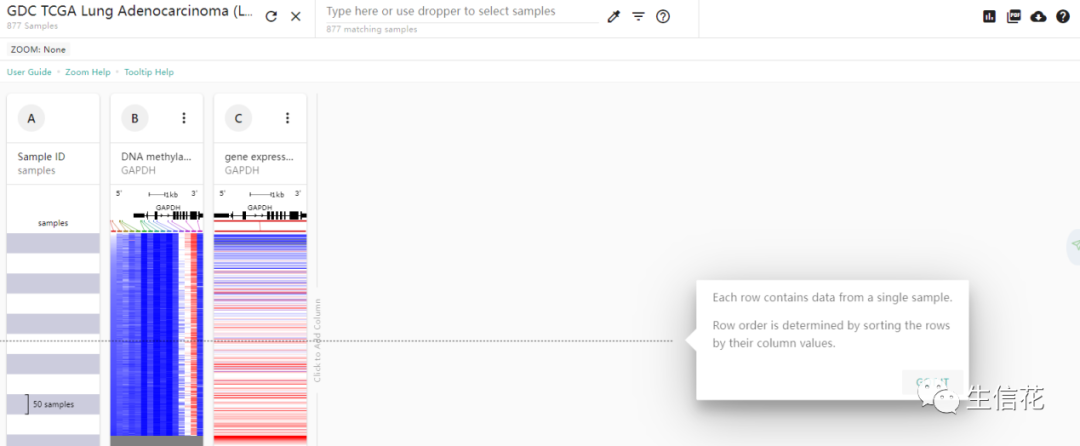
然后点击右上角的下载得到denseDataOnlyDownload.tsv 这样的文件
然后我们还需要一个文件 是450K甲基化芯片的注释信息,这个文件包含每个甲基化探针的位置等信息。这个可以从GEO或者xena下载,这里小花准备了一份放在连接里面illuminaMethyl450_hg38_GDC.txt
接下来就是重头戏了,我们用R语言可视化基因组的位置,这个内容主要包括启动子TSS区和基因boby内。
没有trackViewer包的小伙伴需要自己下载一下哦。
#BiocManager::install("trackViewer")library(trackViewer) #主要画图包methy <- read.delim("denseDataOnlyDownload.tsv") ##这里读取Xena下载的数据anno <- read.delim("illuminaMethyl450_hg38_GDC.txt") ##读取甲基化芯片注释信息ddd <- anno %>% plotly::filter(X.id %in% colnames(methy))SNP <- ddd$chromStartsample.gr <- GRanges("chr17", IRanges(ddd$chromStart, width=1, names=ddd$X.id)) # 设置棒棒图位置library(biomaRt)entrez=c("GAPDH")goids = getBM(attributes = c('hgnc_symbol', 'chromosome_name',"start_position","end_position","strand"),filters = 'hgnc_symbol',values = entrez,mart = ensembl)if (goids$strand== -1){features <- GRanges("chr17", IRanges(c(goids$start_position,goids$end_position+1), # 设置block起使位置width=c(goids$end_position-goids$start_position, 2000), # 设置block 的长度names=c("Gene","TSS"))) # 设置名字features$fill <- c("black","red") #块的颜色}else{features <- GRanges("chr17", IRanges(c(goids$start_position-2001,goids$start_position), # 设置block起使位置width=c(2000, goids$end_position-goids$start_position), # 设置block 的长度names=c("TSS","Gene"))) # 设置名字features$fill <- c("red","black") #块的颜色}sample.gr$color <- sample.int(length(SNP), length(SNP)) #棒子上面的球的颜色sample.gr$border <- sample(c("grey60", "grey50"), length(SNP), replace=TRUE) #棒子的颜色sample.gr$alpha <- 0.6 #设置透明度0-1之间,sample是生成100-200之间的随机数sample.gr$ce <- 0.5# sample.gr$label <- "C" #球内的字符# sample.gr$label.col <- "black" #球内的标签的颜色# features$height <- c(0.02, 0.05, 0.04) #块的高度# sample.gr$score <- sample.int(4, length(sample.gr), replace = TRUE) #设置球的数量lolliplot(sample.gr, features,yaxis = F,ylab = F) #yaxis设置不显示y轴
这样结果就出来啦!

下一步呢,我们还需要做相关性分析:
results <- psych::corr.test(methy$ENSG00000111640.13,methy[3:(ncol(methy)-1)])results <- rbind(data.frame(results[["r"]]),data.frame(results[["p"]])) %>% t() %>% as.data.frame()colnames(results) <- c("cor","p")r.cut <- 0.3results$color <- ifelse(results$cor> r.cut,"red",ifelse(results$cor < -r.cut,"green","black"))results$label <- ifelse(results$p > 0.05,"",ifelse(results$p >0.01,""ifelse(results$p>0.001,"","")))sample.gr <- GRanges("chr17", IRanges(ddd$chromStart, width=1, names= paste(ddd$X.id,results$label))) # 设置棒棒图位置sample.gr$border <- sample(c("grey60", "grey50"), length(SNP), replace=TRUE) #棒子的颜色sample.gr$alpha <- 0.6 #设置透明度0-1之间,sample是生成100-200之间的随机数sample.gr$cex <- 0.5sample.gr$color <- results$color #棒子上面的球的颜色sample.gr$label.col <- "black" #球内的标签的颜色# features$height <- c(0.02, 0.05, 0.04) #块的高度# sample.gr$score <- sample.int(4, length(sample.gr), replace = TRUE) #设置球的数量lolliplot(sample.gr, features,yaxis = F,ylab = F) #yaxis设置不显示y轴
结果:


小伙伴看到这样的分析是不是很心动呢。当然要课下要多多理解甲基化,不能盲目的跟做。多多理解代码的意思,小花在代码后面也标记了注释,小伙伴们要好好去试验。
快动手试试吧!!
欢迎使用:云生信 – 学生物信息学 (biocloudservice.com)
如果想用服务器可以联系微信:18502195490(快来联系我们使用吧!)

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

