一图胜千言:用ggridges画的山脊图,让你的数据说话

公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240218
需要租赁服务器的小伙伴可以扫码添加小果,此外小果还提供生信分析,思路设计,文献复现等,有需要的小伙伴欢迎来撩~

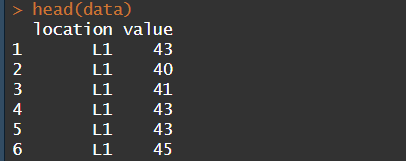
#安装与调用包install.packages('ggridges')library(ggridges)library(ggplot2)library(RColorBrewer)#读入数据data <- read.table('输入数据.txt',sep = 't',header = 1)head(data)
P1 <- ggplot(data, aes(x = value, y = location)) +geom_density_ridges(aes(fill = location))P1

P2 <- ggplot(data, aes(x = value, y = location)) +geom_density_ridges(aes(fill = location)) +scale_fill_manual(values = c("#00AFBB", "#E7B800", "#FC4E07",'black', 'red'))P2
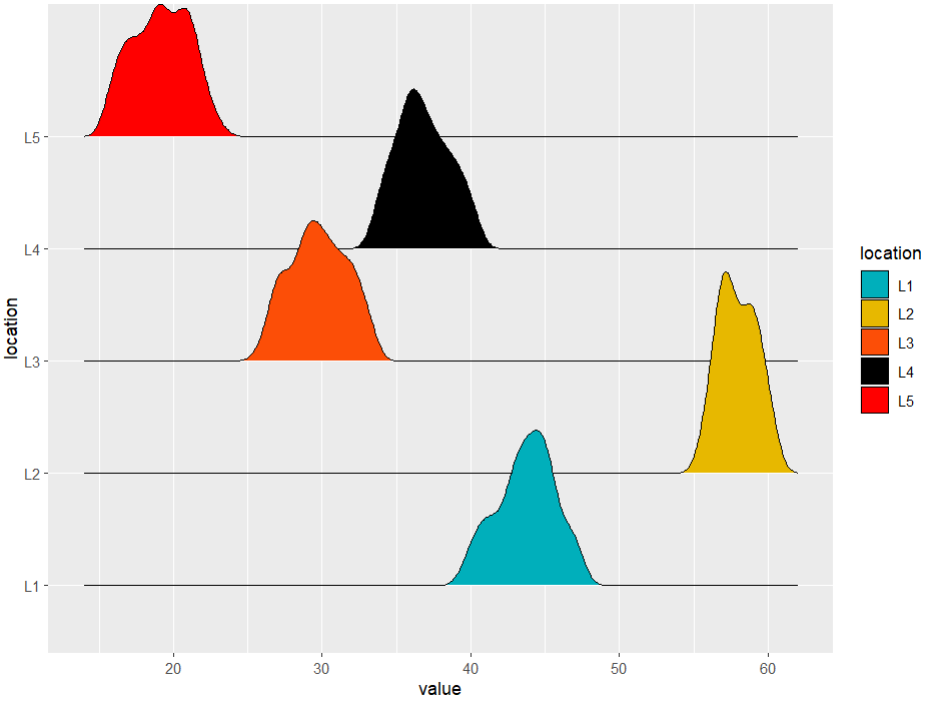
P3 <- ggplot(data, aes(x = value, y = location)) +geom_density_ridges(aes(fill = location)) +scale_fill_manual(values = c(brewer.pal(5,'Set3')))P3

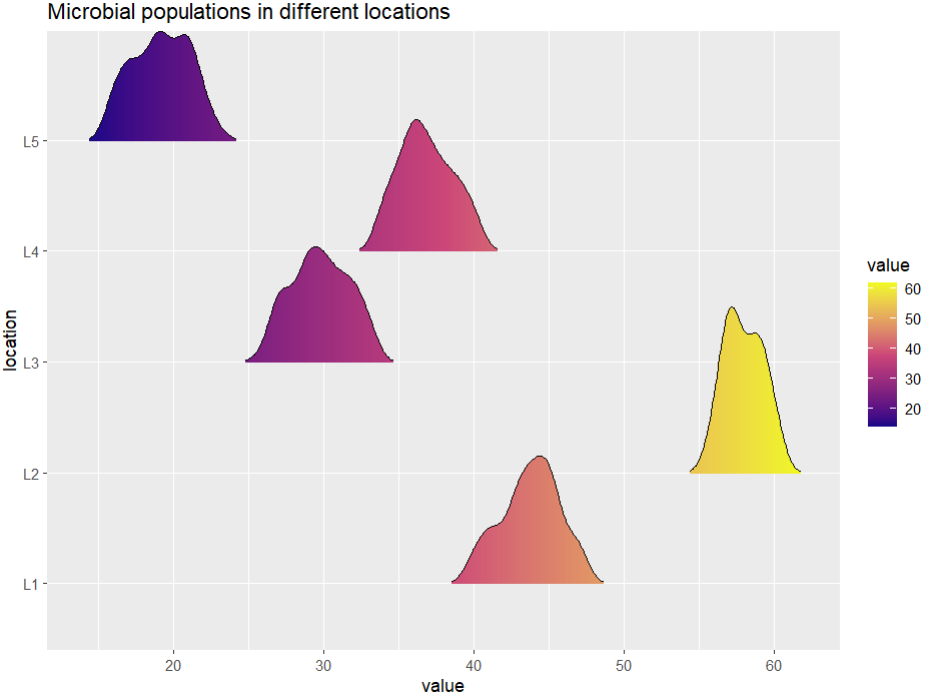
P4 <- ggplot(data,aes(x = value, y = location)) +geom_density_ridges_gradient(aes(fill = ..x..),scale = 1.5,rel_min_height = 0.01) +scale_fill_gradientn(colours = c("#0D0887FF", "#CC4678FF", "#F0F921FF"),name = "value")+labs(title = 'Microbial populations in different locations')P4
P5 <- ggplot(data,aes(x = value, y = location)) +geom_density_ridges_gradient(aes(fill = ..x..),scale = 1.5,rel_min_height = 0.01,jittered_points = TRUE,alpha = 0.5,point_size = 1.3) +scale_fill_gradientn(colours = c("#0D0887FF", "#CC4678FF", "#F0F921FF"),name = "value")+labs(title = 'Microbial populations in different locations') +theme_bw()P5

小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果


往期回顾
|
01 |
|
02 |
|
03 |
|
04 |