小花教学!!教你如何使用R包-ggrepel解决标签之间的重叠问题!!
点击蓝字 关注我们
小伙伴平时肯定有过这样的困惑,在样本比较多的时候,我们想在图形中添加标签,但是发现标签设定完了,特别容易出现标签遮盖的问题。尤其是在扩增子研究中,有很多的相同基因型,不得不都标记出来。
而我们在样本比较或者筛选时候又必须看清这些名字,筛选掉我们不需要或者有异常的标签样本。
这个时候,小花教小伙伴如何使用R包-ggrepel解决这个问题。让我们先简单了解一下这个来自ggplot2的辅助包-ggrepel这个专门处理覆盖问题的专家!我们使用它可以帮助我们在下游分析,样品筛选等更加方便高效!
ggrepel(https://github.com/slowkow/ggrepel)是发表在github上的开源包。
我们学习之前要先安装好这个包,小花在这里给大家几种下载的方式,
install.packages("ggrepel")#我们普通的安装,# 如果在R中,需要选择源或指定源install.packages("ggrepel", repo="http://cran.us.r-project.org")#或者我们安装最新的开发版本。install.packages("devtools", repo="http://cran.us.r-project.org")library(devtools)devtools::install_github("slowkow/ggrepel")安装完成,我们去学习吧!我们先看看正常情况下 geom_text()添加标签的时候是什么效果:library(ggplot2)#使用系统数据集mtcars演示ggplot(mtcars)+ geom_point(aes(wt, mpg), color="red")+geom_text(aes(wt, mpg, label=rownames(mtcars)))+theme_classic(base_size = 16)#小花在这里使用系统的数据集进展演示,小伙伴可以根据这个数据集去自习设置。
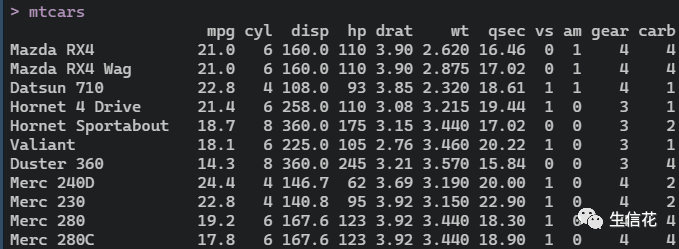
上述图是正常geom_text()的标签小花,是不是很多的标注都是重叠
效果非常的不好。接下来我们就去看看ggrepel包的效果。
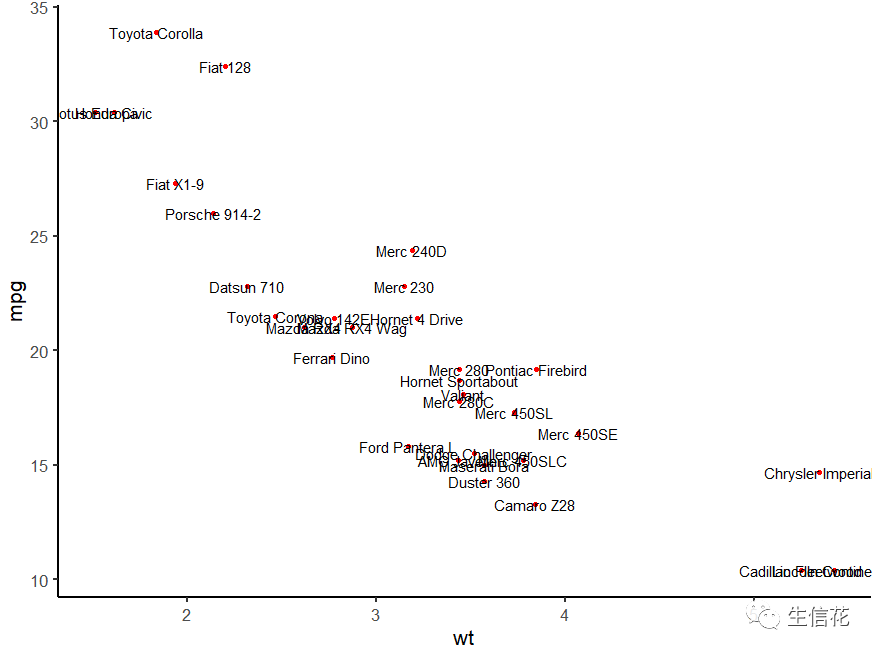
这里效果使用geom_text_re[el()函数去绘制,是不是看着很熟悉这个函数,他是基于geom_text()
#我们在调用ggrepel时候需要先运行ggplot2,谁让他是ggplot2中的辅助包呢。
library(ggrepel)set.seed(123)ggplot(mtcars)+ geom_point(aes(wt, mpg), color="red")+geom_text_repel(aes(wt, mpg, label=rownames(mtcars)))+theme_classic(base_size = 16)
让我们看一下效果:
这与上一张图是不是清楚多了,并且每一个标签都展示出来,没有重叠的。
当然,绘图的上限并不止这些,我们去添加点细节,让这张图变得更精美!
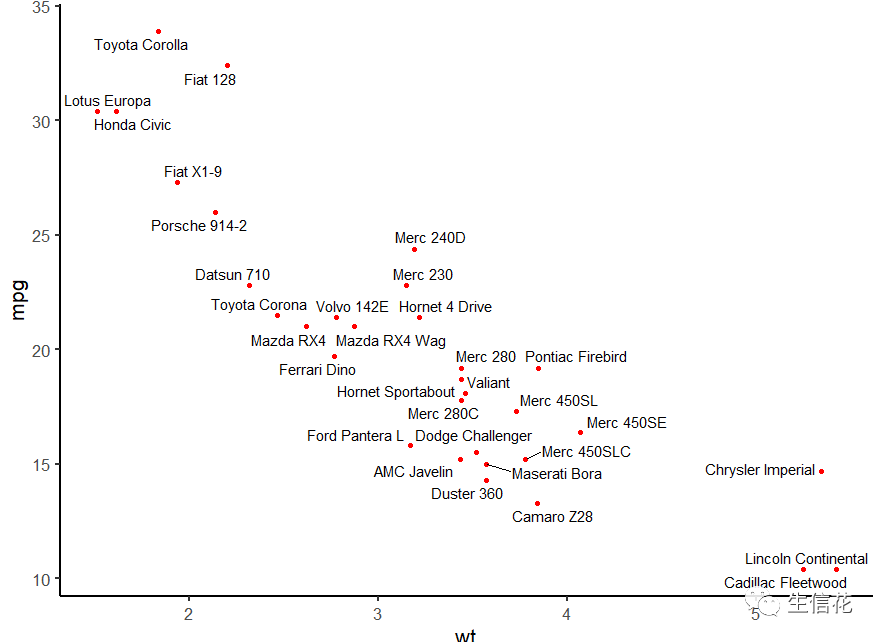
这个包还有geom_label_repel()函数,geom_label_repel()它是基于geom_label(),它可以将标签置于一个小方框中,这个函数可以防标签并且还可以添加背景色,我们去试试:
怎么样,是不是变得更好看
接下来就让小花带大家进行实战演习吧!
测试数据呢是扩增子的数据:
# 读入实验设计design = read.table("E:\生信花\design.txt", header=T, row.names= 1, sep="t")# 读入bray_curtis的距离矩阵bray_curtis = read.table("E:\生信花\bray_curtis_otu_table_css.txt", sep="t", header=T, check.names=F)
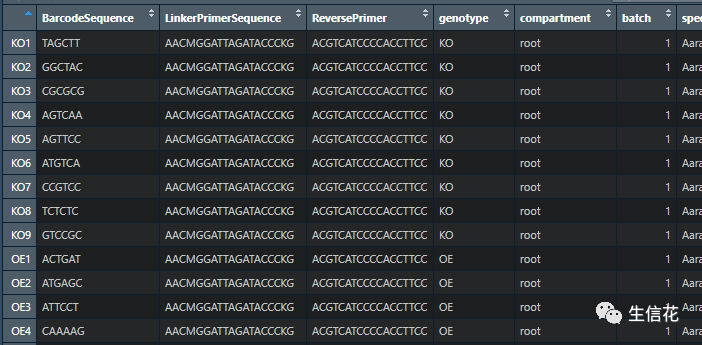
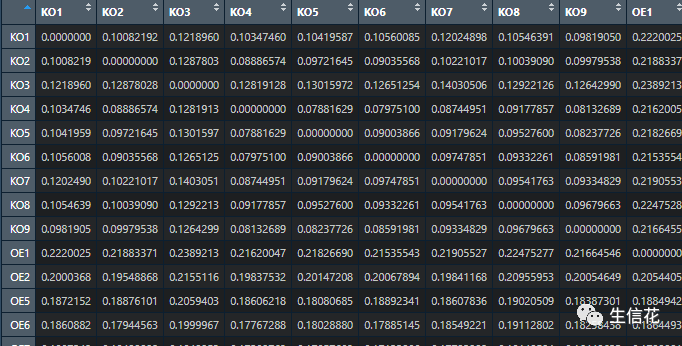
我们先对数据处理一下:
# 过滤数据并排序idx = rownames(design) %in% colnames(bray_curtis)sub_design = design[idx,]bray_curtis = bray_curtis[rownames(sub_design), rownames(sub_design)] # subset and reorder distance matrix# 将距离矩阵进行主坐标轴分析pcoa = cmdscale(bray_curtis, k=3, eig=T) # k is dimension, 3 is recommended; eig is eigenvaluespoints = as.data.frame(pcoa$points) # get coordinate string, format to dataframmecolnames(points) = c("x", "y", "z")eig = pcoa$eigpoints = cbind(points, sub_design[match(rownames(points), rownames(sub_design)), ])接下来我们绘制图片:# 绘制主坐标准轴的第1,2轴p = ggplot(points, aes(x=x, y=y, color=genotype)) +geom_point(alpha=.7, size=2) +labs(x=paste("PCoA 1 (", format(100 * eig[1] / sum(eig), digits=4), "%)", sep=""),y=paste("PCoA 2 (", format(100 * eig[2] / sum(eig), digits=4), "%)", sep=""),title="bray_curtis PCoA")p + geom_text(aes(x, y, label=rownames(points)))+ theme_classic()
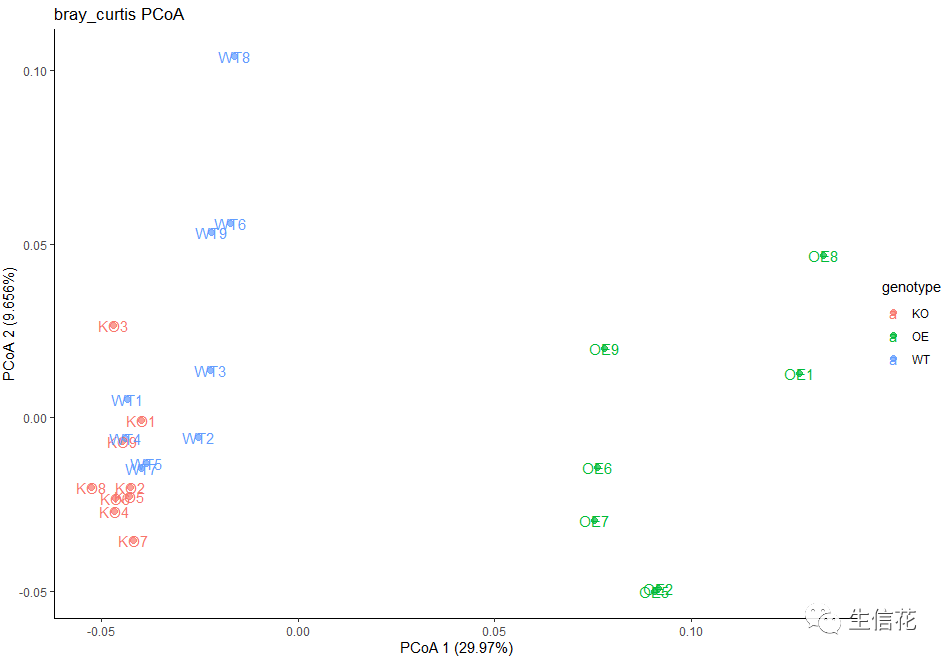
这个实例中,小伙伴是不是发现标签重叠了,所以我们用上面所学的进行修改:
首先我们用geom_text_repel合理的安排位置并添加样品名:
library(ggrepel)p + geom_text_repel(aes(x, y, label=rownames(points)))+ theme_classic()
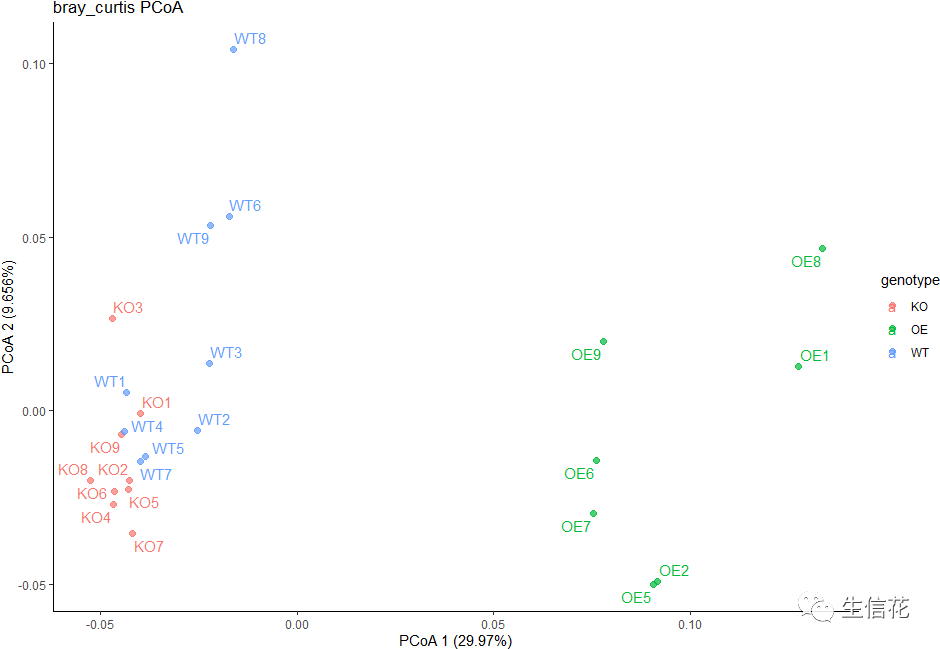
这样一来是不是好多了。
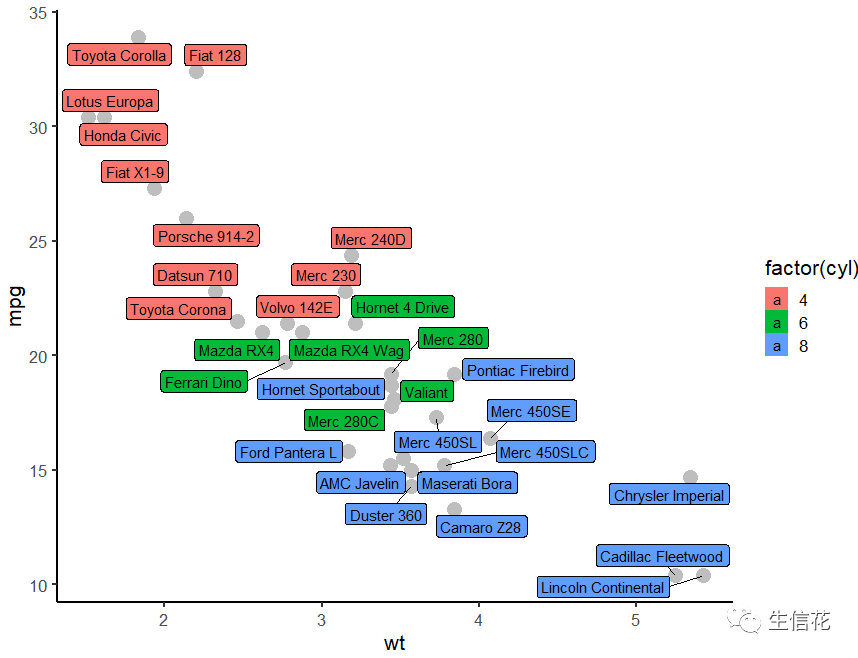
最后我们用geom_label_repel添加标签,然后设置背景填充色。
ggplot(points, aes(x=x, y=y)) +geom_point(alpha=.7, size=2) + geom_label_repel(aes(x, y, fill=factor(genotype), label=rownames(points)))+ theme_classic()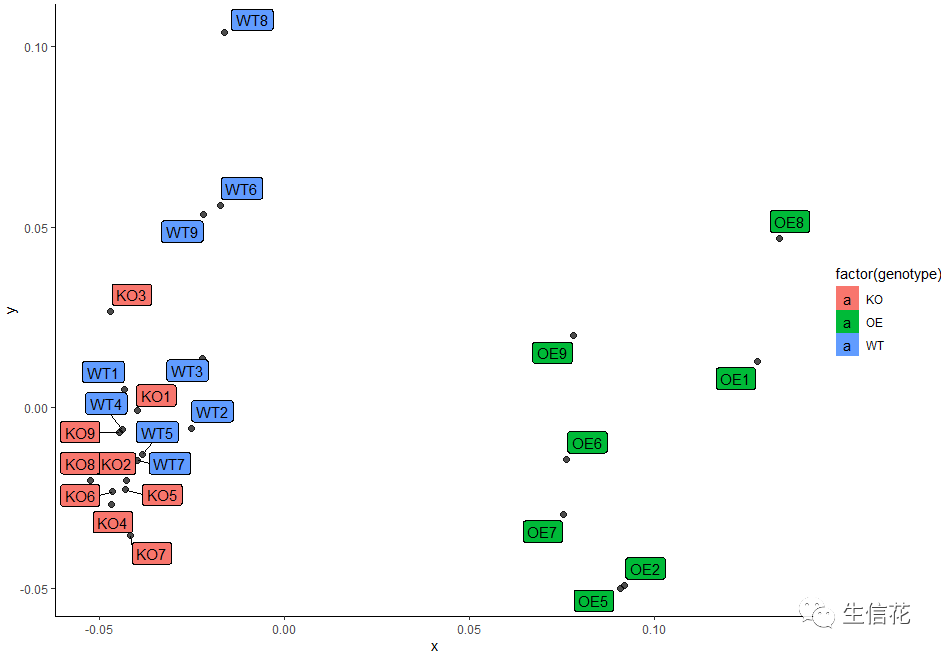
大功告成!这样也以来是不是很容易区分。小伙伴学会了吗,要注意多多理解代码的意义,这样才能绘制出自己想要的图片。小伙伴快去用自己的数据试试吧!
欢迎使用:云生信 – 学生物信息学 (biocloudservice.com)
如果想用服务器可以联系微信:18502195490(快来联系我们使用吧!)

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

