师妹教你R包EnhancedVolcano超详尽,让你的火山图喷发姿势更帅

点击蓝字,关注我们


一、简介
Mid-autumn Festival

火山图是可视化差异表达分析结果的非常常用的图。EnhancedVolcano会使你绘图的技巧更上一层楼。师妹手把手教你掌握EnhancedVolcano,看完绝对会用~

二、包的安装与加载
Mid-autumn Festival

# BiocManager::install('EnhancedVolcano')if (!requireNamespace('devtools', quietly = TRUE))install.packages('devtools')devtools::install_github('kevinblighe/EnhancedVolcano')library(EnhancedVolcano)
三、数据准备
Mid-autumn Festival

数据选择经典的airway数据集, 该数据集是人气道平滑肌细胞(airway smooth muscle cells )的RNA-seq测序的原始counts数据。在实验中针对75名哮喘患者的气道平滑肌细胞分别用地塞米松、沙丁胺醇、地塞米松+沙丁胺醇处理或未处理。关于实验设计的更多信息可以查看PubMed entry 24926665 ,原始数据 GEO entry GSE52778.
library(airway)library(magrittr)data('airway')# %<>%复合赋值操作符, 功能与 %>% 基本是一样的,但多了一项额外的操作,就是把结果写到左侧对象。# 对dex列进行relevel,再把relevel后的结果赋值到airway$dex。airway$dex %<>% relevel('untrt')
一开始的airway处理信息,是trt在前untrt在后

通过relevel()函数讲untrt 换到trt前面,便于操作

四、使用DESeq2创建两组差异分析结果
Mid-autumn Festival

使用DESeq2进行差异表达,以创建两组结果。res1是处理组与对照组对比,res2是两种细胞间对比。
#使用DESeq2创建两组差异分析结果library('DESeq2')dds <- DESeqDataSet(airway, design = ~ cell + dex)dds <- DESeq(dds, betaPrior=FALSE)res1 <- results(dds,contrast = c('dex','trt','untrt'))res1 <- lfcShrink(dds,contrast = c('dex','trt','untrt'), res=res1, type = "ashr")res2 <- results(dds,contrast = c('cell', 'N061011', 'N61311'))res2 <- lfcShrink(dds,contrast = c('cell', 'N061011', 'N61311'), res=res2,type = "ashr" )
查看下数据结果
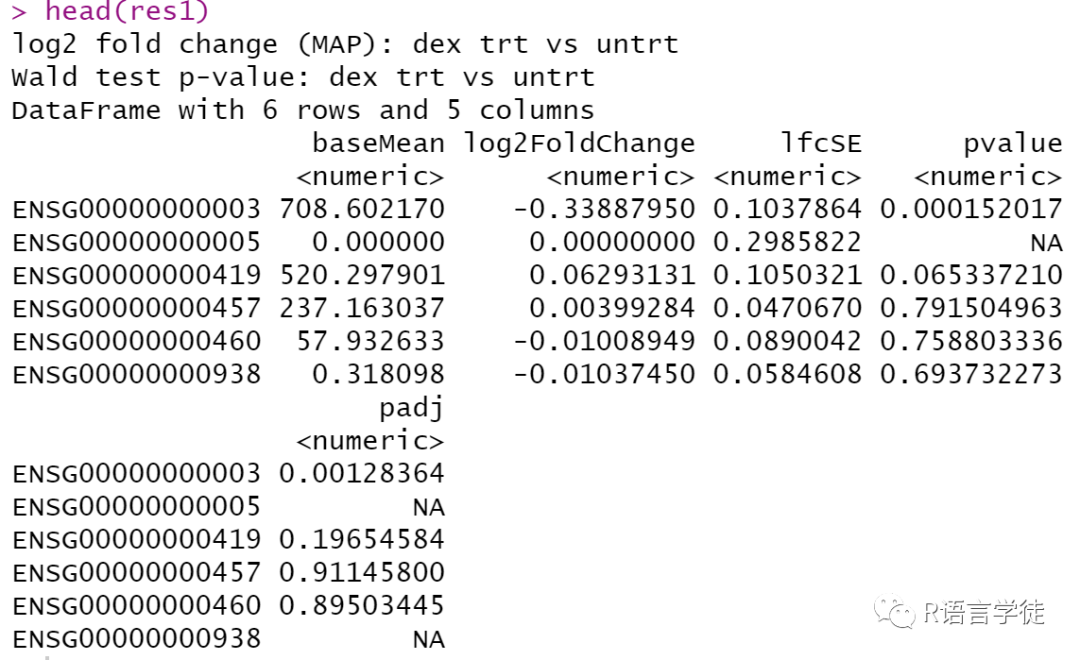
五、绘制火山图
Mid-autumn Festival


1.绘制最基本的火山图
Mid-autumn Festival

首先调用EnhancedVolcano函数。对最基本的火山图来说只需要,以此填入测试结果的矩阵,基因的名称,log2FoldChange,pvalue或者padjust(经过调整的p值),以及x轴的坐标范围。就可以轻松绘制出美观的火山图了。
EnhancedVolcano(res1,
# 基因名字lab = rownames(res1),x = 'log2FoldChange',y = 'pvalue',xlim = c(-5, 8))
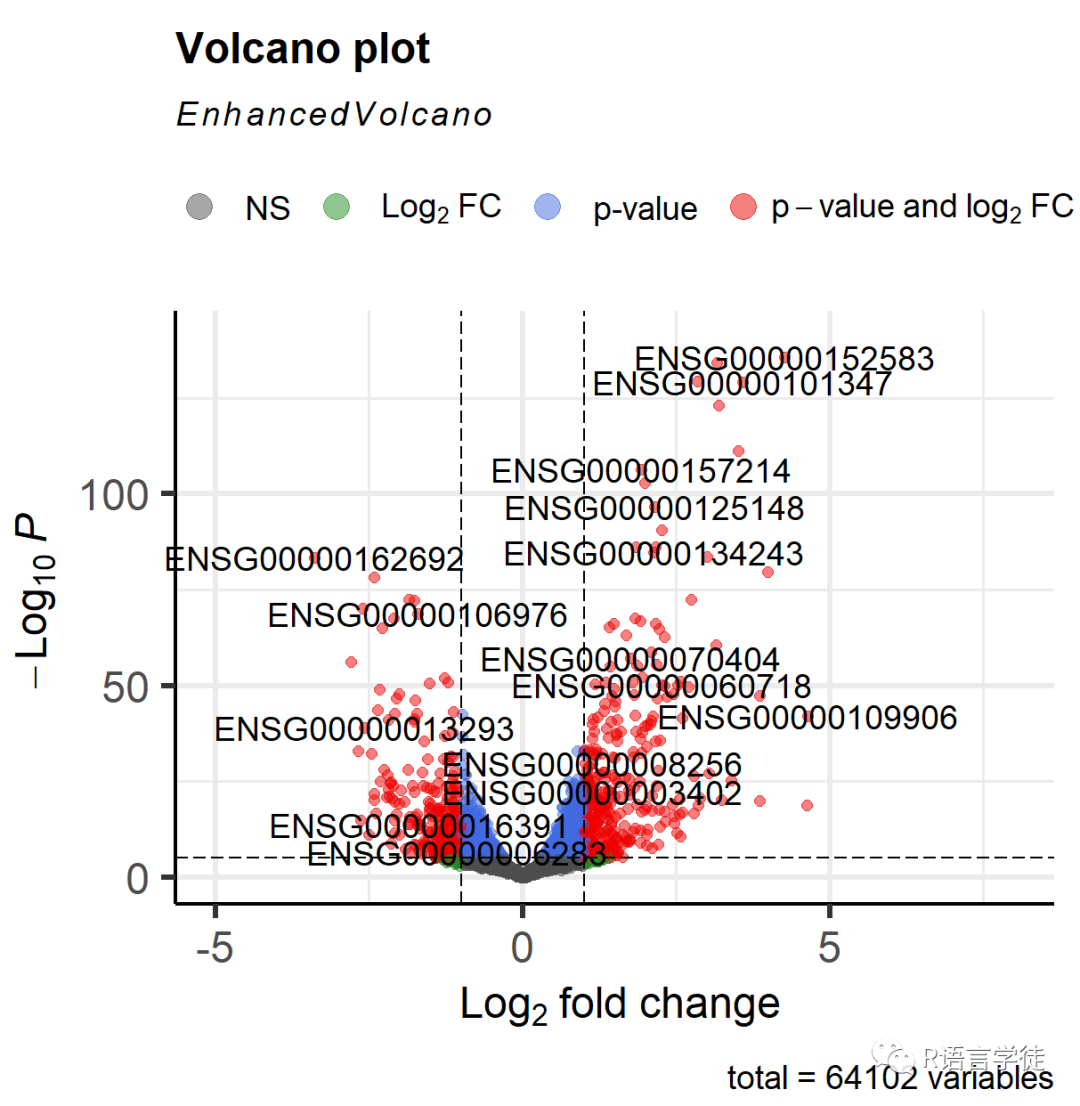
怎么样是不是很棒~

EnhancedVolcano的强大当然不止这些。下面师妹带大家继续探索它的强大功能。
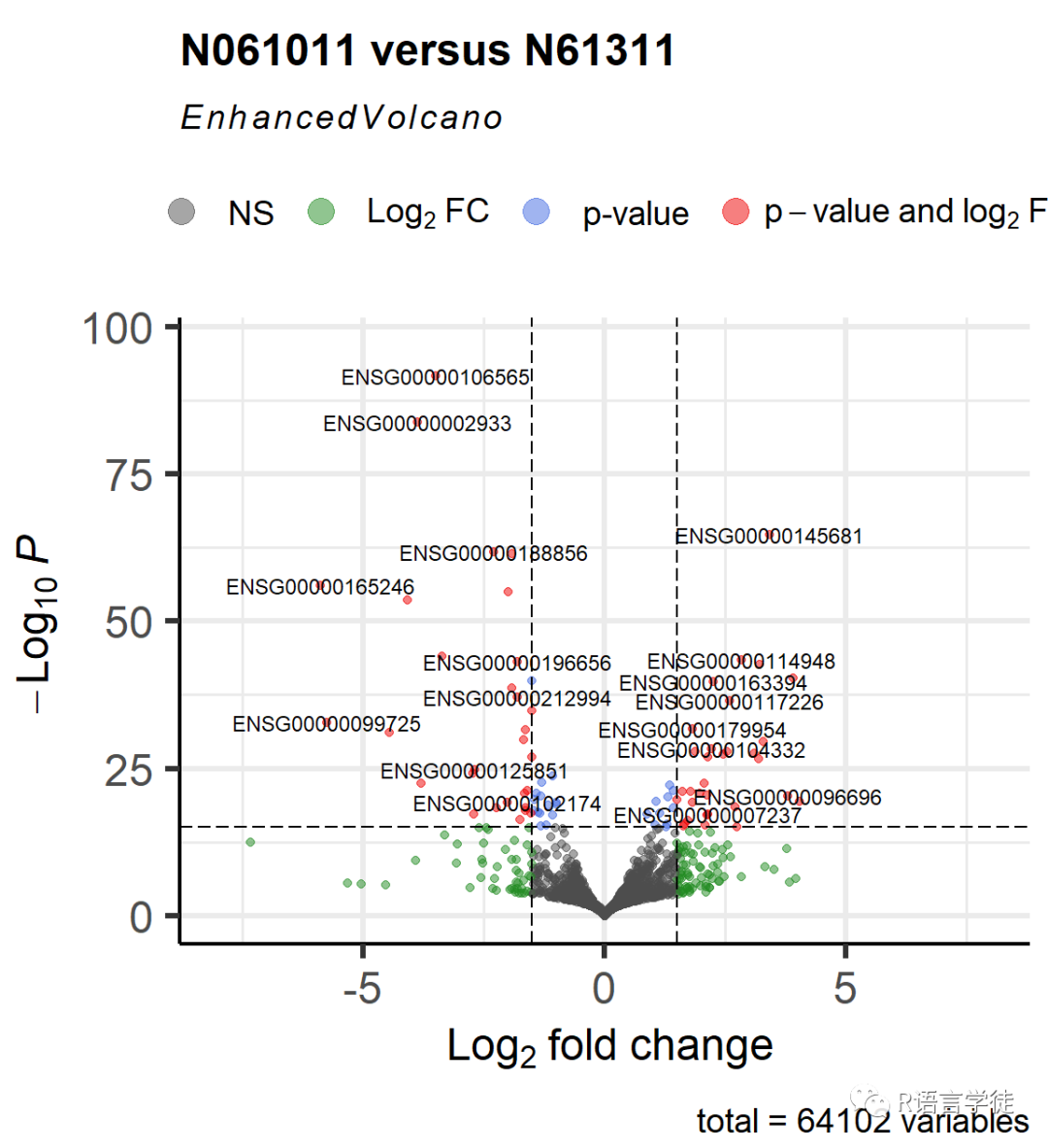
师妹为了更好的演示,进一步调整点和标签大小,来帮助改善火山图的清晰度,以确保更多的转录本进入差异分析中。
log2FC的默认cut-off值是 > | 2 |; P值的默认cut-off值为10e-6。我们这里将cut-off值设置为 > | 1.5 |
EnhancedVolcano(res2,
lab = rownames(res2),x = 'log2FoldChange',y = 'pvalue',xlim = c(-8, 8),title = 'N061011 versus N61311',pCutoff = 10e-16,FCcutoff = 1.5,pointSize = 1.5,labSize = 3.0)

3.调整点的颜色及透明度
Mid-autumn Festival

你可能不喜欢默认的配色,没关系~师妹教你如何更改。这次我们让筛选出来差异的转录本为红色,其他一切都是黑色的,进一步凸显差异基因。还可以通过调整’alpha’的值,:alpha 为1 = 100%不透明; alpha 为0 = 100%透明。
设置点颜色的参数col从左到右依次为无显著,倍数大(分割线左下、右下),pValue小 (分割线中上), 显著差异。我们将除了显著差异外的点都设置为black。
EnhancedVolcano(res2,
lab = rownames(res2),x = 'log2FoldChange',y = 'pvalue',xlim = c(-8, 8),title = 'N061011 versus N61311',pCutoff = 10e-16,FCcutoff = 1.5,pointSize = 1.5,labSize = 3.0,# 无显著,倍数大(左下、右下),P小 (中上), 显著差异# > DEFAULT = c("grey30", "forestgreen", "royalblue", "red2").col=c('black', 'black', 'black', 'red3'),colAlpha = 1)


4.调整点的形状
Mid-autumn Festival

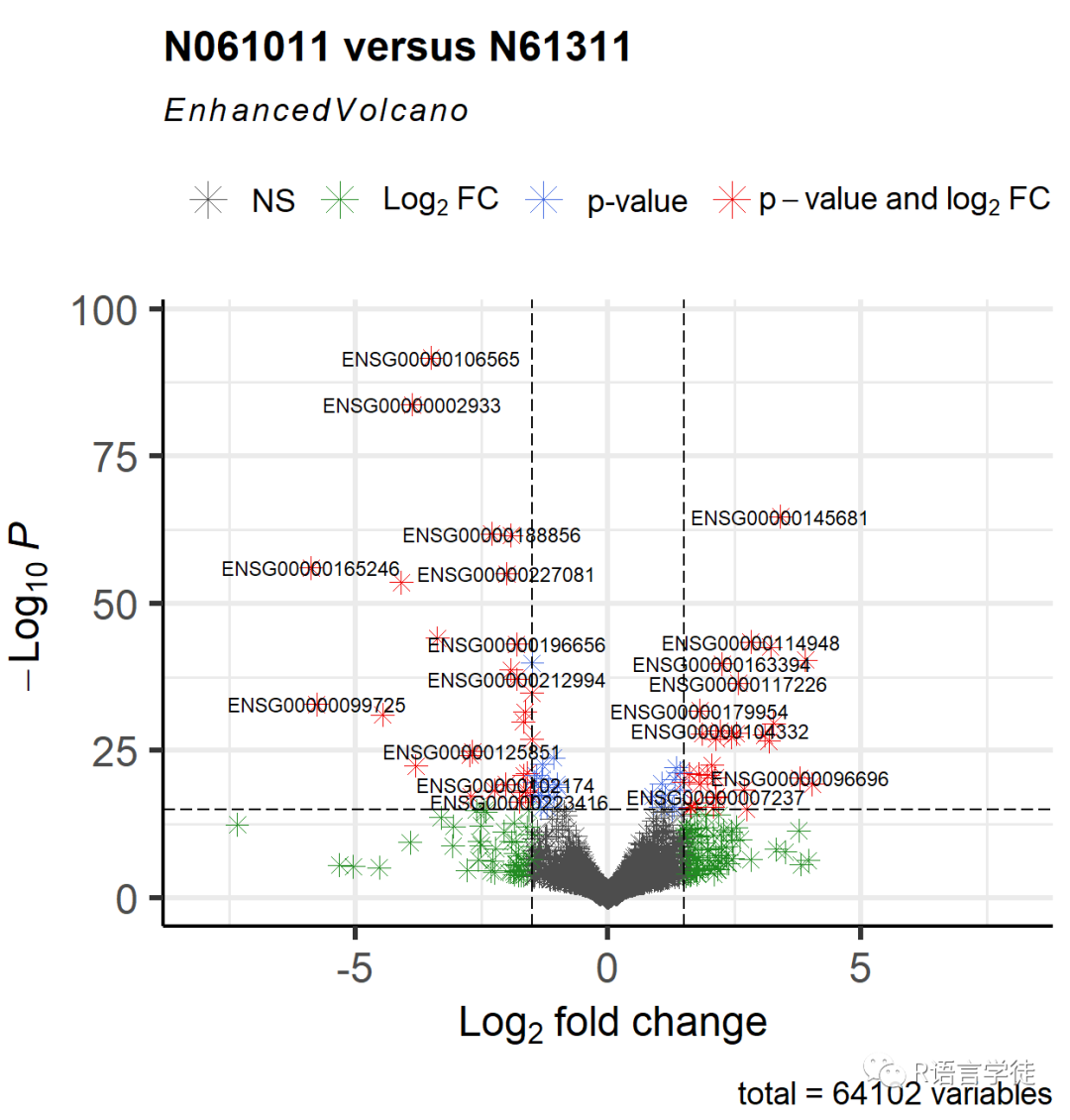
我们还可以通过shape()函数指定形状。可以是所有点的单一数值,也可以是与 legendLabels 指定的默认 4 个图例标签相对应的 4 个数值。
EnhancedVolcano(res2,
lab = rownames(res2),x = 'log2FoldChange',y = 'pvalue',xlim = c(-8, 8),title = 'N061011 versus N61311',pCutoff = 10e-16,FCcutoff = 1.5,pointSize = 3.0,labSize = 3.0,shape = 8,colAlpha = 1)
EnhancedVolcano(res2,
lab = rownames(res2),x = 'log2FoldChange',y = 'pvalue',xlim = c(-8, 8),title = 'N061011 versus N61311',pCutoff = 10e-16,FCcutoff = 1.5,transcriptPointSize = 2.0,transcriptLabSize = 3.0,# 同上面col# 无显著,倍数大(左下、右下),P小 (中上), 显著差异shape = c(1, 4, 23, 25),colAlpha = 1)


5.调整火山图中的线
Mid-autumn Festival

cut-off线可以通过以下函数和参数进行修改。“cutoffLineType”:“blank”, “solid”, “dashed”, “dotted”, “dotdash”, “longdash”, “twodash”;分别有七种不同的形态
cutoff线的颜色和粗细可以通过 ‘cutoffLineCol’ 和 ‘cutoffLineWidth’进行修改。
若是不需要cut-off线,可以设置“cutoffLineType=“blank” or cutoffLineWidth=0.”
也可以通过参数‘hline’ and ‘vline’ 显示其他的cut-off线;并设置不同的颜色形状。
EnhancedVolcano(res2,
lab = rownames(res2),x = 'log2FoldChange',y = 'pvalue',xlim = c(-6, 6),title = 'N061011 versus N61311',pCutoff = 10e-12,FCcutoff = 1.5,pointSize = 1.5,labSize = 3.0,colAlpha = 1,# 取消cutoff线cutoffLineType = 'blank',cutoffLineCol = 'black',cutoffLineWidth = 0.8,hline = c(10e-12, 10e-36, 10e-60, 10e-84),hlineCol = c('grey0', 'grey25','grey50','grey75'),hlineType = 'longdash',hlineWidth = 0.8,gridlines.major = FALSE,gridlines.minor = FALSE)
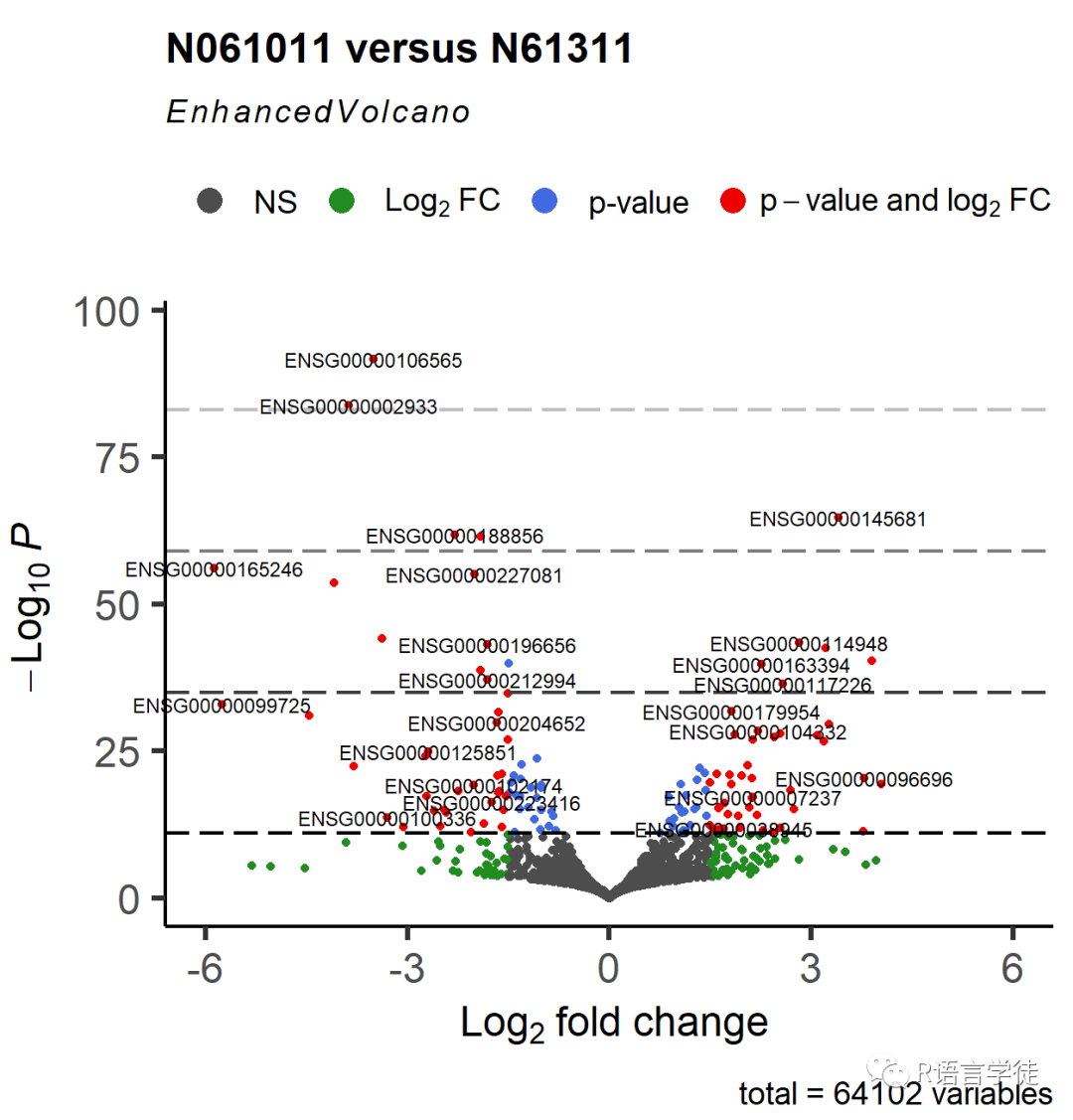

6.调整图例的位置和大小
Mid-autumn Festival

可以用legendLables, legendPosition, legendLabSize, legendIconSize分别调整图例的位置形状大小。
EnhancedVolcano(res2,
lab = rownames(res2),x = 'log2FoldChange',y = 'pvalue',xlim = c(-6, 6),pCutoff = 10e-12,FCcutoff = 1.5,cutoffLineType = 'twodash',cutoffLineWidth = 0.8,pointSize = 1.5,labSize = 3.0,colAlpha = 1,legend=c('NS','Log (base 2) fold-change','P value', 'P value & Log (base 2) fold-change'),legendPosition = 'right',legendLabSize = 16,legendIconSize = 5.0)
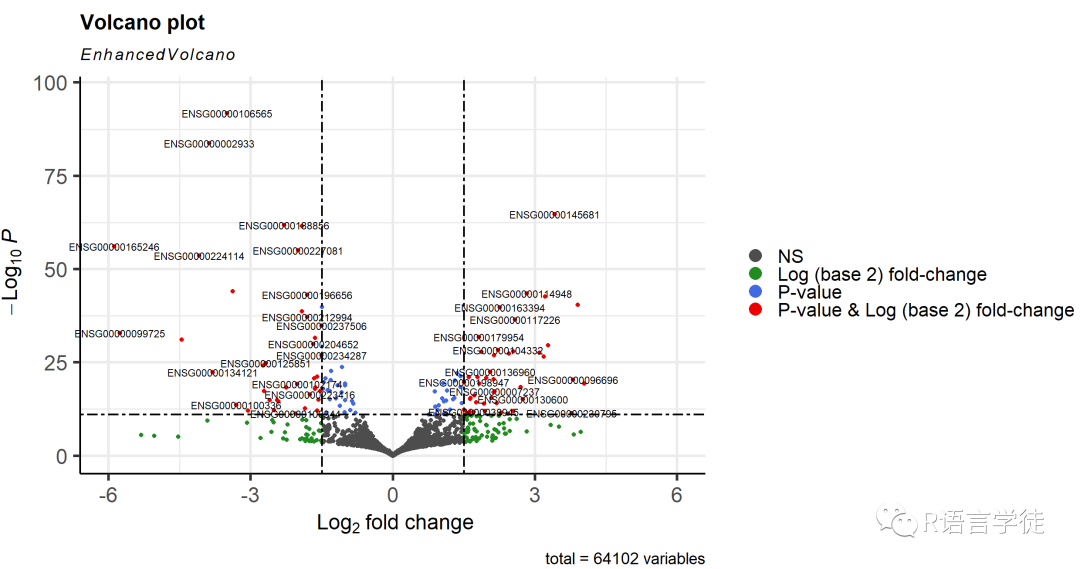

7.标注关键的转录本
Mid-autumn Festival

更多情况下,科研绘图,我们一般只想标注关键我们关注的的转录本。该如何标记呢?我们可以通过selectLab函数来进行标记。注意只有筛选后的才能标记。
EnhancedVolcano(res2,
lab = rownames(res2),x = 'log2FoldChange',y = 'pvalue',## 标记目标基因selectLab = c('ENSG00000106565','ENSG00000187758'),xlim = c(-6,7),xlab = bquote(~Log[2]~ 'fold change'),pCutoff = 10e-14,FCcutoff = 2.0,transcriptPointSize = 3.0,transcriptLabSize = 5.0,shape = c(4, 35, 17, 18),colAlpha = 1,legend=c('NS','Log (base 2) fold-change','P value','P value & Log (base 2) fold-change'),legendPosition = 'bottom',legendLabSize = 14,legendIconSize = 5.0)
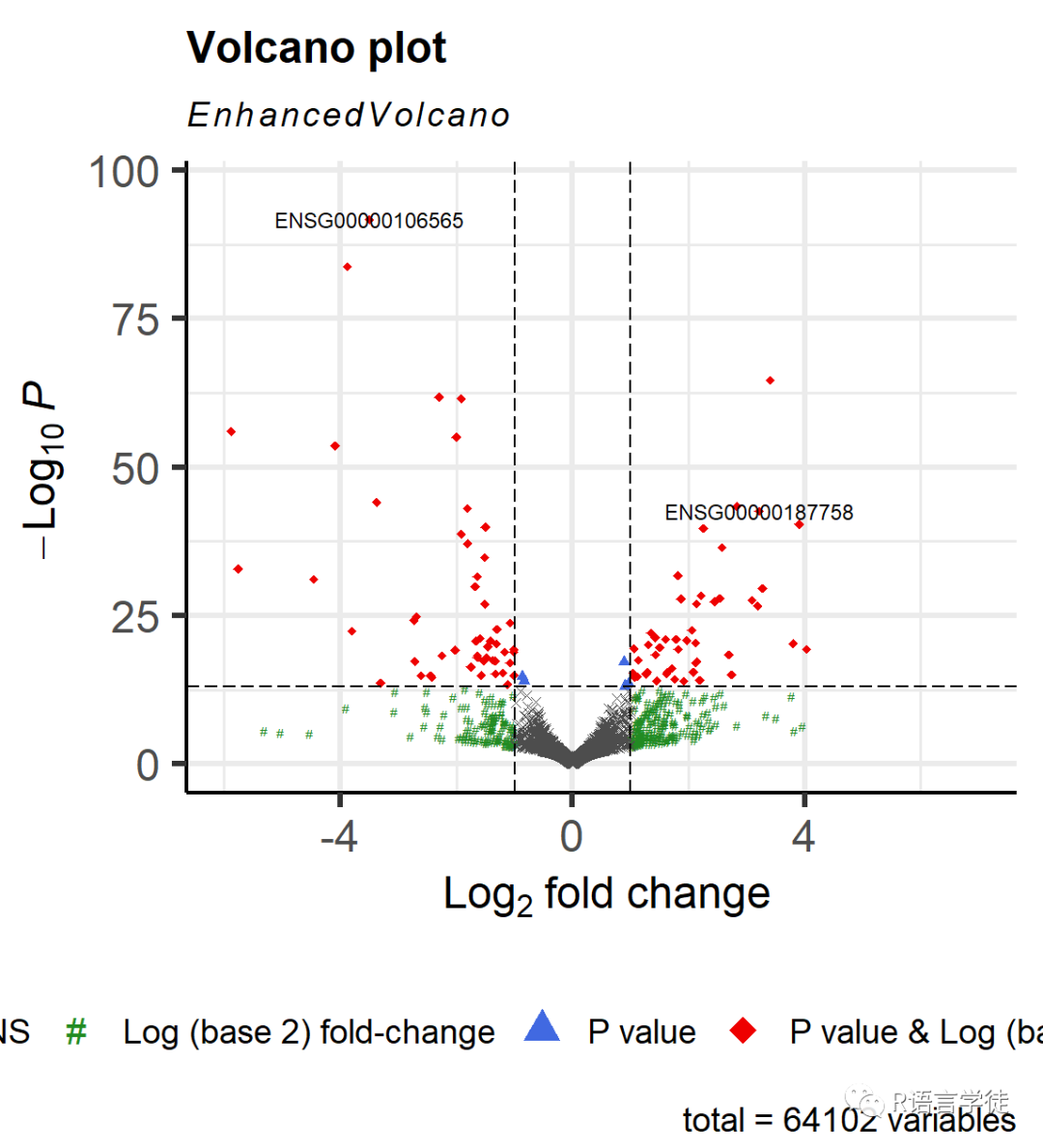

8.给标签加上框
Mid-autumn Festival

EnhancedVolcano(res2,
lab = rownames(res2),x = 'log2FoldChange',y = 'pvalue',selectLab = c('ENSG00000106565','ENSG00000187758','ENSG00000230795', 'ENSG00000164530','ENSG00000143153'),xlim = c(-5.5,8),pCutoff = 10e-14,FCcutoff = 2.0,pointSize = 1.5,labSize = 3.0,labCol = 'black',labFace = 'bold',# 加框boxedLabels = TRUE,colAlpha = 4/5,legendLabels=c('NS','Log (base 2) fold-change','P value','P value & Log (base 2) fold-change'),legendPosition = 'right',legendLabSize = 14,legendIconSize = 4.0,)

怎么样,大家跟着小师妹的步伐有没有学会呢?今天的讲解今天就到这啦,如果有其他问题,欢迎小伙伴们随时与小师妹讨论哦,下次再会~

最后,祝福大家月圆人圆事事团圆,人顺心顺事事都顺~

佳
节
团
圆

如果小伙伴有其他数据分析需求,可以尝试使用本公司新开发的生信分析小工具云平台,零代码完成分析,非常方便奥,云平台网址为:http://www.biocloudservice.com/home.html,包括了用edgeR实现多组差异分析(http://www.biocloudservice.com/289/289.php),DEseq2实现多组差异分析(http://www.biocloudservice.com/287/287.php)等小工具
往期 · 推荐

END

