超详细的使用 r8s进行分歧时间估计分析的教程
点击蓝字 关注我们
“分歧时间” 指的就是物种之间分化的大概时间区域。主要原理是基于系统发育树中已知节点的分歧时间(已被报道的分支时间节点/化石时间)和分子钟计算,来推算其他节点的分歧时间。
从原理可以看出分歧时间估计分析的前提是要有构建好的系统发育树,至于系统发育树的构建方法,小花以前也介绍过了,大家如果有不知道如何构建系统发育树的可以看一下。
目前,用来进行分歧时间估计分析的常见软件有:r8s 、mcmctree以及BEAST等等。今天小花主要和大家分享使用r8s进行分歧时间估计分析的详细流程以及一些注意事项。
r8s是美国加利福尼亚大学戴维斯分校的进化生物学家Mike Sanderson编写的用于估算进化树分化时间的软件,在进化生物学、分子生物地理学等学科有着广泛的应用,已经成为估算分化时间不可或缺的软件之一。
r8s我们可以直接从这个网站(r8s download | SourceForge.net)下载,解压后进入编译安装就行了。
首先进行分歧时间估计分析,我们要先准备一个newick格式的进化树,进化树的构建方式有很多,小花介绍过使用orthofinder基于组学数据构建进化树以及利用在线工具构建进化树。这里就不再介绍了。我们的树文件大概就如下图所示,注意我们构建好的进化树文件一般都含有置信值,这个我们需要删掉,分歧时间估计分析只需要保留枝长信息就行。

r8s运行需要如下的文件作为输入文件,大家可以自己制作,当然我们也可以用脚本命令来制作,这个脚本可以在github上找到,如果有需要也可以找小花。
python2 cafetutorial_prep_r8s.py -i RAxML_bipartitions.result -o r8s_ctl_file.txt -s 275614 -p ‘Tetthe,Partet' -c‘610'#制作r8s输入文件#-I 就是你的进化树文件(newick格式)# -o 输出结果# -s 用于构建进化树的比对序列所含的基因位点信息,可以在bioedit中打开查看# -p 只用用于作为时间矫正点的物种,名字要和进化树里一致# -c 就是上面两个物种之间的分歧时间,这个可以来自文献,也可以从这个网站(TimeTree :: The Timescale of Life)查找,其结果如下图所示。
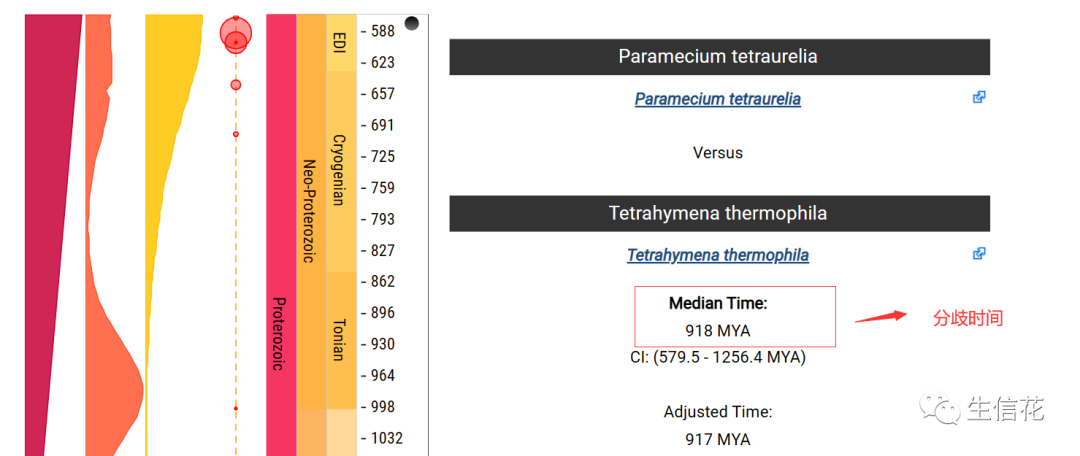
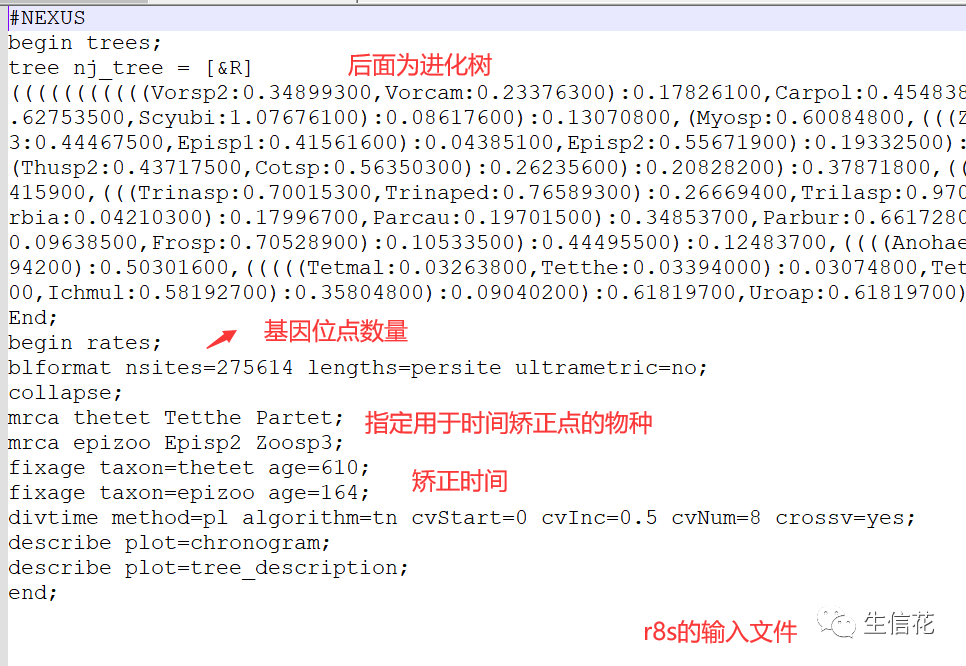
我们构建好如上图所示的r8s输入文件,就可以开始分析了。当然我们也可以对它进行修改比如我们脚本只能指定一个矫正时间点,但是我们可以在输入文件中自己再按照格式加入其他时间矫正点,从而使估计的分歧时间更加准确。
接下来我们就开始运行r8s来进行分歧时间估计分析吧!
r8s -b -f r8s_ctl_file.txt > r8s_tmp.txt #运行r8s$ tail -n 1 r8s_tmp.txt | cut -c 16- > twelve_spp_r8s_ultrametric.txt #从r8s中提取出时间树文件
好了, 上面命令后我们最终就能得到如下图所示的结果文件,现在各节点处就为分歧时间了。当然我们刚得到的结果文件中还包含我们设定的时间节点信息,我们需要将其删掉,这样后续作图软件打开才不会报错。

得到的结果文件,我们可以使用figtree来打开,结果就如下图所示,节点处的数字就是两个类群之间的分歧时间了。大家也可以在figtree里面根据自己的需求对树形进行调整及美化,这个软件具体的使用方法大家如果有需要,小花后续可以出一个相关教程。都调整好后,我们就可以将其导出为图片格式就行了。到此我们就使用r8s完成了分歧时间估计分析了。
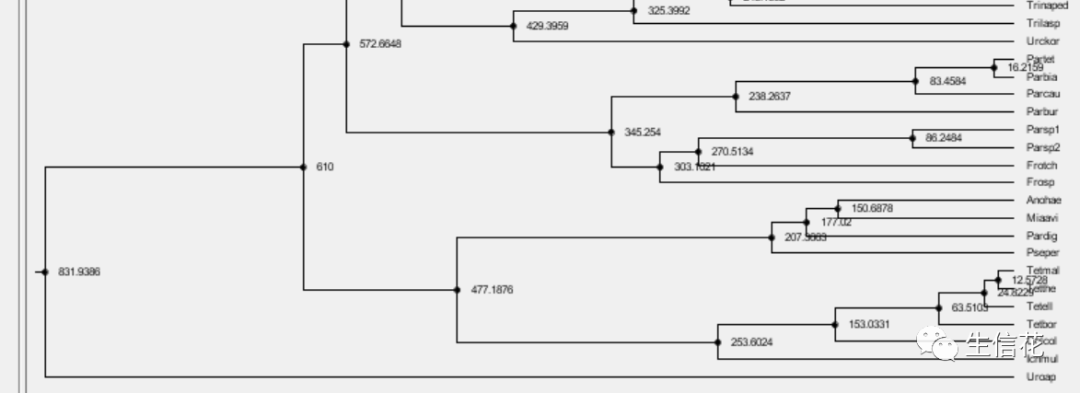
好了以上就是小花为大家分享的内容,是不是很简单呀!后续大家在分析过程中有任何问题都可以来找小花讨论哦!那我们就下期再见咯!
欢迎使用:云生信 – 学生物信息学 (biocloudservice.com)
如果想用服务器可以联系微信:18502195490(快来联系我们使用吧!)

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

