保姆级教程!跟着小果优雅的绘制你的单细胞数据-scCustomize!
 看过小果前几期对单细胞标准流程处理的小伙伴肯定熟悉单细胞的结果,但是有的小伙伴不知道如何将结果可视化,或者觉得普通的可视化结果已经满足不了小伙伴的胃口,那么这次小果给小伙伴带来一期专门绘制单细胞相关分析的R包scCustomize的使用!
看过小果前几期对单细胞标准流程处理的小伙伴肯定熟悉单细胞的结果,但是有的小伙伴不知道如何将结果可视化,或者觉得普通的可视化结果已经满足不了小伙伴的胃口,那么这次小果给小伙伴带来一期专门绘制单细胞相关分析的R包scCustomize的使用!
下面我们开始吧
我们这里先了解一下scCustomize包,scCustomize目的在帮助使用R对单细胞数据进行可视化和分析。小伙伴要注意下scCustomize的安装需要在4.0以的R版本上。
我们这里使用的数据是前几期小果教大家的单细胞处理流程最终的结果,不清楚的小伙伴可以回过头再看一下,小果也把数据放在这里,
我们先#加载绘图所需要的包
正常的安装就可以
#install.packages("scCustomize")library(tidyverse)library(patchwork)library(viridis)library(Seurat)library(scCustomize)library(qs)
下面我们加载数据
# 基础分析得到的数据pbmc <- readRDS("pbmc3k_final.rds")# 向pbmc数据中添加一些随机元数据变量,后面绘图使用pbmc$sample_id <- sample(c("sample1", "sample2", "sample3", "sample4"), size = ncol(pbmc),replace = TRUE)pbmc$treatment <- sample(c("Treatment1", "Treatment2"), size = ncol(pbmc),replace = TRUE)
公众号后台回复“111”领取本篇代码、基因集或示例数据等文件;
文件编号:231203
果粉福利:生信人必备神器——服务器
平时生信分析学习中有要的小伙伴可以联系小果租赁,粉丝福利都是市场超低价格,赶快找小果领取免费的试用账号吧!
接下来我们对高变基因进行绘制
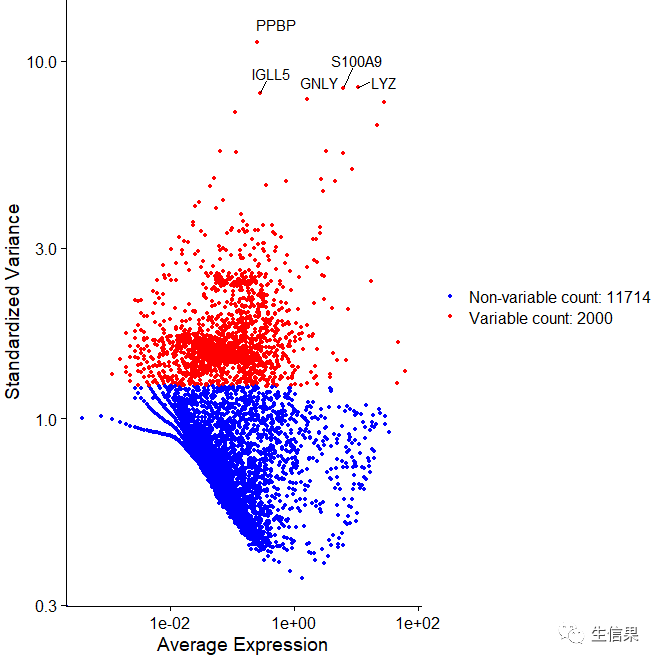
上述参数中num_features:基因数量 label:是否进行标记 repel:连接点和标签 y_axis_log:对y轴进行取对数 colors_use:设置颜色 pt.size:点的大小设置。
接下来就是绘制一些特征图
# 设置颜色pal <- viridis(n = 10, option = "D")FeaturePlot_scCustom(seurat_object = pbmc, features = "GNLY", order = F)
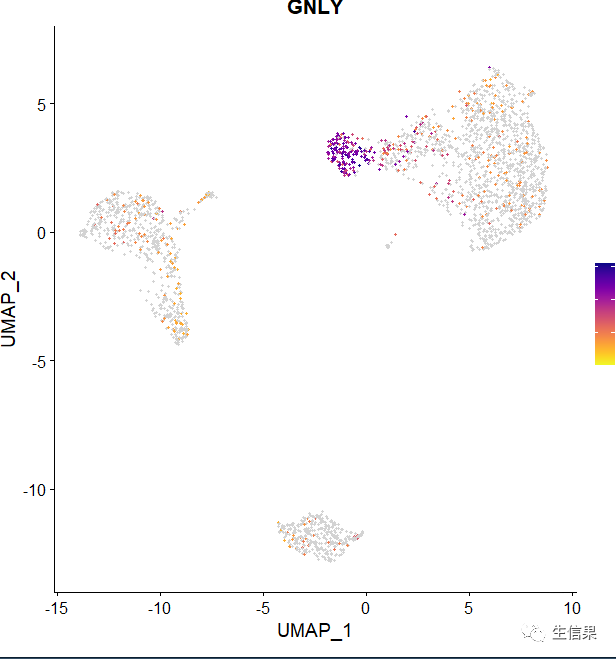
FeaturePlot_scCustom(seurat_object = pbmc, features = "GNLY")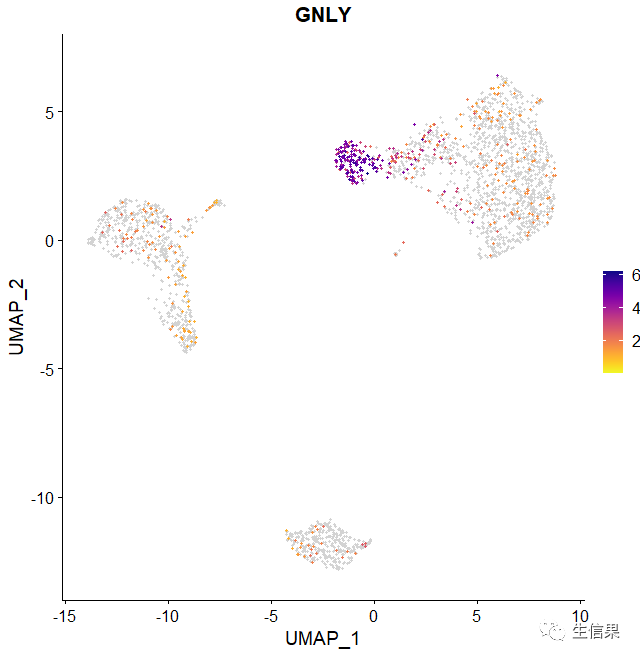
FeaturePlot_scCustom(seurat_object = pbmc, features = "GNLY", colors_use = pal)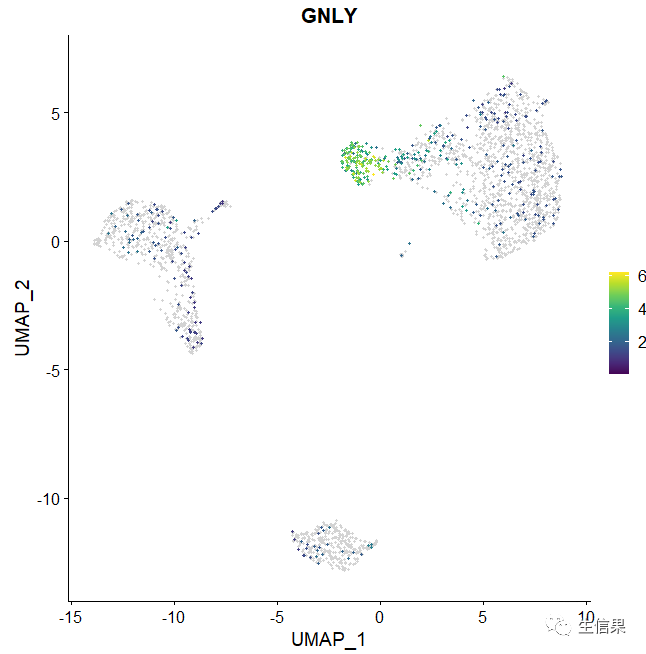

其中呢,order=F,可能会导致非表达细胞绘制在表达细胞之上。有时order=T可能会导致绘制的点不够集中,可以由绘制基因的情况根据需要来进行调整,小伙伴要注意下哦!
al <- viridis(n = 10, option = "C", direction = -1)FeaturePlot_scCustom(seurat_object = pbmc, features = "CD3E", order = T,alpha_exp = 0.75,colors_use = pal)
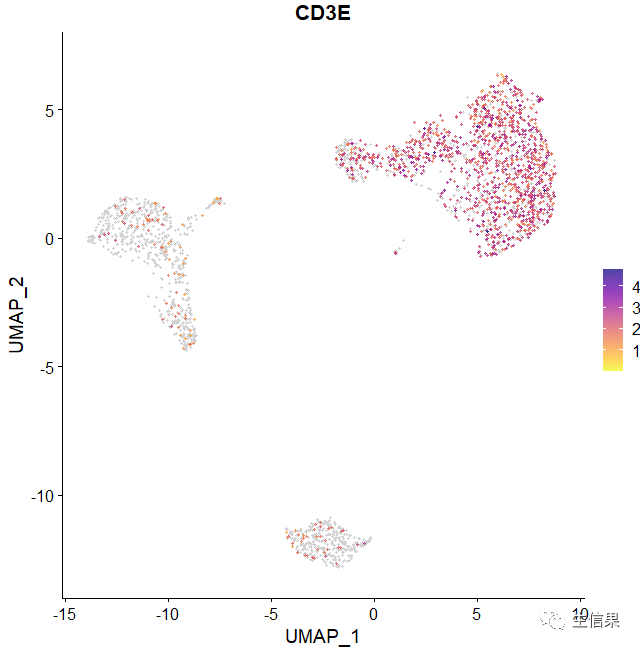
FeaturePlot_scCustom(seurat_object = pbmc, features = "CD3E", order = F,alpha_exp = 0.75,colors_use = pal)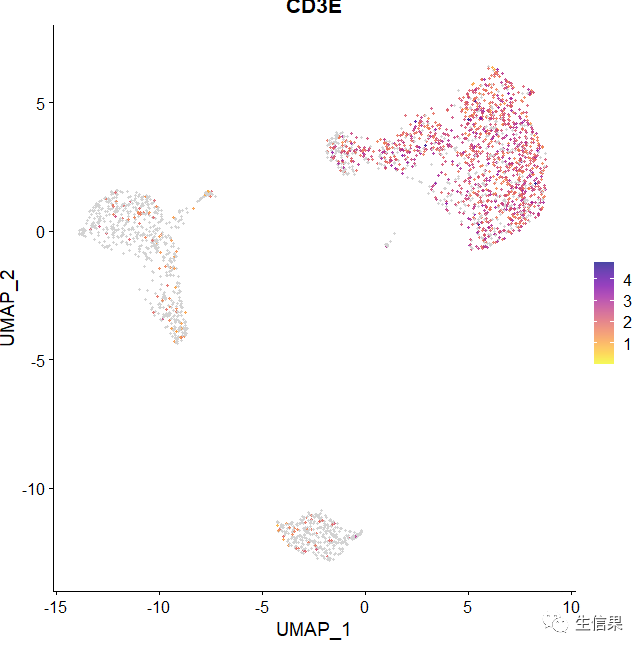
我们换个好看的颜色,注意下参数alpha_exp:更改点的透明度 colors_use:设置颜色。
后面到了我们的关键图之一了,绘制下密度图,但是密度图的绘制需要Nebulosa包进行配色,可惜的是Nebulosa仅支持使用5个viridis调色板进行绘图:“viridis”、“magma”、“cividis”、“inferno”和“plasma”。不过没事够我们使用了,来看一下
library(Nebulosa)#小伙伴需要下载一下这个包一般使用#BiocManager::install("Nebulosa")就可以Plot_Density_Custom(seurat_object = pbmc, features = "GNLY")
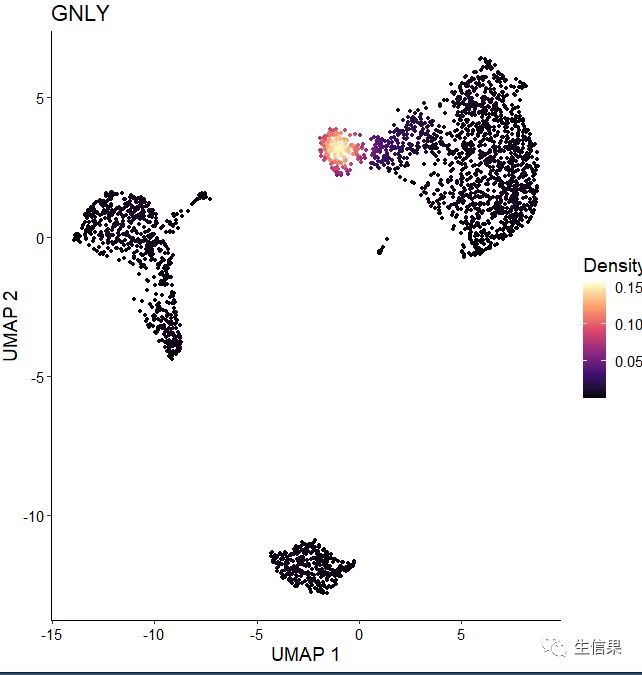
Plot_Density_Custom(seurat_object = pbmc, features = "GNLY", custom_palette = PurpleAndYellow(),)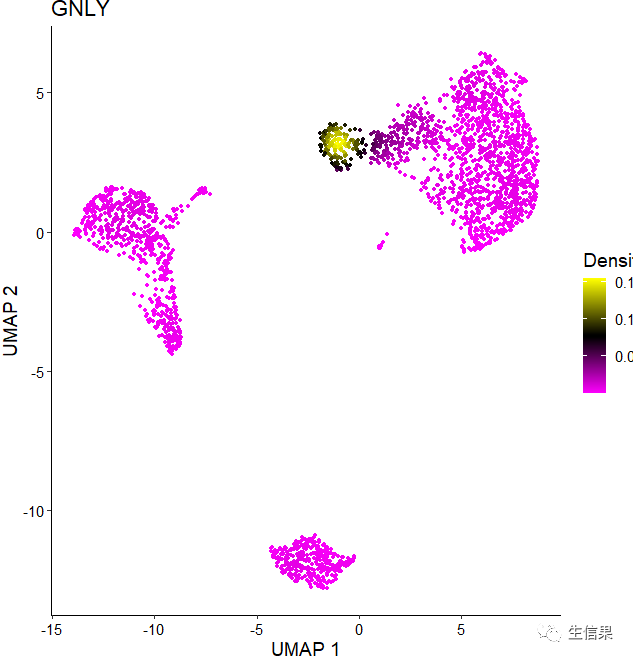
我们还可以使用Plot_Density_Joint_Only()可以绘制联合密度图。
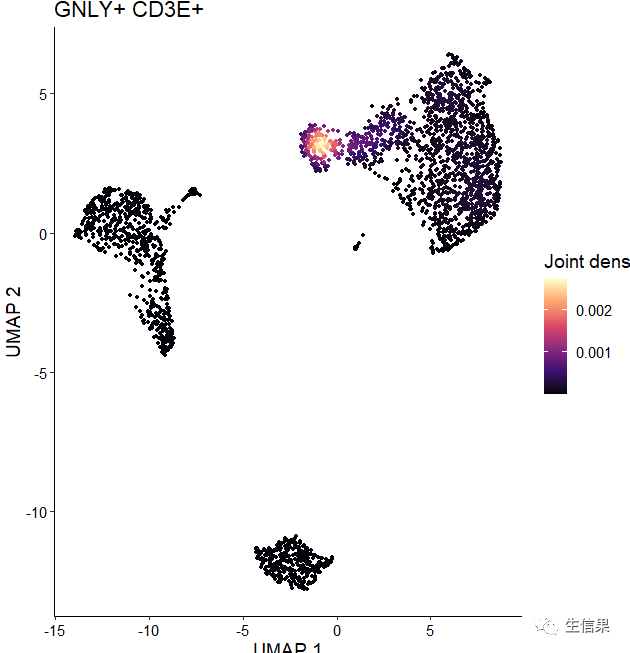
是不是不一样了呢
最后一个呢,我们要了解一下使用双数据绘制图,因为在某些情况下,可能需要从同一个对象内绘制两个不同数据处理的图。例如,当对象包含原始计数和校正后数时,可能需要绘制两个数据中的相同基因以查看差异。
这里使用昨天保存的pbmc_tutorial.rds数据,存到pbmc的RAW数据中并重新标准化,只是为了演示函数使用
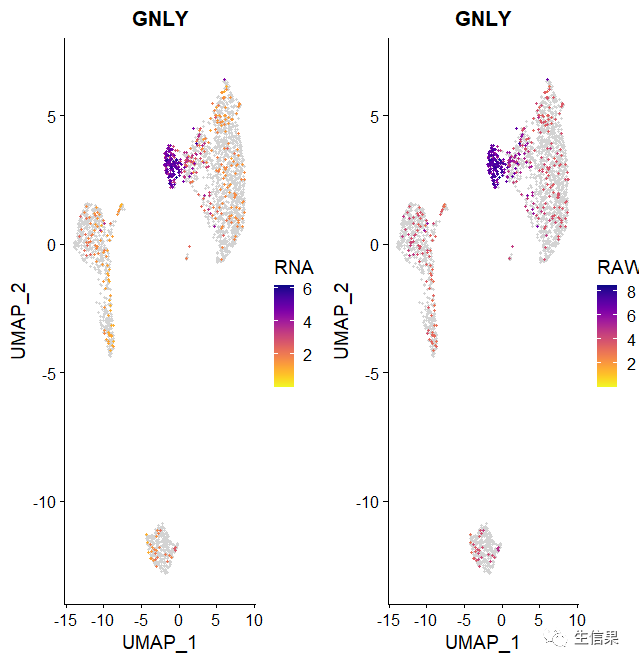
这样一来就大功告成了,以上就是小果关于scCustomize包的单细胞可视化的教程,其实这个包的用途还有很多,小伙伴要多多关注我们公众号,后面我们会带给小伙伴更多的内容,这个包的小提琴图的绘制还以聚类点图还以颜色的调整等等很多,最后小果还以提醒小伙伴要多多理解代码的意义,才能绘制出自己想要的图片哦


往期推荐