超级超级牛的IOBR包,一包解决7种免疫浸润分析方法
点击蓝字 关注我们
今天小花为大家带来的分享灰常灰常有用,对于很多小伙伴来说,免疫浸润分析是非常熟悉的分析内容,免疫浸润分析方法主要包括xcell,MCP-counter,timer,quantiseq,epic,cibersort,estimate等方法,但是这几种方法都有各自的R包来进行分析,有几种方法就需要安装和学习几种R包,对小白和初学者来说非常不友好,需要花费大量的时间,基于这种情况,小花为小伙伴推荐一个R包IOBR,该包直接可以完成多种算法的免疫浸润分析,每个算法都只需要一句代码完成分析,非常简单奥!小花强烈推荐,接下来跟着小花开始今天的分享学习。
1.安装IOBR包
首先小花为大家简单介绍一下如何安装和学习IOBR包,小花为小伙伴们附上网址:https://iobr.github.io/IOBR/IOBR-VIGNETTE.html,想深入学习的小伙伴可以到该网址学习。

#需要注意的是官网推荐使用github的方法来安装,但是由于网络问题,通过github安装非常非常慢,一般都不能安装成功,因此小花推荐直接下载安装包,进行本地安装,安装速度非常快!安装包可以到该网址下载:https://github.com/IOBR/IOBR。
#首先安装依赖的R包if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager")depens<-c('tibble', 'survival', 'survminer', 'sva', 'limma', "DESeq2","devtools",'limSolve', 'GSVA', 'e1071', 'preprocessCore', 'ggplot2', "biomaRt",'ggpubr', "devtools", "tidyHeatmap", "caret", "glmnet", "ppcor", "timeROC","pracma")for(i in 1:length(depens)){depen<-depens[i]if (!requireNamespace(depen, quietly = TRUE))BiocManager::install(depen,update = FALSE)}#安装IOBR包install.packages("IOBR-master.tar.gz",repos=NULL,type="source")#加载IOBR包library(IOBR)
2.几种免疫算法的简单介绍
#quanTIseq算法简单介绍
quanTIseq是用于根据人类RNA-seq数据量化肿瘤免疫状况,通过反卷积量化样本中存在的10种不同免疫细胞类型的比例以及其他未表征细胞的比例。
#xcell算法简单介绍
xcell是基于ssGSEA的方法,可根据64种免疫细胞和基质细胞类型的基因表达数据进行细胞类型富集分析。
#TIMER算法简单介绍
TIMER用反卷积方法估算32种癌症中六种免疫细胞丰度,需要注意的是TIMER有在线的网站可以直接进行分析。
#EPIC算法简单介绍
EPIC使用约束最小二乘回归将负性约束条件纳入反卷积问题,从大量肿瘤样本的表达谱数据进行分析。
3.读取输入数据
#EXP.txt基因表达矩阵,行名为基因名,列名为样本名
dat <- read.table("EXP.txt",sep = "t",row.names = 1,header = T,stringsAsFactors = F,check.names = F)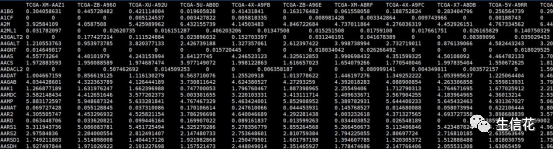
4.通过IOBR包采用多种算法进行免疫浸润分析
#利用CIBERSORT算法进行免疫浸润分析
cibersort<-deconvo_tme(eset = dat, method = "cibersort", arrays = FALSE, perm = 200 )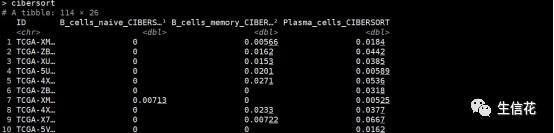
#利用EPIC算法进行免疫浸润分析
epic<-deconvo_tme(eset = dat, method = "epic", arrays = FALSE)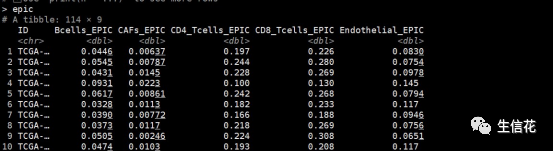
#利用mcpcounter算法进行免疫浸润分析
mcp<-deconvo_tme(eset = dat, method = "mcpcounter")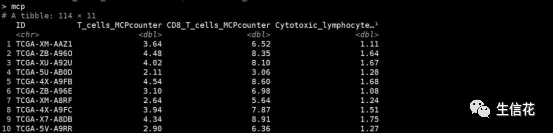
#利用xCELL算法进行免疫浸润分析
xcell<-deconvo_tme(eset = dat, method = "xcell",arrays = FALSE)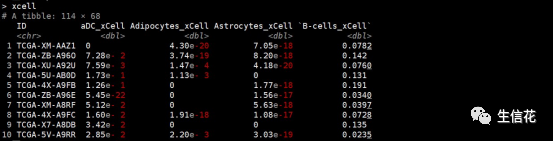
#利用estimate算法进行免疫浸润分析
estimate<-deconvo_tme(eset = dat, method = "estimate")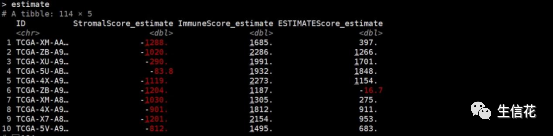
#利用TIMER算法进行免疫浸润分析
timer<-deconvo_tme(eset = dat, method = "timer", group_list = rep("stad",dim(dat)[2]))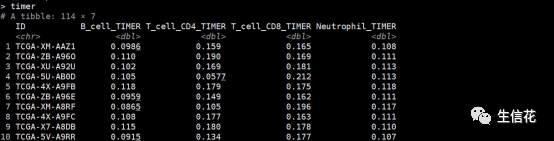
#利用quanTIseq算法进行免疫浸润分析quantiseq<-deconvo_tme(eset = dat, tumor = TRUE, arrays = FALSE, scale_mrna = TRUE, method = "quantiseq")
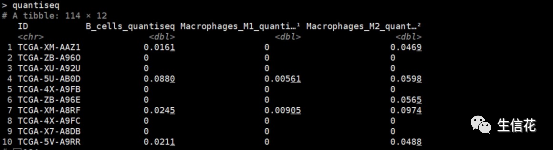
tme_combine<-cibersort %>%inner_join(.,mcp,by = "ID") %>%inner_join(.,xcell,by = "ID") %>%inner_join(.,epic,by = "ID") %>%inner_join(.,estimate,by = "ID") %>%inner_join(.,timer,by = "ID") %>%inner_join(.,quantiseq,by = "ID")
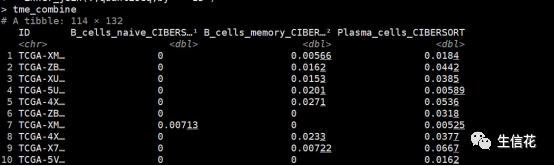
#IPS免疫表型评分计算
ips<-deconvo_tme(eset = dat, method = "ips", plot= FALSE)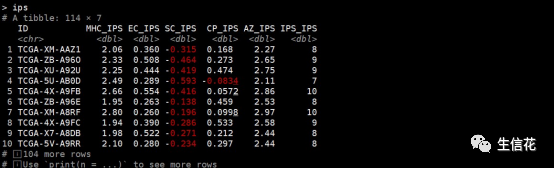
最终小花成功的利用IOBR包算法进行了免疫浸润分析,包括CIBERSORT,EPIC,mcpcounter,xCELL,estimate,TIMER,quanTIseq 7种算法,最后还计算了免疫表型评分(IPS),该包分析效果非常不错,欢迎大家和小花一起讨论学习呀!免疫浸润相关分析内容,例如单样本富集算法分析免疫浸润丰度(http://www.biocloudservice.com/106/106.php),计算64种免疫细胞相对
含量(http://www.biocloudservice.com/107/107.php),panImmune免疫浸润分析(http://www.biocloudservice.com/782/782.php),基于基因表达谱估算样本免疫基质评分和肿瘤纯度(http://www.biocloudservice.com/114/114.php)等都可以用本公司新开发的零代码云平台生信分析小工具,一键完成该分析奥,感兴趣的小伙伴欢迎来尝试奥,网址:http://www.biocloudservice.com/home.html。今天小花的分享就到这里,下期在见奥。



长按识别二维码关注我们哟

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

