Kmergenie + GapCloser让你的基因组组装事半功倍

大家好,小果最近在用SOAP denovo2对二代测序结果进行组装,在SOAP denovo2的命令中有一个参数k-mer,那么问题来了,k-mer代表什么呢?该如何设置结果才会更好呢?拼接后的coting序列中有一些未知碱基N,可不可以减少未知碱基呢?小果今天给大家推荐的k-mer频率统计软件kmergenie和补洞工具GapCloser就可以很好的帮我们解决这两个难题。
1. kmergenie
1.1 kmergenie是什么?
kmergenie是一个开源的软件工具,用于估计最佳k-mer的大小,它有助于后续的基因组组装。k-mer是指长度为k的DNA序列,它可以从测序数据中提取出来。不同的k-mer大小会影响组装的质量和效率,因此选择一个合适的k-mer大小是很重要的。kmergenie可以根据测序数据的特征,自动计算出一个最佳的k-mer大小,从而提高组装的准确性和速度。
1.2 如何下载与安装kmergenie?
kmergenie可以在Linux和Mac OS X系统上运行,它需要一些依赖软件,如gcc,make,python和zlib。
wget http://kmergenie.bx.psu.edu/kmergenie-1.7051.tar.gztar -xzvf kmergenie-1.7051.tar.gzcd kmergenie-1.7051python setup.py install#添加可执行权限chmod -R 755 *

这样就完成了kmergenie的安装,可以使用以下命令来测试是否成功:
kmergenie --version如果你看到了kmergenie的版本号,那么恭喜你,你已经成功安装了kmergenie!
1.3 如何运行kmergenie?
要运行kmergenie,你需要准备一些输入文件,包括测序数据和配置文件。测序数据可以是FASTA或FASTQ格式的文件,也可以是压缩过的.gz或.bz2格式的文件。配置文件是一个文本文件,其中每一行包含一个测序数据文件的路径和相应的权重。权重是一个介于0和1之间的数值,表示该文件在总测序数据中所占的比例。例如,如果你有两个测序数据文件,一个是100M,另一个是150M,那么你可以给第一个文件分配0.4的权重,给第二个文件分配0.6的权重。配置文件的示例如下:

/home/user/seq1.fastq 0.4/home/user/seq2.fastq.gz 0.6
当你准备好输入文件后,就可以使用以下命令来运行kmergenie:
kmergenie config.txt -o output其中config.txt是配置文件的路径,-o output是指定输出目录的选项。也可以使用其他一些选项来调整kmergenie的参数,如-k指定最小和最大的k-mer大小,-l指定输出结果的格式等。你可以使用以下命令来查看所有可用的选项:
kmergenie --help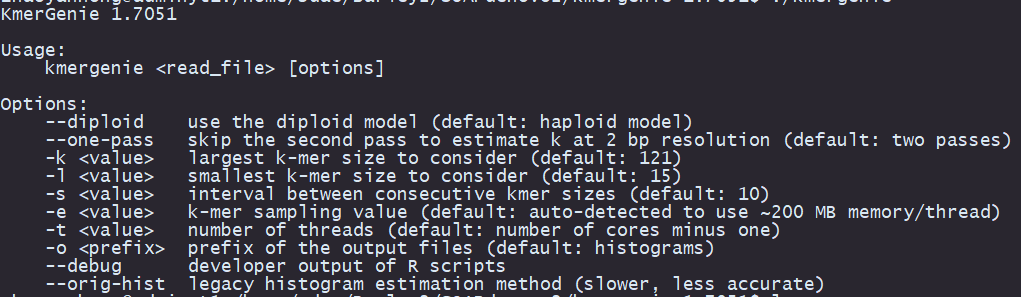
--diploid:使用二倍体模型(默认值:单倍体模型)--one-pass:跳过第二次传递以估计2 bp分辨率下的k(默认值:两次传递)-k:要考虑的最大k-mer大小(默认值:121)-l:要考虑的最小k-mer大小(默认值:15)-s:连续kmer大小之间的间隔(默认值:10)-e:k-mer采样值(默认值:自动检测以使用约200 MB内存/线程)-t:线程数(默认值:核心数减一)-o:输出文件的前缀(默认值:直方图)--debug:R脚本的开发人员输出--orig-hist:旧版直方图估计方法(速度较慢,准确性较低)。

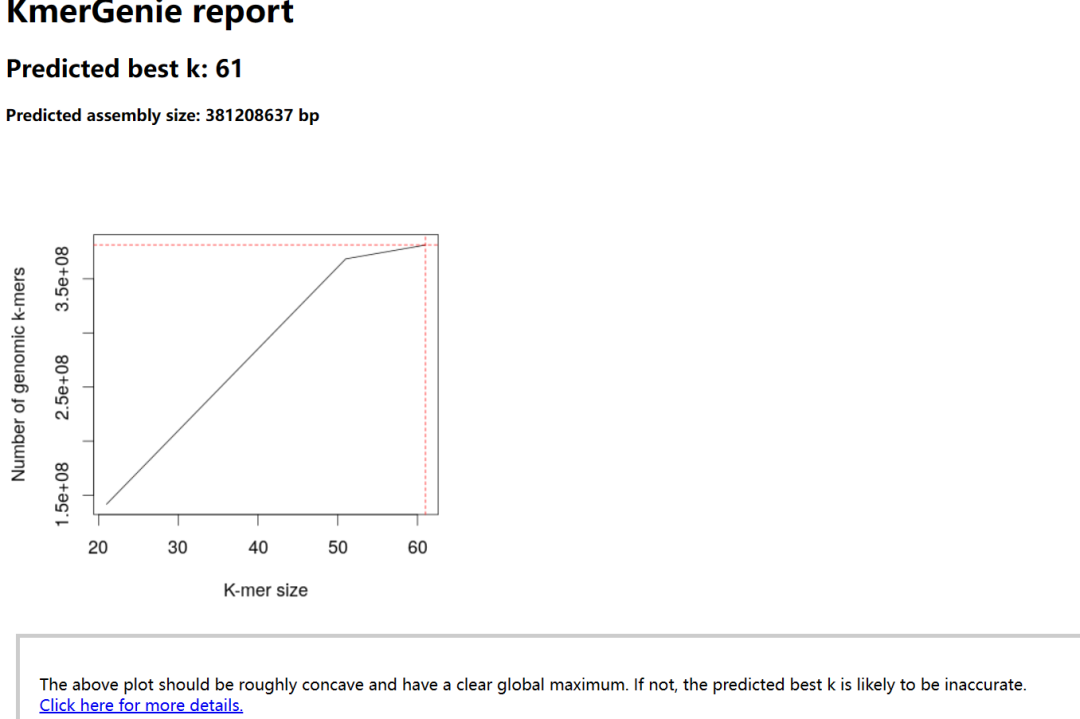
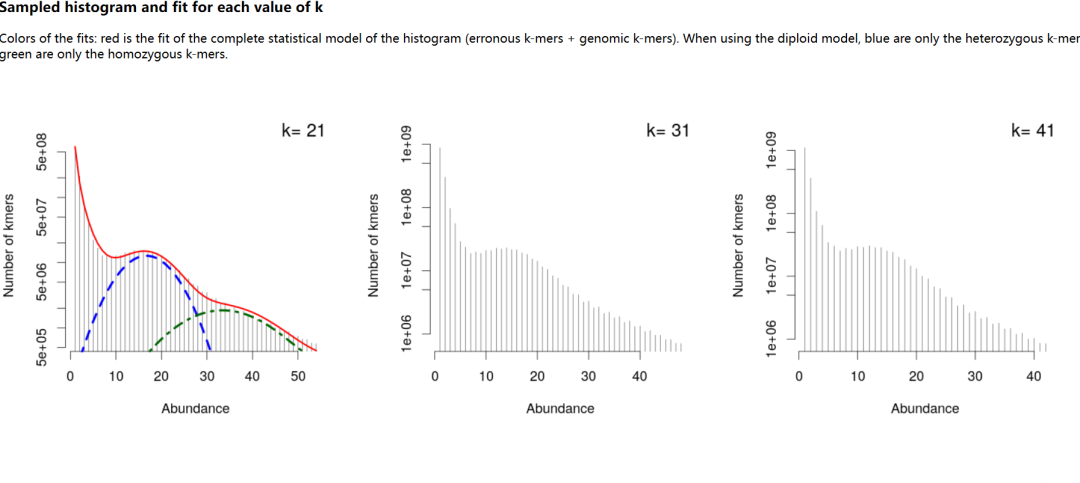

2. GapCloser
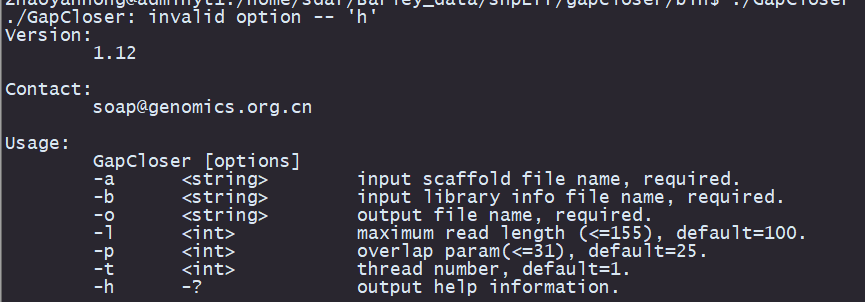
建文件夹并进入:mkdir GapCloser && cd GapCloser下载安装包并解压:wget https://anaconda.org/bioconda/soapdenovo2-GapCloser/1.12/download/linux-64/soapdenovo2-GapCloser-1.12-1.tar.bz2tar -jxvf soapdenovo2-GapCloser-1.12-1.tar.bz2进入bin目录:cd bin/查看是否安装成功:./GapCloser -h
max_rd_len=150[LIB]avg_ins=500reverse_seq=0asm_flags=3map_len=32q=/path/to/single/reads.fqq1=/path/to/read1.fqq2=/path/to/read2.fq
./GapCloser -a /path/to/contig.fa -b /path/to/config_file -o /path/to/output_file -t number_of_threadsgcc:https://gcc.gnu.org/install/download.htmlmake:https://www.gnu.org/software/make/zlib:https://www.zlib.net/kmergenie:http://kmergenie.bx.psu.edu/gapclode:Files :: Anaconda.org

往期推荐
