IF12+,最快1个月接收,国人占比63%,关键还纯生信友好,这本期刊太牛了!不信看华科团队这篇新作,0实验轻松拿下!



研究背景
数据来源
本文综合分析了多个数据来源以构建和验证NEPC风险预测模型。收集并深入研究了11个scRNA-seq数据集,其中包括9个发现数据集和2个验证数据集,这些数据集覆盖了原发性HSPC、CRPC、mCRPC和NEPC等类型,最终保留了来自66个肿瘤样本的210,879个高质量细胞和20,870个基因。为了更全面地研究前列腺癌,从15个公开的人类前列腺癌批量数据集中收集了3000多个样本,这些数据集涵盖了多种前列腺癌类型和阶段,RNA-seq数据来源于UCSC XENA、ICGC、cBioPortal等多个权威平台,从GEO下载了空间转录组测序数据。
研究思路
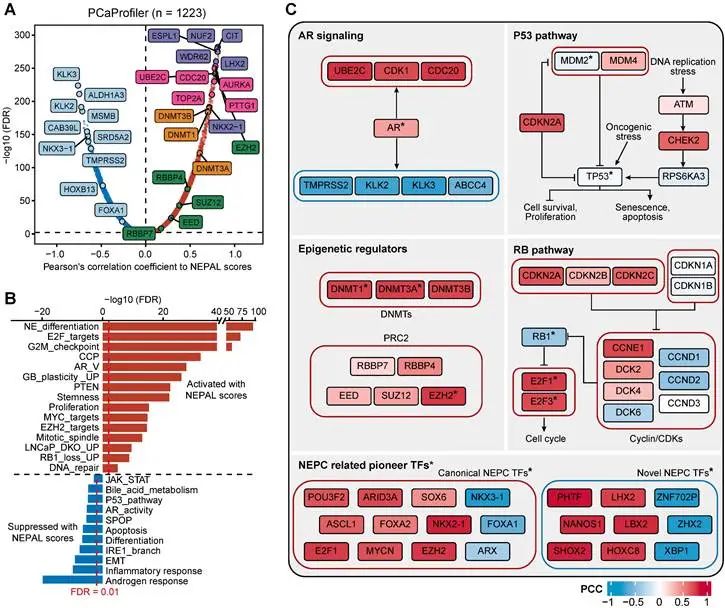
本研究整合了11个NE相关基因集、11个scRNA-seq队列、15个bulk转录组队列及13个PCa实验模型的数据,运用先进算法,构建了一个稳健的NEPC风险预测模型。在评估了包含21万余个单细胞的scRNA-seq图谱后,发现现有NE基因集存在不一致性。因此开发了一个新的分析流程,筛选出762个高质量的NE标记,并基于这些标记创建了NEPAL风险评估模型。经验证,该模型能准确预测NEPC风险,描绘PCa进展,甚至预测预后和治疗反应,为NEPC的鉴定和前列腺癌进展的监测提供了有力工具。
主要结果
1.先前的 NEPC 基因组显示出低一致性和低功率






文章小结
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果

往期回顾
|
01 |
|
02 |
|
03 |
|
04 |