想要挖掘lncRNA的靶点miRNA?那你一定不能错过这个!
点击蓝字 关注我们
小伙伴们大家好!我是大海哥,今天介绍文章主题之前,我想先提出一个问题。
现代生物学的开端是什么?
大海哥觉得,一定是中心法则!
如果说, 细胞学说和达尔文进化论充分体现了近代自然科学的理论特征,把联系与发展的观点引入了生物学, 从而成为近代生物学的理论基石, 那么可以说,中心法则充分体现了现代自然科学的理论特征,把多因子的、动态的、复杂的相互作用引入生物学, 其可以视为现代生物学的理论基石。
中心法则大家都知道,大海哥这里就不多解释,今天讨论的主题就是中心法则三个步骤中的转录和翻译都密切相关的miRNA-lncRNA潜在关联。miRNA和lncRNA都是非编码RNA的一类,它们在细胞内起着调节基因表达的重要角色。部分miRNA和lncRNA可以相互作用,形成复杂的调控网络。某些lncRNA可以作为miRNA的底物,通过结合miRNA来调节miRNA对目标基因的调控。这种相互作用可以在细胞中形成复杂的调控回路。同时miRNA和lncRNA可以共同调控同一组基因,从而协调细胞内的基因表达。这种协同作用可能导致特定的生物过程或生理功能的调控。
在这个联合调控过程中,部分lncRNA能够将miRNA吸附过来,相当于竞争性结合,不让miRNA对mRNA发挥作用,因此形成了lncRNA-miRNA-mRNA的调控网络,简称ceRNA。在做生信分析时,如果我们通过数据库筛选了一些调控自己靶基因mRNA的lncRNA,可以使用在线数据库来预测lncRNA结合的miRNA。
但是问题就来了,大部分网站通常都是输入一个lncRNA,预测与其结合miRNA。如果我们筛选到了几十个甚至几百个lncRNA,如何批量预测lncRNA靶向的miRNA。所以大海哥向大家推荐一个在线数据库:mircode
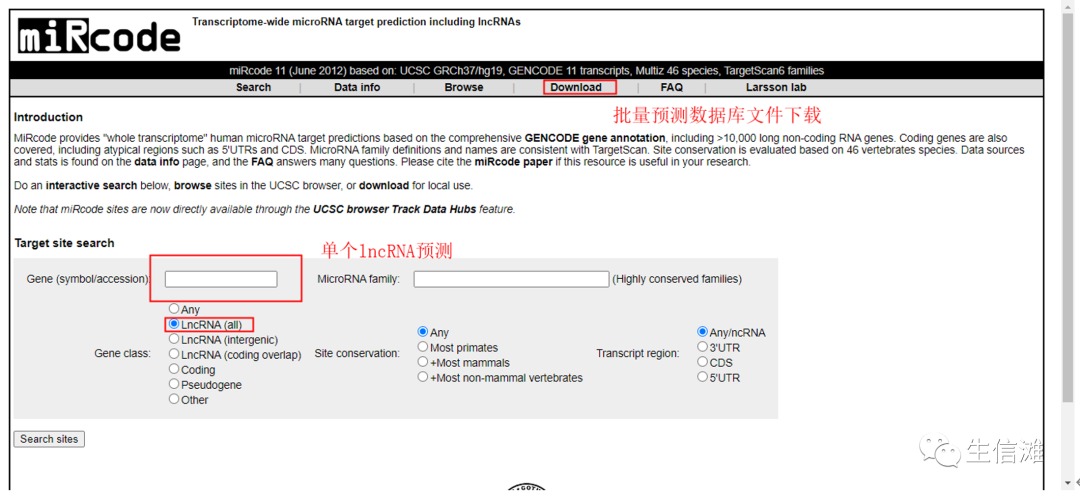
数据库页面如上图,左侧红框位置可以支持我们单个gene查询哦,不仅支持lncRNA,还支持coding、Pseudogene等多种类型,我们先试一下单个预测
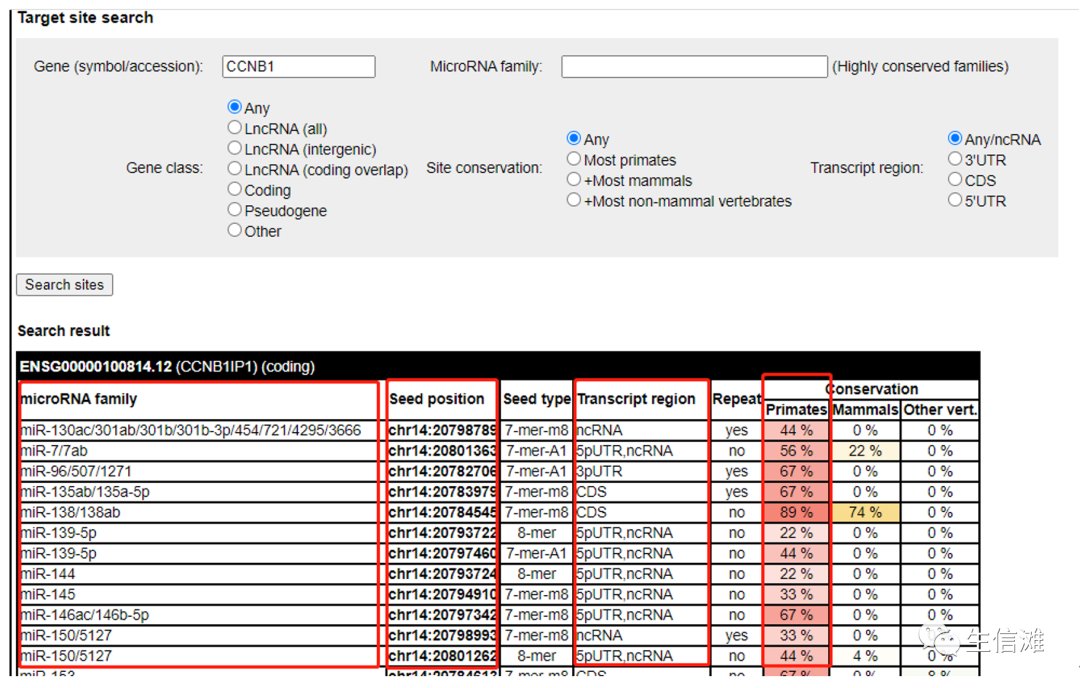
图中预测结果表格包含靶向预测基因的miRNA名字,染色体位点,区域,保守性等等
当然也可以输入某个miRNA family,查看此miRNA靶向的mRNA或者lnRNA
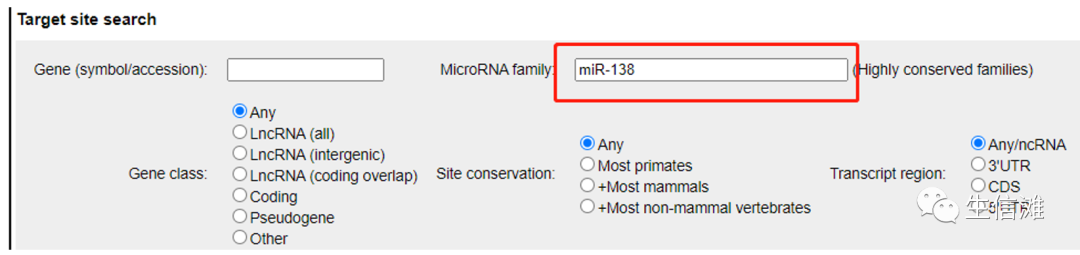
就可以查询到该miRNA靶向的编码或者非编码RNA。如下图
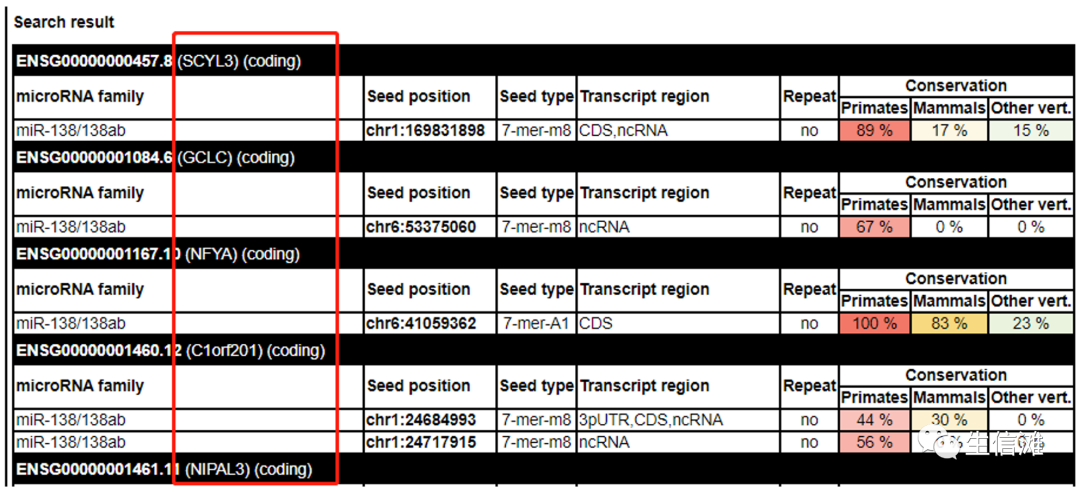
当然了,这些都是单次查询,正如大海哥开始所说的,如果有100个基因或者1000个基因呢?总不能一个一个的查吧!
所以主页右上角也可以下载数据库文件,可以用代码实现批量预测!
让我们直接点击download!

一般我们都会选择图中标记的高保守的miRNA数据,下载后,让我们看看数据长什么样子

可以看到图中有几列标记数据是我们批量分析所需要用的,然后我们再准备好需要进行预测的lncRNA列表,
上图就是待会需要进行预测的lncRNA,然后我们开始代码部分吧!
setwd("C:/Users/Administrator/Desktop")## 设置工作路径dir()lncRNA<-read.table("lncRNA.txt",header=T,sep="t")## 读取自己筛选的差异lncRNAhead(lncRNA)miRNA<-read.table("mircode_highconsfamilies.txt",header=T,sep="t")## 读取mircode下载的文件head(miRNA)miRNA<-miRNA[miRNA$gene_symbol %in% lncRNA$SYMBOL,] ## 从miRNA中提取我们筛选的差异lncRNAdim(miRNA)

head(miRNA)#看看预测结果
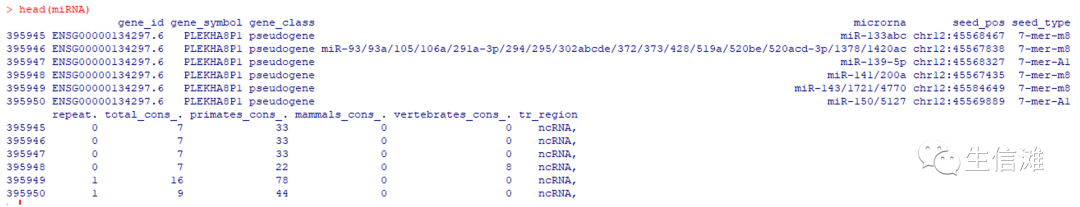
#选择我们需要的几列,保留基因ID和基因名以及基因类型,大家自行选择哦!result<-miRNA[,c(1:4)]#保存结果write.csv(result,"lncRNA结合的miRNA.csv")
点击“阅读原文”进入网址