创建反向互补序列函数
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

还在为如何入门生信而彷徨吗?还在为Linux系统而感到无助吗?快关注小果的微信公众号,小果手把手带你入门生信,没有门槛、没有难度,跟着效果走,啥问题都没有。
小果今天给大家分享一个可以对序列进行反向互补的函数,在碱基编辑二代测序数据分析中常常涉及到序列反向互补的问题,大家一般的方法为去网页搜索在线的反向互补工具,首先在没有网络的情况下就无法使用这种在线工具,其次这种工具一般需要我们一条一条的数据进行复制粘贴,不利于批量的操作,所以小果就想到用python写一个可以在本地运行的函数,可以去实现批量的反向互补的操作。
创建互补函数
#创建互补函数def complement(sequence):sequence = sequence.upper()sequence = sequence.replace('A', 't')sequence = sequence.replace('T', 'a')sequence = sequence.replace('C', 'g')sequence = sequence.replace('G', 'c')return sequence.upper()
创建反向函数
#创建反向函数def reverse(sequence):sequence = sequence.upper()return sequence[::-1]
创建反向互补函数
#创建反向互补函数def complement_reverse(sequence): #反向互补函数return complement(reverse(sequence))
这样小果的反向互补函数就创建出来了,当我们只有几条序列想要进行反向互补操作时就可以直接print。
print(complement_reverse('CCATCAATGCC') ,complement_reverse('CGATGGCGATA'),complement_reverse('ACACAAGCACC'),complement_reverse('CCGTTTCGACG'),complement_reverse('GGAAGTAGACC'))输出结果:
GGCATTGATGG TATCGCCATCG GGTGCTTGTGT CGTCGAAACGG GGTCTACTTCC当我们想要批量实现反向互补操作时就可以把序列复制粘贴到一个txt文件下,用遍历的方式完成。
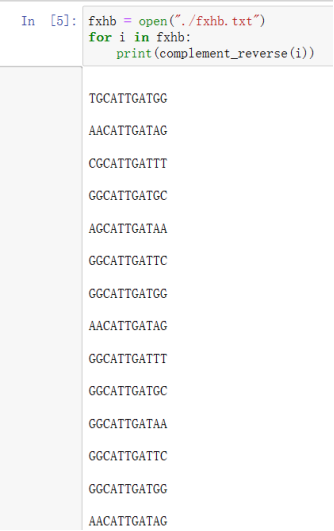
fxhb = open("./fxhb.txt") ##改成自己二代测序的fq文件for i in fxhb:print(complement_reverse(i))
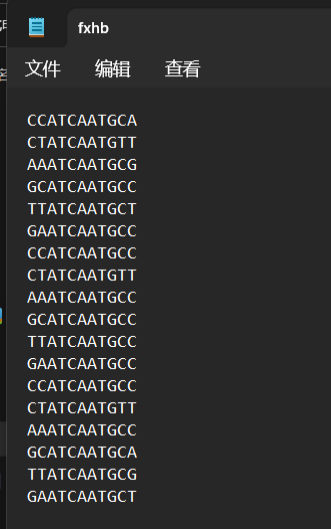
图左:输入序列
图右:输出序列
这样一个小函数就轻松解决了我们在没有网络时无法使用反向互补在线工具的尴尬窘境,也解决了批量操作的问题,大大减少了我们的工作量。好了,今天小果的函数分享就到这里啦,小伙伴们有什么问题欢迎来和小果讨论分享呀。

这里推荐一下小果新开发的零代码云生信分析工具平台包含超多零代码小工具,上传数据一键出图,感兴趣的小伙伴欢迎了解~
网址:http://www.biocloudservice.com/home.html
今天小果的分享就到这里,欢迎大家和小果一起讨论学习,下期再见哦!
小果友情推荐
好用又免费的工具安利


