跟着小果学领略绘图的魅力!轻松复现IF16.6高分文献中的精美Chord图!!

在科学研究中,数据可视化是展示研究成果并传递信息的重要方式。其中,Chord图以其独特的视觉效果和强大的信息传达能力,在展示实体间关系和联系方面扮演着重要角色。
小果今天分享的这份高分文献中,Chord图以IF16.6的高影响因子荣登其中,为读者呈现了一场视觉盛宴。这些图表不仅令人赏心悦目,更是深藏着研究的宝贵信息。
所以小果带领小伙伴去学习这些高分文献中数据可视化的方法,并轻松复现这些高分文献中精美Chord图的绘制过程。我们先了解一下Chord图,其实它是通过环形布局展示了不同实体之间的关系网络,以弦的方式连接各个实体,线条的粗细、颜色的深浅都隐含着数据背后的丰富内涵。每一条线都是一个故事,连接着数据中的关联和联系,而每一次绘制都是对数据本身意义的再次诠释。
这次呢我们就去踏上Chord图的绘制之旅。通过解读高分文献中的数据结构和可视化手法,探索一下Chord图背后的绘制技巧和方法。从数据的准备,到Chord图的布局和装饰,我们将一步步解析每个环节,让复杂的图形变得易于理解和操作。
我们来看一下如何这次分享的文献中的图,是来自《A latitudinal gradient of deep-sea invasions for marine fishes》的图3,是来自IF16.6顶级期刊:
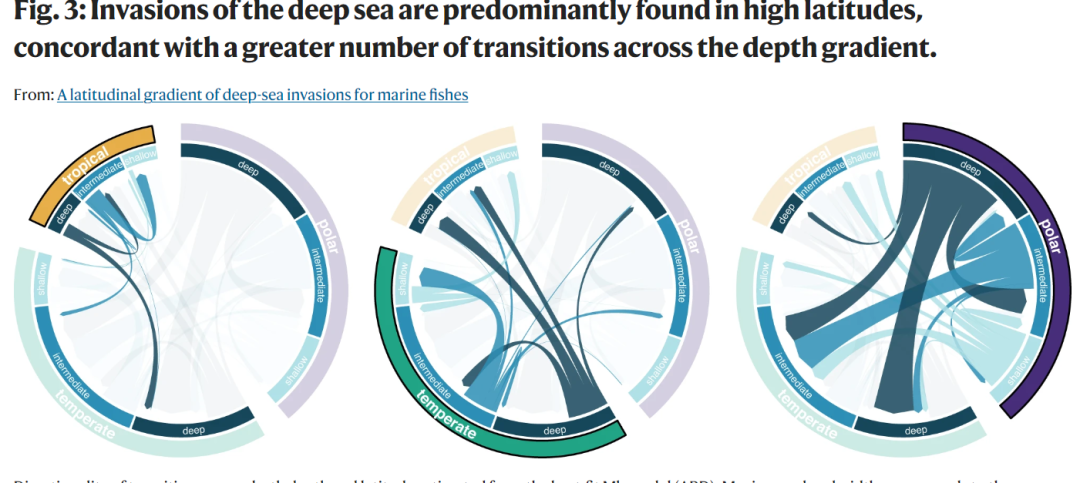
上述这个图,和我们平时见到的一些细节不一样,小伙伴也不知道如何绘制,下面我们就开始绘制吧
这里呢小果还要说明一下,我们这次做的数据分析数据量是非常大的,我们操作占用内存比较大,所以建议各位小伙伴可以使用服务器去运行,如果没有自己的服务器也没有关系,可以欢迎联系我们使用服务器租赁~
公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240202
需要租赁服务器的小伙伴可以扫码添加小果,此外小果还提供生信分析,思路设计,文献复现等,有需要的小伙伴欢迎来撩~

我们先看一下数据的格式
dat<-read_excel("data1.xlsx")dat

小伙伴可以按照这样的数据自行去设置
然后我们加载一下使用的包
library(readxl)library(circlize)
后面就可以进行绘制了,绘制之前呢,我们先设置一下Circos 图的参数
#circos.par定义 Circos 图的整体参数circos.par(start.degree = 90, #控制了起始角度#gap.degree = 4,points.overflow.warning = FALSE,#控制点溢出的警告gap.after=c("A1"=5,"A2"=5,"A3"=15,#控制在特定区块后的间隔"B1"=5,"B2"=5,"B3"=15,"D1"=5,"D2"=5,"D3"=15))这是对基础的设置了一下,还有去设置游戏额颜色的变量#颜色向量,用于设定不同区块的颜色grid.col<-c("A1"="#B0E0E6","A2"="#2C8EB5","A3"="#16465B","B1"="#B0E0E6","B2"="#2C8EB5","B3"="#16465B","D1"="#B0E0E6","D2"="#2C8EB5","D3"="#16465B")颜色设置完成后,就是对不同区域进行分,这样才能分清楚那个区域匹配那个#区块组的标签向量,用于将不同的区块分组group<-c("A1"="A","A2"="A","A3"="A","B1"="B","B2"="B","B3"="B","D1"="D","D2"="D","D3"="D")设置完,我们就创建和线图了一些代码,小果都进行了注释,就不一一举例,很好理解#创建 Chord 图chordDiagram(dat,grid.col = grid.col,#用于区块颜色的向量col = rand_color(nrow(dat)),#通过 rand_color 函数随机生成用于连线的颜色group = group,#指定区块所属的组transparency = 0.25,#设置图形透明度。directional = 1,#指定连接是否有方向性direction.type = c("arrows", "diffHeight"), #设定连接线的类型diffHeight = -0.04,#调整连接线的高度annotationTrack = "grid", #用于添加注释轨道和设置其高度。annotationTrackHeight = c(0.08, 0.1),#用于添加注释轨道和设置其高度。link.arr.type = "big.arrow", #指定连接箭头的类型# link.sort = TRUE,# link.decreasing = TRUE,link.largest.ontop = TRUE,#确保最大的连接位于最上层#调整 Chord 图中轨道的外观和布局preAllocateTracks = list(track.height = 0.1,#设置轨道的高度为 0.1#设定轨道的边距,第一个数字(0.01)代表轨道内部元素的上边距#第二个数字(0)代表轨道内部元素的下边距track.margin = c(0.01, 0)))
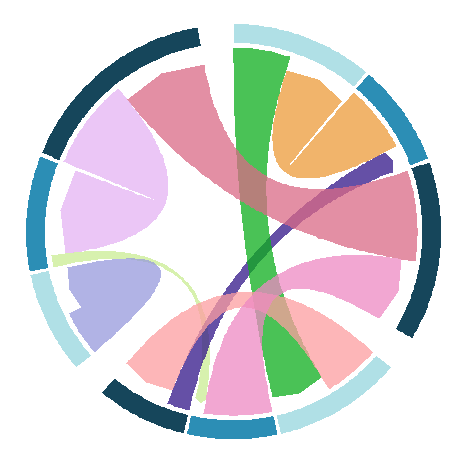
这样基础的图形就出来了,后面我们还需要进行细节的调整
去自定义一下轨道上的内容,和文献中一样,这就需要用到通过 circos.trackPlotRegion() 函数
来看一下如何绘制的
circos.trackPlotRegion(track.index = 2, #指定了要绘制的轨道的索引bg.border = NA, #设置轨道的背景边框为透明#自定义函数,用于在轨道上绘制内容#获取了当前绘图区域的 x 轴限制panel.fun = function(x, y) {#从细胞元数据中获取了部分信息,处理区块的索引或标签。xlim = get.cell.meta.data("xlim")#sector.index = get.cell.meta.data("sector.index")sector.index = gsub("[a-z]+_", "", get.cell.meta.data("sector.index"))# 用于在轨道上添加文字标签circos.text(x = mean(xlim), #指定了文本标签的 x 轴位置,绘图区域 x 轴限制的平均值作为位置y = 0.5, #文本标签的 y 轴位置设定为 0.5,指定了轨道的中心位置col = "white",#本标签的颜色设定为白色labels = sector.index, #将区块的索引或标签作为标签添加到图上facing = "bending", #设定了文本标签的朝向cex = 1,#指定了文本标签的缩放比例niceFacing = TRUE#用于确保文本标签朝向的良好显示)})

不过到这步骤,我们的基础图形就绘制的差不多,为了进一步复现文献中,我们还需要进行细节上的调整,就是突出显示 Circos 图中特定区块的部分
就需要用
highlight.sector()names(group[7:9])i<-1我们先调用完上面的分组,然后进行绘制这里就分三个模块进行了绘制,原理都一样,所以就一起绘制了:highlight.sector(names(group[7:9]),track.index = 1,facing = "bending",font = 2,col = ifelse(i == 1, "#E6AE48FF", "#E6AE483a"),border = ifelse(i == 1, TRUE, FALSE),lwd = ifelse(i == 1, 2, 0.01),text = "tropical",cex = 1.5,text.col = "white",niceFacing = TRUE)
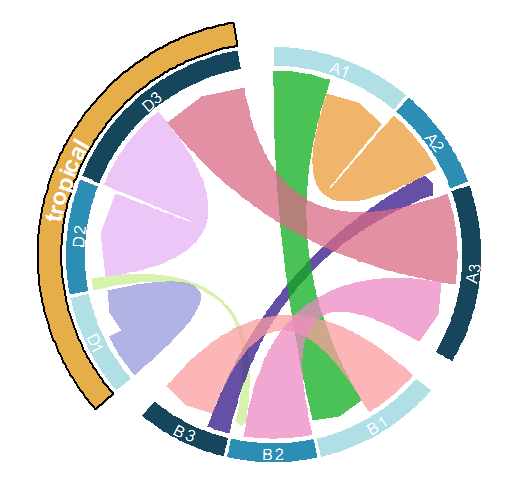
代码中ifelse根据条件选择不同的颜色和属性来突出显示区块
highlight.sector(names(group[4:6]),track.index = 1,facing = "bending",font = 2,border = ifelse(i == 2, TRUE, FALSE),lwd = ifelse(i == 2, 2, 0.01),col = ifelse(i == 2, "#20A486", "#20A4863a"),text = "temperate",cex = 1.5,text.col = "white",niceFacing = TRUE)

highlight.sector(names(group[1:3]),track.index = 1,facing = "bending",font = 2,col = ifelse(i == 3, "#472D7B", "#472D7B3a"),border = ifelse(i == 3, TRUE, FALSE),lwd = ifelse(i == 3, 2, 0.01),text = "polar",cex = 1.5,text.col = "white",niceFacing = TRUE)}
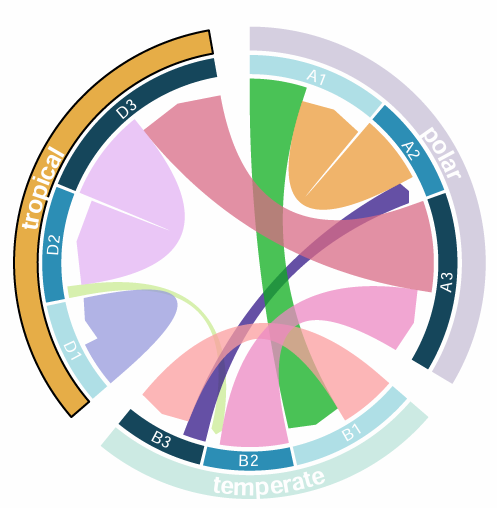
里面的字小果都按照文献中的内容进行了复现,
到现在我们的绘制就完成了,上述内容除了数据和颜色有一点不一样,小伙伴可以按照自己的喜爱去设置,记得多多理解代码的意思,这样才可以绘制出自己想要的图形,快去动手试试吧!!
最后呢,小果给小伙伴带个小福利,如果小伙伴觉得自己运行代码太麻烦,可以试试我们的云生信小工具哟,里面有成熟的代码,只要输入合适的数据就可以直接出想要的图呢。这是我们的网址
http://www.biocloudservice.com/home.html
多多关注我们公众号,一来学生信!
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果


往期回顾
|
01 |
|
02 |
|
03 |
|
04 |