测序大数据下载之最优解—Aspera connect
今天小果为大家介绍一款SRA和fastq文件的下载软件。在各位小伙伴的研究中,下载数据想必是必不可少的一环,无论是复现文献还是说进行数据库的挖掘工作。而今天介绍的这款aspera是一款与IDM与数据库合作的一款免费的命令,可以安全高效的传输文件,相对于prefetch 和 wget/crul 的方式,在速度上具有十分明显的优势!接下来,就让小果带着大家一起实践学习一下吧!
# 下载Aspera connect安装包
小果在这边为大家提供链接,各位小伙伴可以在本地下载后进行上传。(https://www.ibm.com/aspera/connect/)同样,我们也可以选择直接使用命令进行下载哦~命令小果也为大家贴在下面啦
公众号后台回复“111″,领取代码,代码编号:231003
wget -c https://d3gcli72yxqn2z.cloudfront.net/downloads/connect/latest/bin/ibm-aspera-connect_4.2.6.393_linux_x86_64.tar.gz# step 1
安装成功后,我们对压缩包进行解压与安装,命令也很简单哦,一共两条命令,用时不到10s中。
tar -zxvf ibm-aspera-connect_4.2.6.393_linux_x86_64.tar.gzbash ibm-aspera-connect_4.2.6.393_linux_x86_64.sh
# step 2
在安装结束后,想检查有没有安装成功怎么办呢?我们可以回到我们的根目录进行查看哦,因为是个隐藏目录,因此呀,所需要的命令稍微有点不同哦,下面小果为大家贴出命令。
cd ~ # 回到根目录ls -aL

# 在根目录下我们可以看到红色框框框起来的隐藏文件就证明我们已经下载成功啦~# step 3
那么这时候我们直接敲ascp命令还是会提示command not found的哦,因为小果这呀,还缺少了最后一步,我们最后需要将ascp命令配置到我们的环境中来,命令也很简单,也只有两步而已啦!
echo 'export PATH=~/.aspera/connect/bin:$PATH' >> ~/.bashrcsource .bashrcascp -h # 最终运行

到此我们就安装成功啦!

# 下载数据
紧接下来,就让小果带着大家开始实战的操作吧
第一步,小果建议大家找到SRA的编号后进入ENA数据库进行下载(https://www.ebi.ac.uk/ena/browser/home)我们在输入编号后,我们可以看到这样的一个界面
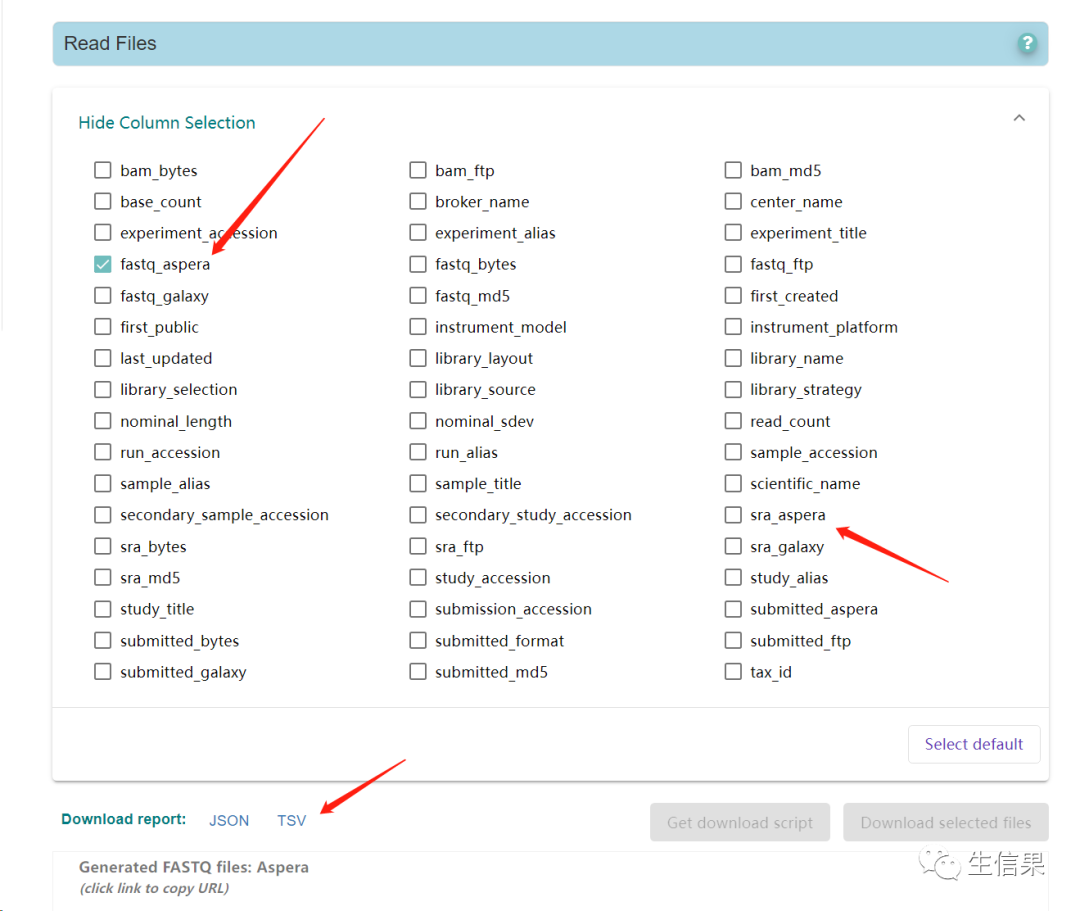
这里也可以发现小果建议大家使用ENA数据库的原因,我们从这里面可以直接选择sra文件或是fastq文件的aspera链接,点击下方的TSV便可以将我们选择的参数汇总下载成一个tsv的文件格式,如下图哦

这也是各位小伙伴在进行aspera下载的过程中最重要的一个组成部分哦!
#aspera下载命令
ascp -v -i ~/.ssh/id_rsa -P33001 -k 1 -T -l200m era-fasp@fasp.sra.ebi.ac.uk:/vol1/fastq/SRR358/006/SRR3581696/SRR3581696.fastq.gz ~/数据下载路径小果先为大家展示一下命令,然后逐个参数为各位小伙伴进行解释哦~
-v 这个参数会将下载的过程展示出来;
-i 是下载的过程中需要的密钥,不知道是不是只有小果会出现这个问题呢?小果在安装成功后,并没有在常见的文件中看到密钥文件,正常的情况下呢,密钥文件会生成在下面的这个文件下 ~/.aspera/connect/etc/asperaweb_id_dsa.openssh,而小果的密钥文件生成在了 ~/.ssh/id_sra 中,小果这边已经进行过验证啦,如果有和小果发生同样问题的小伙伴可以据此进行修改哦
-P 这是一个端口参数,在正常的使用过程中,小果建议大家就将端口设置为33001哦
-k 这个参数设置了我们的下载过程使用断续下载,他的意思就是如果我们发生了网路的波动,下载取消了,我们依然可以保留已下载的部分,是一个保证哦
-T 字面上的意思是取消加密,这个参数会更有利于各位小伙伴的下载
-l 可以通过这个参数设置下载的网速,小果建议大家设置在200m~500m之间哦,如果不设置的话,下载的速度可能会稍微慢点
以上就是我们会用上的所有参数的讲解啦
# 批量下载
小果也明白,大家都已经开始学习aspera了,那肯定是希望下载大量的数据,而小果上面给的例子仅仅有一个文件,那肯定远远不能满足各位小伙伴的需求,小果在这将批处理的代码也给到大家哦!
# step 1
我们先对从ENA数据库下载好的数据进行提取
awk -F't' 'NR>1 {print $2}' filereport_read_run_PRJNA314076_tsv.txt > extracted_data.txt这里小果用到了awk命令

# step 2
在提取完第二列后,我们就可以看到,这个就是小果之前的示例中的下载下载地址,通过下面的命令,就可以开始进行下载啦
while read -r id; doascp -v -i ~/.ssh/id_rsa -k 1 -T -l200m era-fasp@"$id" ~/biostar/aspera/done < extracted_data.txt
# step 3
但这里面有个问题,在下载的过程中,哪怕是使用aspera也是一个极其消耗时间的工作,这个时候,我们不嫩将命令放在前台进行,这样会阻碍我们进行服务器的使用,因此小果建议大家将这个命令作为bash命令,放在后台进行
vim download.sh输入命令后将上面的命令复制到里面。
过后我们就可以使用nohop 命令将下载命令挂在后台进行下载啦
nohup bash download.sh &# step 4
在运行的过程中,小果这边可以通过使用 ps 命令与 grep 命令结合来查看与 ascp 相关的进程。
ps aux | grep ascp通过这行命令可以查看到ascp命令的进程ID(PID),状态、CPU 使用率、内存使用率等
而一旦我们知道PID后,我们就可以对这个命令进行暂停,继续和停止操作
## 暂停kill -STOP <PID>## 继续kill -CONT <PID>## 停止kill -TERM <PID>
以上就是小果今天关于aspear下载命令的分享啦,以后大家在以后的下载中可以省下宝贵的时间呀!
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~
往期推荐

