你真的会画KM曲线吗?一文掌握R语言绘制KM曲线!
点击蓝字 关注我们
小伙伴们大家好啊,今天大海哥看了很多论文,发现有些文章中的生存曲线不太标准,于是今天心血来潮,想分享一期生存曲线画图教程,那么分享之前,先来了解一下什么是生存曲线?
生存曲线又称Kaplan-Meier曲线,我们一般简称KM曲线,是临床研究中最常用的图片之一,旨在描述各组患者的生存状况。KM曲线一般用于显示在研究期间生存或事件发生的概率。它考虑了样本中可能的截断和不完整数据,这些数据通常是由于失访、随访时间结束或其他原因导致的。KM曲线是一种非参数的方法,它不需要对数据进行特定的假设或分布假设。所以一张漂亮的、专业的生存曲线图不仅可以令编辑、读者和审稿专家眼前一亮,同时也能为论文增色不少。所以小伙伴们可不要轻视了它哦!
绘制KM曲线的过程一般分为4步:
1、收集生存数据:需要记录每个观察对象的起始时间和结束时间,以及是否发生感兴趣的事件(如死亡、疾病复发等)。
2、对数据进行排序:根据事件发生时间对观察对象进行排序,以便计算生存概率。
3、计算生存概率:根据事件发生情况和样本数量计算每个时间点的生存概率。在某个时间点,生存概率表示在该时间点之前生存的观察对象比例。
4、绘制KM曲线:使用累积的生存概率来绘制曲线。在每个时间点上,绘制横坐标为时间,纵坐标为生存概率的点,然后用直线将这些点连接起来。
描述了这么多,我们开始实战吧!本次分析用到的R包主要包含两个,survival和survminer。
#首先导入R包library(survival)library(survminer)#导入临床数据df<-read.csv("clinical.csv",row.names=1,header=T,check.names=F)#获取想要的生存数据,包含生存期、死亡状态、风险类型(分组)rt=df[,c("Overallsurvival","Deadstatus","RiskType")]colnames(rt)[3]="Type"head(rt)

#看看共有几组类别groupNum=length(levels(factor(rt[,"Type"])))#生存分析diff=survdiff(Surv(Overallsurvival,Deadstatus)~Type,data=rt)

#卡方检验P值为0.02,证明两组生存分析存在显著差异#设置一下阈值保留,大于0.001就保留三位小数,否则就为p<0.001pValue=diff$pif(pValue <0.001){pValue="p<0.001"}else{ pValue=paste0("p=",sprintf("%.03f",pValue))}#拟合生存曲线fit <- survfit(Surv(Overallsurvival, Deadstatus) ~ Type, data = rt)#开始画图surPlot=ggsurvplot(fit,data=rt,conf.int=F, #置信区间pval=pValue,pval.size=4,legend.labs=levels(factor(rt[,"Type"])),legend.title= "Group",xlab="Time(Day)",break.time.by = 700)
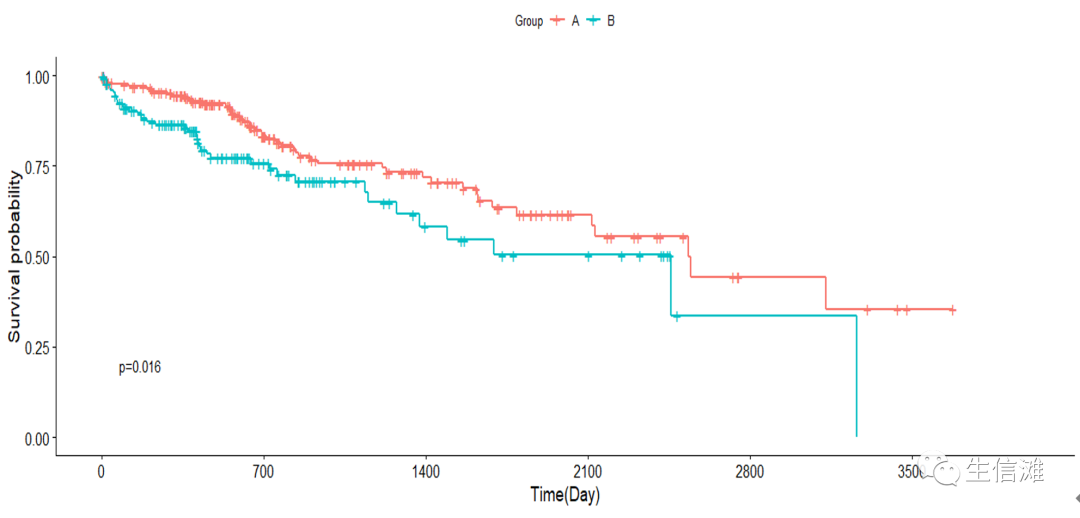
#按照标准我们还需要添加分组样本信息#加上risk.table=T参数surPlot=ggsurvplot(fit,data=rt,conf.int=F, #置信区间pval=pValue,pval.size=4,legend.labs=levels(rt$Type),legend.title="Group",xlab="Time(Day)",break.time.by = 700,risk.table=T,risk.table.height=.25)
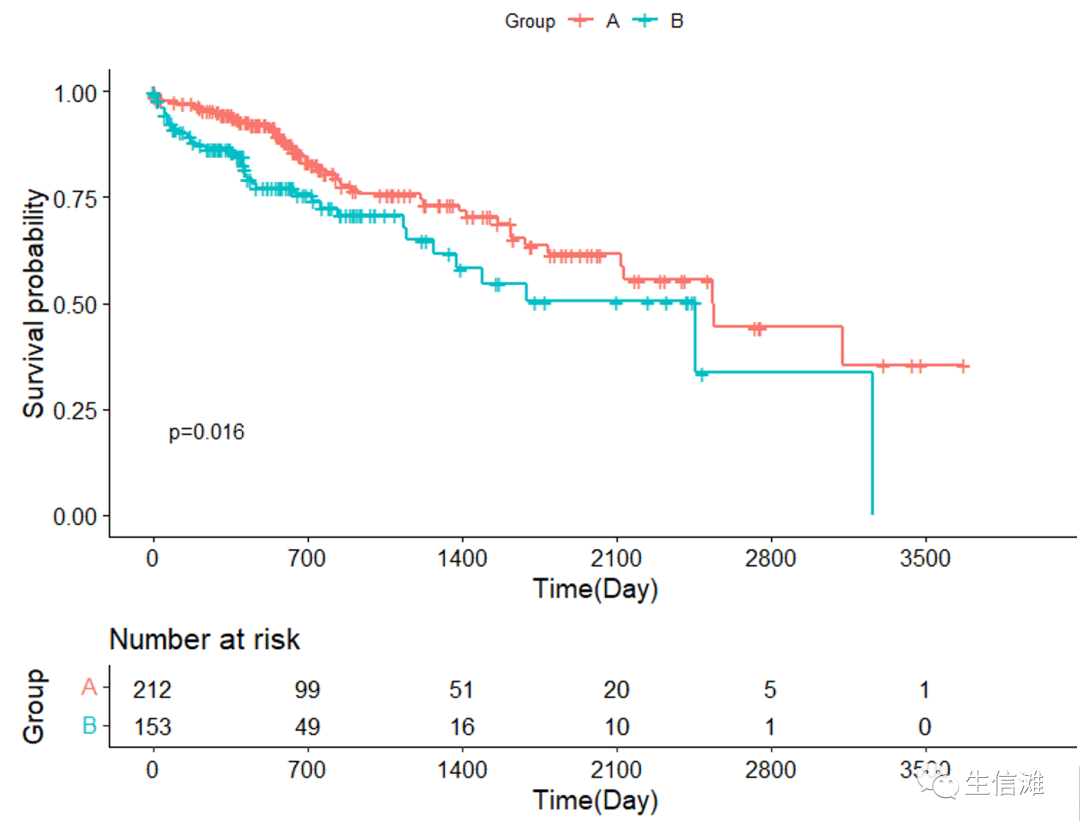
#这次就好多了,然后还少了HR值和置信区间,也需要加上去#通过coxph函数计算HR值res_cox<-coxph(Surv(Overallsurvival, Deadstatus) ~ Type, data = rt)

#然后就可以添加上去了surPlot$plot = surPlot$plot +ggplot2::annotate("text",x = 280, y = 0.14, #添加内容的所在位置,可以自己调整,大海哥这里是在左下角的位置label = paste("HR :",round(summary(res_cox)$conf.int[1],2))) #添加HR值+ ggplot2::annotate("text",x = 280, y = 0.08,label = paste("(","95%CI:",round(summary(res_cox)$conf.int[3],2),"-",#添加置信区间round(summary(res_cox)$conf.int[4],2),")",sep = ""))surPlot
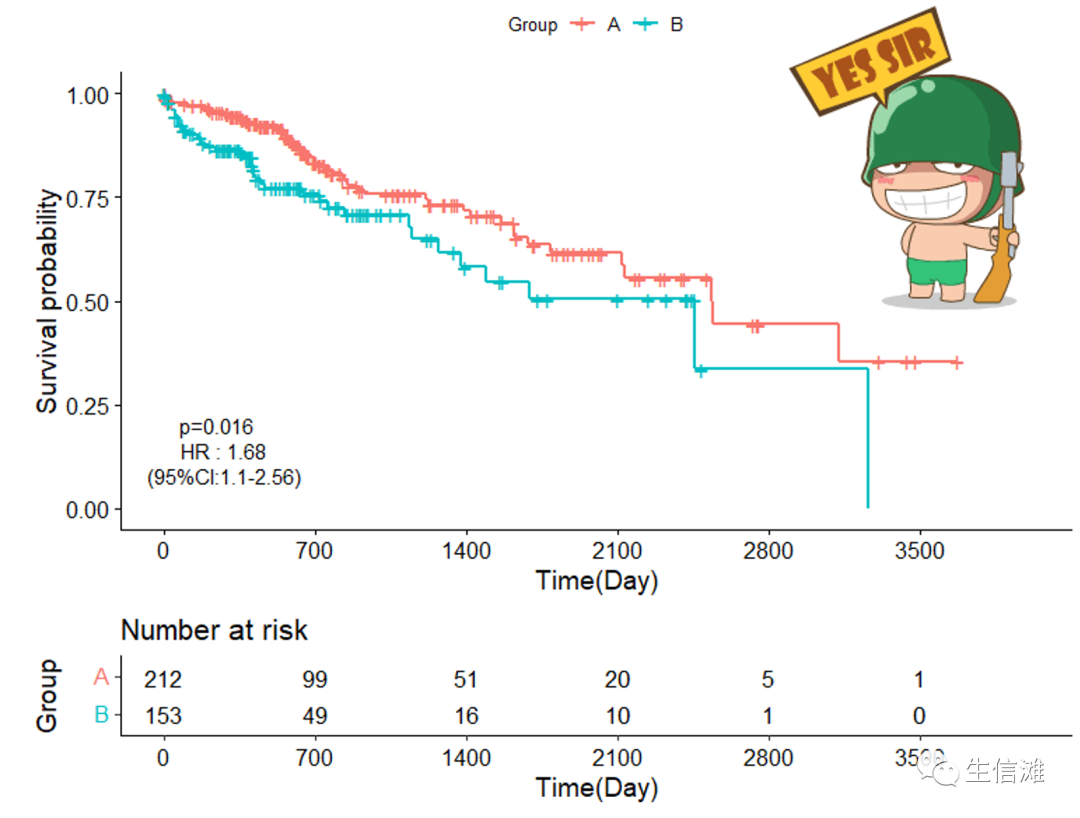
可以看到现在的KM曲线图才是一张完美的KM曲线图,但是其中有几个很重要但是很多人都会忽视的地方,大海哥来一一介绍,
首先,理想的生存曲线应该标明删失值。在上述例子中,我们注意到,生存曲线第0天和第700天之间有很多突出的小点,表示表示该点有删失病例。实际上,这才是最正规、最具有信息量的生存曲线。
其次在生存曲线的下方,最好能标识下每组的样本量,分组的依据,就是上图的下面一部分。
最后要注意的就是一定要确保数据准确哦,同时数据规模要相对较大,不然曲线会过于平滑,不具有说服力!
点击“阅读原文”进入网址